Sinalización parácrina
- "Parácrino" redirixe aquí.
A sinalización parácrina é un tipo de comunicación celular na cal unha célula produce unha molécula sinalizadora que induce cambios nas células veciñas, alterando o comportamento ou a diferenciación de ditas células. As moléculas sinalizadoras que realizan este labor denomínanse factores parácrinos, e difunden ata distancias relativamente curtas (teñen unha acción local), o que o oposto do que fan os factores endócrinos (que son hormonas que viaxan a distancias moito maiores por vía circulatoria), ou do que ocorre nas interaccións xustácrinas (nas que hai unha conexión célula-célula directa pola que se envía o sinal), ou autócrinas (nas que a molécula sinalizadora actúa sobre a membrana da mesma célula que a produciu). As células que producen os factores parácrinos segregan estes nas contornas extracelulares inmediatas. Os factores despois viaxan ata as células veciñas nas cales o gradiente do factor que reciben determina o efecto. Con todo, a distancia exacta á que os factores parácrinos poden viaxar non é moi precisa.
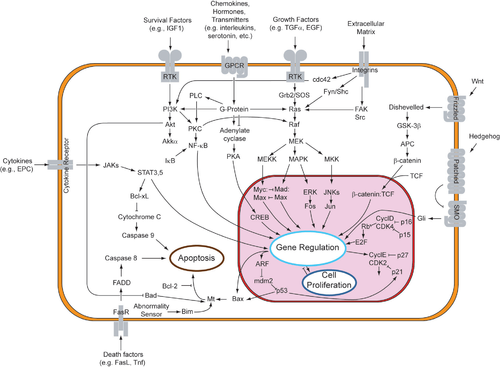
Aínda que a sinalización parácrina orixina respostas diversas nas células inducidas, a maior parte dos factores parácrinos utilizan un conxunto relativamente simplificado de receptores e vías. De feito, diferentes órganos do corpo (e mesmo de diferentes especies) utilizan conxuntos similares de factores parácrinos no desenvolvemento diferencial. Estes receptores e vías moi conservados poden estar organizados en catro familias principais baseadas en estruturas similares: familia do factor de crecemento de fibroblastos (FGF), familia de Hedgehog, familia Wnt, e superfamilia do TGF-β. A unión dun factor parácrino ao seu respectivo receptor dá inicio a fervenzas de transdución de sinais, que orixinan diferentes respostas.
Factores parácrinos que inducen unha resposta en células competentes
editarPara que os factores parácrinos induzan con éxito unha resposta na célula receptora, a célula debe ter dispoñibles os receptores axeitados na membrana celular para recibir o sinal, é dicir, debe ser competente. Ademais, a célula que responde debe tamén ter a capacidade de ser inducida por medio dalgún mecanismo.
Familia do factor de crecemento de fibroblastos (FGF)
editarAínda que a familia do factor de crecemento de fibroblastos (FGF) de factores parácrinos ten unha ampla variedade de funcións, hai evidencias importantes que apoian a idea de que o que fan principalmente é estimular a proliferación e diferenciación.[1][2] Para realizar as súas diversas funcións, os FGFs poden sufrir un splicing alternativo ou mesmo ter diferentes codóns de iniciación para así orixinar centos de isoformas diferentes de FGF.[3]
Unha das funcións máis importantes dos receptores de FGF (FGFR) é interviren no desenvolvemento das extremidades. Esta sinalización implica a nove isoformas do receptor formadas por splicing alternativo.[4] Fgf8 e Fgf10 son dúas das moléculas fundamentais para o desenvolvemento das extremidades. Na iniciación da formación do antebrazo e no crecemento das patas en ratos, sinais axiais procedentes do mesoderma intermedio producen Tbx5, o cal despois envía sinais ao propio mesoderma para que produza Fgf10. Despois, o Fgf10 envía sinais ao ectoderma para que empece a produción de Fgf8, o cal tamén estimula a produción de Fgf10. A deleción de Fgf10 orixina ratos sen patas.[5]
Ademais, a sinalización parácrina de Fgf é esencial no desenvolvemento do ollo dos polos. O ARNm de Fgf8 está localizado na parte que se diferenciará despois como a retina neural da copa óptica. Estas células están en contacto coas células ectodérmicas externas, que acabarán formando o cristalino.[3]
O fenotipo e a supervivencia dos ratos despois de sufriren un knockout dalgúns xenes de FGFR é:[4]
| Xene de FGFR con knockout | Supervivencia | Fenotipo |
|---|---|---|
| Fgf1 | Viable | Non está claro |
| Fgf3 | Viable | Oído interno, diferenciación do esqueleto (rabo) |
| Fgf4 | Letal | Proliferación da masa celular interna |
| Fgf8 | Letal | Defectos na gastrulación, desenvolvemento do sistema nervioso central, desenvolvemento das patas |
| Fgf10 | Letal | Desenvolvemento de múltiples órganos (patas, timo, pituitaria...) |
| Fgf17 | Viable | Desenvolvemento cerebelar |
Vía da receptor tirosina quinase (RTK)
editarA sinalización parácrina por medio de factores de crecemento dos fibroblastos e os seus respectivos receptores utiliza a vía da receptor tirosina quinase. Esta vía de sinalización foi moi estudada en ollos de Drosophila e cancros humanos.[6]
A unión do FGF ao FGFR fosforila a quinase inactiva da vía, activando a vía RTK. Esta vía empeza na superficie da membrana plasmática, onde un ligando se une ao seu receptor específico. Os ligandos que se unen a RTKs poden ser os factores de crecemento de fibroblastos, factores de crecemento epidérmico, factores de crecemento derivados de plaquetas, e factor de célula nai.[6] Isto dimeriza o receptor transmembrana con outro receptor RTK, o que causa a autofosforilación e o subseguinte cambio conformacional do receptor homodimerizado. Este cambio conformacional activa a quinase dormente de cada RTK nun residuo de tirosina. Debido a que o receptor abrangue todo o grosor da membrana (desde o medio extracelular atravesando toda a bicapa lipídica, e chega ao citoplasma), a unión do receptor ao ligando tamén causa a transfosforilación do dominio citoplasmático do receptor.[7]
Unha proteína adaptadora (como a SOS) recoñece a tirosina fosforilada no receptor. Esta proteína funciona coma unha ponte que conecta a RTK a unha proteína intermedia (como GNRP), dando comezo á fervenza de sinalización intracelular. Á súa vez, a proteína intermediaria estimula a Ras unida a GDP á Ras unida a GTP activada. A proteína GAP finalmente devolve a Ras ao seu estado inactivo. A activación de Ras ten o potencial de iniciar tres vías de sinalización que están máis abaixo na fervenza: a vía Ras→Raf→quinase MAP, a vía PI3 quinase, e a vía RaI. Cada vía leva á activación de factores de transcrición, que entran no núcleo para alterar a expresión xénica.[8]
Receptor RTK e cancro
editarA sinalización parácrina por factores de crecemento entre células veciñas exacerba a carcinoxénese. As formas mutantes dunha soa RTK poden ter un papel causal en moitos tipos de cancro. O protooncoxene Kit codifica un receptor tirosina quinase que ten como ligando unha proteína parácrina chamada factor de células nai (SCF), que é importante na hematopoese (formación de células sanguíneas).[9] O receptor Kit e os receptores tirosina quinase relacionados en realidade son inhibitorios e suprimen efectivamente o disparo do receptor. As formas mutantes do receptor Kit, que disparan constitutivamente de modo independente de ligando, encóntranse en diversos tumores malignos.[10]
Vía RTK e cancro
editarAs investigcións sobre o cancro de tiroide serviron para aclarar a teoría de que a sinalización parácrina pode axudar a crear microambientes tumorais. A transcrición de quimiocinas está regulada á alza cando Ras está no seu estado unido a GTP. As quimiocinas son despois liberadas da célula, e quedan libres para unirse a outras células próximas. Este bucle de retroalimentación positiva é creado pola sinalización parácrina entre as células veciñas. Así, a transcrición constitutiva de proteínas reguladas á alza forman uns ambientes ideais para que se orixinen tumores.[11] As múltiples unións de ligandos aos receptores RTK sobreestimulan a vía Ras-Maf-MAP, o cal potencia a capacidade mitoxénica e invasiva das células.[12]
Vía Jak-STAT
editarAdemais da vía RTK, os factores de crecemento de fibroblastos poden tamén activar a fervenza de sinalización Jak-STAT. Os receptores Jak-STAT, en vez de levaren dominios de tirosina quinase asociados covalentemente, forman complexos non covalentes con tirosina quinases da clase Jak (quinase Xano, en inglés Janus kinase). Estes receptores son para a eritropoetina (importante para a eritropoese), trombopoetina (importante para a formación de plaquetas), e o interferón (importante para mediar na función das células inmunitarias).[13]
Despois da dimerización dos receptores de citocinas que segue á unión de ligandos, as Jaks transfosforílanse unhas a outras. As fosfotirosinas resultantes atraen a proteínas STAT. As proteínas STAT dimerízanse e entran no núcleo para actuar como factores de transcrición para alterar a expresión xénica.[13] En particular, a STATS transcribe xenes que colaboran na proliferación e supervivencia celular, como myc.[14]
O fenotipo e a supervivencia de ratos que se observa despois de facer un knockout dalgúns xenes Jak ou STAT móstrase na táboa:[15]
| Xene con knockout | Supervivencia | Fenotipo |
|---|---|---|
| Jak1 | Letal | Déficits neurolóxicos |
| Jak2 | Letal | Eritropoese deficiente |
| Stat1 | Viable | Ananismo humano e síndromes de craniosinostoses |
| Stat3 | Letal | Fenotipos específicos de tecidos |
| Stat4 | Viable | Diferenciación de células Th1 guiada por IL-12 defectuosa, incremento da susceptibilidade a patóxenos intracelulares |
Vía Jak-STAT aberrante e mutacións no óso
editarA vía Jak-STAT é instrumental no desenvolvemento das extremidades, especificamente pola súa capacidade de regular o crecemento óseo por medio de sinalización parácrina con citocinas. Porén, as mutacións nesta vía foron implicadas en graves formas de ananismo: displasia tanatofórica (letal) e ananismo acondroplásico (viable).[16] Isto débese a unha mutación nun xene Fgf, que causa unha activación prematura e constitutiva do factor de transcrición Stat1. A división das células condrocíticas remata de forma prematura, dando como resultado un ananismo letal. O crecemento dos ósos das extremidades e das costelas vese alterado, e a incapacidade de expandirse da caixa torácica impide que o neonato respire.[17]
Vía Jak-STAT e cancro
editarA investigación da sinalización parácrina na vía Jak-STAT revelou o seu potencial para activar o comportamento invasivo das células epiteliais de ovario. Esta transición epitelial-mesenquimal é moi evidente nas metástases.[18] A sinalización parácrina a través da vía Jak-STAT é necesaria para a transición de células epiteliais estacionarias a células mesenquimais móbiles, que son quen de invadir os tecidos que as rodean. Só a vía Jak-STAT induce as células migratorias.[19]
Familia Hedgehog
editarA familia proteica de Hedgehog está implicada na indución de tipos celulares e na creación de fronteiras entre tecidos e patróns corporais e encóntrase en todos os organismos bilaterais. As proteínas Hedgehog foron descubertas e estudadas en Drosophila. A palabra significa "ourizo cacheiro" pola forma das larvas con mutacións para esta proteína. As proteínas Hedgehog producen sinais fundamentais para o establecemento das extremidades e do plan corporal das moscas da froita e para a homeostase dos tecidos adultos, implicadas na embrioxénese tardía e na metamorfose. En vertebrados atopáronse polo menos tres homólogos da hedgehog de "Drosophila", que son: sonic hedgehog, desert hedgehog, e Indian hedgehog. A sonic hedgehog (SHH) exerce varias funcións no desenvolvemento de vertebrados, actuando como mediadora de sinalizacións e regulando a organización do sistema nervioso central, extremidades e a polaridade das somitas. A desert hedgehog (DHH) exprésase nas células de Sertoli e está implicada na espermatoxénese. A Indian hedgehog (IHH) exprésase no intestino e cartilaxe, e é importante no crecemento óseo posnatal.[20][21][22]
Vía de sinalización Hedgehog
editarProteínas da famila de Hedgehog actúan uníndose a un receptor transmembrana chamado "Patched", que esta unido á proteína "Smoothened", por medio da cal o sinal de Hedgehog pode transducirse. En ausencia de Hedgehog, o receptor Patched inhibe a acción da proteína Smoothened. A inhibición de Smoothened fai que o complexo proteico "Cos"", "Cubitus interruptus" (Ci) e "Fused" unido aos microtúbulos permaneza intacto. Nesta conformación, a proteína Ci é clivada para que unha porción da proteína poida entrar no núcleo e actúe como represor transcricional. En presenza de Hedgehog, Patched xa non inhibe a Smoothened. Entón, a proteína Smoothened activa pode inhibir a proteína quinase A (PKA) e a Slimb, para que a proteína Ci non sexa clivada. Esta proteína Ci intacta pode entrar no núcleo, asóciase coa proteína CPB e actúa como un activador transcricional, inducindo a expresión dos xenes de resposta a Hedgehog.[22][23][24]
Vía de sinalización Hedgehog e cancro
editarA vía de sinalización de Hedgehog é esencial para que se forme o patrón normal dos tecidos e a súa correcta orientación durante o desenvolvemento normal de moitos animais. As proteínas Hedgehog inducen a proliferación celular en certas células e a diferencición doutras. A activación aberrante da vía Hedgehog foi implicada en varios tipos de cancros, expecialmente no carcinoma de célula basal. Esta activación incontrolada das proteínas Hedgehog pode ser causada por mutacións na vía de sinalización, que serían independentes do ligando, ou por unha mutación que causa a sobreexpresión da proteína Hedgehog, que sería dependente de ligando. Esta conexión entre a vía de sinalización Hedgehog e os cancros humanos pode utilizarse para idear posibles tratamentos para eses cancros. A vía de sinalización Hedgehog está implicada na regulación normal das poboacións de células nai, e é necesaria para o crecemento normal e rexeneración de órganos danados. Isto pode ser outra posible ruta para a xénese de tumores pola vía de Hedgehog.[25][26][27]
Familia Wnt
editarA familia da proteína Wnt inclúe un gran número de glicoproteínas ricas en cisteína. As proteínas Wnt activan fervenzas de transdución de sinais por medio de tres vías diferentes, a vía Wnt canónica, a vía da polaridade celular planar (PCP) non canónica, e a vía Wnt/Ca2+ non canónica. As proteínas Wnt parecen controlar un amplo rango de procesos de desenvolvemento e son necesarias para o control da orientación do fuso mitótico, polaridade celular, adhesión mediada por cadherinas, e o desenvolvemento temperán de embrións en moitas especies. As investigacións actuais indican que a desregulación da sinalización por Wnt xoga un papel na formación de tumores, porque a nivel celular, as proteínas Wnt a miúdo regulan a proliferación celular, morfoloxía celular, mobilidade celular, e a función final dunha célula.[28]
A vía de sinalización Wnt canónica
editarNa vía Wnt canónica, as proteínas Wnt únense ao seu receptor de membrana, que pertence á familia das proteínas Frizzled. A unión de Wnt a unha proteína Frizzled activa a proteína Dishevelled. No seu estado activo a proteína Dishevelled inhibe a actividade do encima glicóxeno sintase quinase 3 (GSK3). Normalmente, a GSK3 activa impide a disociación da β-catenina da proteína APC, o que orixina a degradación da β-catenina. Así, cando a GSK3 está inhibida, a β-catenina pode disociarse da APC, acumúlase, e viaxa ao núcleo. No núcleo a β-catenina asóciase co factor de transcrición Lef/Tcf, o cal xa está actuando sobre o ADN como represor, inhibindo a transcrición dos xenes aos que está unido. A unión da β-catenina ao Lef/Tcf fai que este funcione como activador da transcrición, activando a transcrición dos xenes de resposta a Wnt.[29][30][31]
As vías de sinalización Wnt non canónicas
editarAs vías Wnt non canónicas funcionan como unha vía de transdución para Wnt na que non está implicada a β-catenina. Nas vías non canónicas, a Wnt afecta á actina, ao citoesqueleto microtubular e á transcrición xénica.
A vía da polaridade celular planar (PCP) non canónica
editarA vía PCP non canónica regula a morfoloxía, división e movemento celular. Unha vez que as proteínas Wnt se unen e activan a Frizzled para que Frizzled active á proteína Dishevelled, que está ligada á membrana plasmática a través da proteína Prickle e a proteína transmembrana Stbm. A Dishevelled activada á súa vez activa á GTPase RhoA por medio do activador da morfoxénese 1 asociado a Dishevelled (Daam1) e a proteína Rac. A RhoA activa pode inducir cambios no citoesqueleto ao activar a quinase asociada a Roh (ROCK) e afecta directamente á transcrición xenética. A Rac activa pode inducir directamente cambios no citoesqueleto e afectar á transcrición por medio da activación de JNK.[29][30][31]
A vía Wnt/Ca2+ non canónica
editarA vía Wnt/Ca2+ non canónica regula os niveis de calcio intracelular. Unha vez máis Wnt únese a Frizzled. Pero neste caso a Frizzled activada causa que unha proteína G acoplada active unha fosfolipase (PLC), que interacciona con PIP2, escindíndoo en DAG e IP3. O IP3 pode despois unirse a un receptor do retículo endoplasmático para liberar os almacéns de calcio intracelular, para inducir a expresión xénica dependente de calcio.[29][30][31]
A vía de sinalización Wnt e o cancro
editarAs vías de sinalización Wnt son críticas na sinalización célula-célula durante o desenvolvemento normal e a embrioxénese e son necesarios para o mantemento do tecido adulto, por tanto, non é difícil comprender por que a alteración das vías de sinalización Wnt poden promover nos humanos enfermidades dexenerativas e cancro.
As vías de sinalización Wnt son complexas, implican moitos elementos diferentes, e, por tanto, teñen moitas dianas nas que pode haber unha mala regulación. As mutacións que causan a activación constitutiva da vía de sinalización Wnt orixinan a formación dun tumor e cancro. A activación anormal da vía Wnt pode causar un aumento da proliferación celular. A investigación actual está enfocada na acción da vía de sinalización Wnt na regulación da elección que fan as células nai de proliferar e autorrenovarse. Esta acción da sinalización de Wnt no posible control e mantemento de células nai, pode proporcionar un posible tratamento para cancros que mostran unha sinalización Wnt anormal.[32][33][34]
Superfamilia do TGF-β
editarO “factor de crecemento transformante” (TGF) é unha familia de 33 proteínas que comprenden polipéptidos segregados diméricos que regulan o desenvolvemento.[35] Moitos procesos de desenvolvemento están baixo o seu control, como a gastrulación, simetría axial do corpo, morfoxénese de órganos, e a homeostase dos tecidos en adultos.[36] Todos os ligandos TGF-β se unen a receptores de tipo I ou II, creando complexos heterotetraméricos.[37]
Vía do TGF-β
editarA vía do TGF-β regula moitos procesos celulares no embrión en desenvolvemento e en organismos adultos, como o crecemento celular, diferenciación, apoptose, e homeostase. Hai cinco clases de receptores de tipo II e sete clases de tipo I en humanos e outros mamíferos. Estes receptores coñécense como “quinases de especificidade dual” porque o seu dominio quinase citoplasmático ten unha feble actividade de tirosina quinase pero unha actividade forte de serina/treonina quinase.[38] Cando un ligando da superfamilia TGF-β se une a un receptor de tipo II, recruta un receptor de tipo I e actívao ao fosforilar os residuos serina ou treonina da súa caixa “GS”.[39] Isto forma un complexo de activación que pode despois fosforilar proteínas SMAD.
Vía SMAD
editarHai tres clases de proteínas SMAD:
Exemplos de SMADs de cada clase:[40][41][42]
| Clase | SMADs |
|---|---|
| R-SMAD | SMAD1, SMAD2, SMAD3, SMAD5 e SMAD8/9 |
| Co-SMAD | SMAD4 |
| I-SMAD | SMAD6 e SMAD7 |
A superfamilia TGF-β activa a membros da familia proteica SMAD, que funcionan como factores de transcrición. Especificamente, o receptor de tipo I, activado polo receptor de tipo II, fosforila R-SMADs, as cales despois se unen ao co-SMAD, SMAD4. O R-SMAD/Co-SMAD forma un complexo coa importina e entra no núcleo, onde actúa como factor de transcrición e pode regular á alza ou á baixa a expresión dun xene diana determinado.
Os ligandos TGF-β específicos darán lugar á activación das R-SMADs SMAD2/3 ou SMAD1/5. Por exemplo, cando un ligando activina, Nodal, ou TGF-β se une aos receptores, o complexo receptor fosforilado pode activar a SMAD2 e SMAD3 por fosforilación. Porén, cando un ligando BMP se une aos receptores, o complexo receptor fosforilado activa a SMAD1 e SMAD5. Despois, os complexos Smad2/3 ou Smad1/5 forman un complexo dímero con SMAD4 e funcionan como factores de transcrición. Aínda que nesta vía interveñen moitas R-SMADs, hai só unha co-SMAD, que é SMAD4.[43]
Vía non SMAD
editarAs proteínas de sinalización non Smad contribúen ás respostas da vía do TGF-β de tres xeitos. Primeiro, as vías de sinalización non Smad fosforilan as Smads. Segundo, as Smads sinalizan directamente a outras vías ao comunicárense directamente con outras proteínas sinalizadoras, como as quinases. Finalmente, os receptores TGF-β fosforilan directamente as proteínas non Smad.[44]
Membros da superfamilia do TGF-β
editar1. Familia do TGF-β
editarEsta familia inclúe a TGF-β1, TGF-β2, TGF-β3, e TGF-β5. Están implicados na regulación positiva e negativa da división celular, a formación da matriz extracelular entre as células, a apoptose, e a embrioxénese. Únense ao receptor de TGF-β tipo II (TGFBRII).
O TGF-β1 estimula a síntese do coláxeno e da fibronectina e inhibe a degradación da matriz extracelular. Finalmente, incrementa a produción de matriz extracelular por parte das células epiteliais.[37] As proteínas TGF-β regulan os epitelios ao controlaren onde e cando se ramifican para formar os condutos dos riles, pulmóns, e glándulas salivares.[37]
2. Familia da proteína morfoxenética do óso (BMPs)
editarAtopouse orixinalmente que membros da familia BMP inducían a formación de óso, como suxire o seu nome. Porén, as BMPs son moi multifuncionais e poden tamén regular a apoptose, migración celular, división celular, e diferenciación celular. Tamén especifican o eixe anterior/posterior do corpo, o índice de crecemento, e regulan a homeostase.[35]
As BMPs únense ao receptor da proteína morfoxenética ósea tipo II (BMPR2). Algunhas das proteínas da familia BMP son BMP4 e BMP7. A proteína BMP4 promove a formación de óso, causa a morte celular, ou sinaliza a formación de epiderme, dependendo do tecido onde actúe. A BMP7 é fundamental para o desenvolvemento dos riles, síntese de esperma, e polarización do tubo neural. Tanto a BMP4 coma a BMP7 regulan a estabilidade e procesamento de ligandos maduros, como a degradación de ligandos en lisosomas.[35] s BMPs actúan difundíndose desde as células nas que se produciron.[45]
Outros membros da superfamilia do TFG-β e as súas funcións
editar- Familia Vg1
- Familia da activina (activina e inhibina)
- Implicación na embrioxénese e osteoxénese
- Regulación da insulina e pituitaria, e hormonas gonadais e hipotalámicas
- Factores de supervivencia das células nerviosas
- 3 activinas: Activina A, Activina B e Activina AB.
- Factor neurotrófico derivado da liña glial (GDNF)
- Necesario para a diferenciación dos riles e as neuronas entéricas
- Factor inhibidor mülleriano
- Implicado na determinación do sexo dos mamíferos.
- Nodal
- Únese ao receptor de activina A tipo 2B
- Forma un complexo receptor co receptor de activina A tipo 1B ou co receptor de activina A tipo 1C.[46]
- Factores de crecemento e diferenciación (GDFs)
Táboa resumo da vía de sinalización do TFG-β
editar| Ligando da superfamilia do TGF-β | Receptor de tipo II | Receptor de tipo I | R-SMADs | Co-SMAD | Inhibidores ligandos |
|---|---|---|---|---|---|
| Activina A | ACVR2A | ACVR1B (ALK4) | SMAD2, SMAD3 | SMAD4 | Follistatina |
| GDF1 | ACVR2A | ACVR1B (ALK4) | SMAD2, SMAD3 | SMAD4 | |
| GDF11 | ACVR2B | ACVR1B (ALK4), TGFβRI (ALK5) | SMAD2, SMAD3 | SMAD4 | |
| Proteínas morfoxenéticas ósea | BMPR2 | BMPR1A (ALK3), BMPR1B (ALK6) | SMAD1 SMAD5, SMAD8 | SMAD4 | Noggina, Cordina, DAN |
| Nodal | ACVR2B | ACVR1B (ALK4), ACVR1C (ALK7) | SMAD2, SMAD3 | SMAD4 | Lefty |
| TGFβs | TGFβRII | TGFβRI (ALK5) | SMAD2, SMAD3 | SMAD4 | LTBP1, THBS1, Decorina |
Exemplos
editarOs factores de crecemento e de coagulación son axentes de sinalización parácrinos. A acción local da sinalización por factores de crecemento xoga un papel especialmente importante no desenvolvemento dos tecidos. Ademais, o ácido retinoico, que é a forma activa da vitamina A, funciona de maneira parácrina para regular a expresión xénica durante o desenvolvemento embrionario nos animais superiores.[47] Nos insectos, a alatostatina controla o crecemento por medio dunha acción parácrina sobre os corpos alados.[48]
Nos organismos maduros, a sinalización parácrina está implicada en respostas a alérxenos, reparación de tecidos, formación de tecido de cicatrización, e coagulación do sangue.[49]
Notas
editar- ↑ Gospodarowicz, D.; Ferrara, N.; Schweigerer, L.; Neufeld, G. (1987). "Structural Characterization and Biological Functions of Fibroblast Growth Factor". Endocrine Reviews 8 (2): 95–114. PMID 2440668. doi:10.1210/edrv-8-2-95.
- ↑ Rifkin, Daniel B.; Moscatelli, David (1989). "Recent developments in the cell biology of basic fibroblast growth factor". The Journal of Cell Biology 109 (1): 1–6. JSTOR 1613457. PMC 2115467. PMID 2545723. doi:10.1083/jcb.109.1.1.
- ↑ 3,0 3,1 Lappi, Douglas A. (1995). "Tumor targeting through fibroblast growth factor receptors". Seminars in Cancer Biology 6 (5): 279–88. PMID 8562905. doi:10.1006/scbi.1995.0036.
- ↑ 4,0 4,1 Xu, J.; Xu, J; Colvin, JS; McEwen, DG; MacArthur, CA; Coulier, F; Gao, G; Goldfarb, M (1996). "Receptor Specificity of the Fibroblast Growth Factor Family". Journal of Biological Chemistry 271 (25): 15292–7. PMID 8663044. doi:10.1074/jbc.271.25.15292.
- ↑ Logan, M. (2003). "Finger or toe: The molecular basis of limb identity". Development 130 (26): 6401–10. PMID 14660539. doi:10.1242/dev.00956.
- ↑ 6,0 6,1 Fantl, Wendy J; Johnson, Daniel E; Williams, Lewis T (1993). "Signalling by Receptor Tyrosine Kinases". Annual Review of Biochemistry 62: 453–81. PMID 7688944. doi:10.1146/annurev.bi.62.070193.002321.
- ↑ Yarden, Yosef; Ullrich, Axel (1988). "Growth Factor Receptor Tyrosine Kinases". Annual Review of Biochemistry 57: 443–78. PMID 3052279. doi:10.1146/annurev.bi.57.070188.002303.
- ↑ Katz, Michael E; McCormick, Frank (1997). "Signal transduction from multiple Ras effectors". Current Opinion in Genetics & Development 7 (1): 75–9. PMID 9024640. doi:10.1016/S0959-437X(97)80112-8.
- ↑ Zsebo, Krisztina M.; Williams, David A.; Geissler, Edwin N.; Broudy, Virginia C.; Martin, Francis H.; Atkins, Harry L.; Hsu, Rou-Yin; Birkett, Neal C.; Okino, Kenneth H.; Murdock, Douglas C.; Jacobsen, Frederick W.; Langley, Keith E.; Smith, Kent A.; Takeish, Takashi; Cattanach, Bruce M.; Galli, Stephen J.; Suggs, Sidney V. (1990). "Stem cell factor is encoded at the SI locus of the mouse and is the ligand for the c-kit tyrosine kinase receptor". Cell 63 (1): 213–24. PMID 1698556. doi:10.1016/0092-8674(90)90302-U.
- ↑ Rönnstrand, L. (2004). "Signal transduction via the stem cell factor receptor/c-Kit". Cellular and Molecular Life Sciences 61 (19–20): 2535–48. PMID 15526160. doi:10.1007/s00018-004-4189-6.
- ↑ Melillo, Rosa Marina; Castellone, Maria Domenica; Guarino, Valentina; De Falco, Valentina; Cirafici, Anna Maria; Salvatore, Giuliana; Caiazzo, Fiorina; Basolo, Fulvio; Giannini, Riccardo; Kruhoffer, Mogens; Orntoft, Torben; Fusco, Alfredo; Santoro, Massimo (2005). "The RET/PTC-RAS-BRAF linear signaling cascade mediates the motile and mitogenic phenotype of thyroid cancer cells". Journal of Clinical Investigation 115 (4): 1068–81. PMC 1062891. PMID 15761501. doi:10.1172/JCI22758.
- ↑ Kolch, Walter (2000). "Meaningful relationships: The regulation of the Ras/Raf/MEK/ERK pathway by protein interactions". The Biochemical journal 351 (2): 289–305. PMC 1221363. PMID 11023813. doi:10.1042/0264-6021:3510289.
- ↑ 13,0 13,1 Aaronson, David S.; Horvath, Curt M. (2002). "A Road Map for Those Who Don't Know JAK-STAT". Science 296 (5573): 1653–5. Bibcode:2002Sci...296.1653A. PMID 12040185. doi:10.1126/science.1071545.
- ↑ Rawlings, Jason S.; Rosler, Kristin M.; Harrison, Douglas A. (2004). "The JAK/STAT signaling pathway". Journal of Cell Science 117 (8): 1281–3. PMID 15020666. doi:10.1242/jcs.00963.
- ↑ O'Shea, John J; Gadina, Massimo; Schreiber, Robert D (2002). "Cytokine signaling in 2002: new surprises in the Jak/Stat pathway". Cell 109 (2): S121–31. PMID 11983158. doi:10.1016/S0092-8674(02)00701-8.
- ↑ Shiang, Rita; Thompson, Leslie M.; Zhu, Ya-Zhen; Church, Deanna M.; Fielder, Thomas J.; Bocian, Maureen; Winokur, Sara T.; Wasmuth, John J. (1994). "Mutations in the transmembrane domain of FGFR3 cause the most common genetic form of dwarfism, achondroplasia". Cell 78 (2): 335–42. PMID 7913883. doi:10.1016/0092-8674(94)90302-6.
- ↑ Kalluri, Raghu; Weinberg, Robert A. (2009). "The basics of epithelial-mesenchymal transition". Journal of Clinical Investigation 119 (6): 1420–8. PMC 2689101. PMID 19487818. doi:10.1172/JCI39104.
- ↑ Silver, Debra L.; Montell, Denise J. (2001). "Paracrine Signaling through the JAK/STAT Pathway Activates Invasive Behavior of Ovarian Epithelial Cells in Drosophila". Cell 107 (7): 831–41. PMID 11779460. doi:10.1016/S0092-8674(01)00607-9.
- ↑ Ingham, P. W.; McMahon, AP (2001). "Hedgehog signaling in animal development: Paradigms and principles". Genes & Development 15 (23): 3059–87. PMID 11731473. doi:10.1101/gad.938601.
- ↑ Bitgood, Mark J.; McMahon, Andrew P. (1995). "Hedgehog and Bmp Genes Are Coexpressed at Many Diverse Sites of Cell–Cell Interaction in the Mouse Embryo". Developmental Biology 172 (1): 126–38. PMID 7589793. doi:10.1006/dbio.1995.0010.
- ↑ 22,0 22,1 Jacob, L.; Lum, L. (2007). "Hedgehog Signaling Pathway". Science's STKE 2007 (407): cm6. PMID 17925577. doi:10.1126/stke.4072007cm6.
- ↑ Johnson, Ronald L; Scott, Matthew P (1998). "New players and puzzles in the Hedgehog signaling pathway". Current Opinion in Genetics & Development 8 (4): 450–6. PMID 9729722. doi:10.1016/S0959-437X(98)80117-2.
- ↑ Nybakken, K; Perrimon, N (2002). "Hedgehog signal transduction: Recent findings". Current Opinion in Genetics & Development 12 (5): 503–11. PMID 12200154. doi:10.1016/S0959-437X(02)00333-7.
- ↑ Collins, R. T.; Cohen, SM (2005). "A Genetic Screen in Drosophila for Identifying Novel Components of the Hedgehog Signaling Pathway". Genetics 170 (1): 173–84. PMC 1449730. PMID 15744048. doi:10.1534/genetics.104.039420.
- ↑ Evangelista, M.; Tian, H.; De Sauvage, F. J. (2006). "The Hedgehog Signaling Pathway in Cancer". Clinical Cancer Research 12 (20): 5924–8. PMID 17062662. doi:10.1158/1078-0432.CCR-06-1736.
- ↑ Taipale, Jussi; Beachy, Philip A. (2001). "The Hedgehog and Wnt signalling pathways in cancer". Nature 411 (6835): 349–54. PMID 11357142. doi:10.1038/35077219.
- ↑ Cadigan, K. M.; Nusse, R. (1997). "Wnt signaling: A common theme in animal development". Genes & Development 11 (24): 3286–305. PMID 9407023. doi:10.1101/gad.11.24.3286.
- ↑ 29,0 29,1 29,2 Dale, Trevor C. (1998). "Signal transduction by the Wnt family of ligands". The Biochemical journal 329 (Pt 2): 209–23. PMC 1219034. PMID 9425102.
- ↑ 30,0 30,1 30,2 Chen, Xi; Yang, Jun; Evans, Paul M; Liu, Chunming (2008). "Wnt signaling: The good and the bad". Acta Biochimica et Biophysica Sinica 40 (7): 577–94. PMC 2532600. PMID 18604449. doi:10.1111/j.1745-7270.2008.00440.x.
- ↑ 31,0 31,1 31,2 Komiya, Yuko; Habas, Raymond (2008). "Wnt signal transduction pathways". Organogenesis 4 (2): 68–75. PMC 2634250. PMID 19279717. doi:10.4161/org.4.2.5851.
- ↑ Logan, Catriona Y.; Nusse, Roel (2004). "The Wnt Signaling Pathway in Development and Disease". Annual Review of Cell and Developmental Biology 20: 781–810. PMID 15473860. doi:10.1146/annurev.cellbio.20.010403.113126.
- ↑ Lustig, B; Behrens, J (2003). "The Wnt signaling pathway and its role in tumor development". Journal of cancer research and clinical oncology 129 (4): 199–221. PMID 12707770. doi:10.1007/s00432-003-0431-0.
- ↑ Neth, Peter; Ries, Christian; Karow, Marisa; Egea, Virginia; Ilmer, Matthias; Jochum, Marianne (2007). "The Wnt Signal Transduction Pathway in Stem Cells and Cancer Cells: Influence on Cellular Invasion". Stem Cell Reviews 3 (1): 18–29. PMID 17873378. doi:10.1007/s12015-007-0001-y.
- ↑ 35,0 35,1 35,2 Bandyopadhyay, Amitabha; Tsuji, Kunikazu; Cox, Karen; Harfe, Brian D.; Rosen, Vicki; Tabin, Clifford J. (2006). "Genetic Analysis of the Roles of BMP2, BMP4, and BMP7 in Limb Patterning and Skeletogenesis". PLoS Genetics 2 (12): e216. PMC 1713256. PMID 17194222. doi:10.1371/journal.pgen.0020216.
- ↑ Attisano, Liliana; Wrana, Jeffrey L. (2002). "Signal Transduction by the TGF-β Superfamily". Science 296 (5573): 1646–7. Bibcode:2002Sci...296.1646A. PMID 12040180. doi:10.1126/science.1071809.
- ↑ 37,0 37,1 37,2 Wrana, Jeffrey L.; Ozdamar, Barish; Le Roy, Christine; Benchabane, Hassina (2008). "Signaling Receptors of the TGF-β Family". En Derynck, Rik; Miyazono, Kohei. The TGF-β Family. pp. 151–77. ISBN 978-0-87969-752-5.
- ↑ ten Dijke, Peter; Heldin, Carl-Henrik (2006). "The Smad family". En ten Dijke, Peter; Heldin, Carl-Henrik. Smad Signal Transduction: Smads in Proliferation, Differentiation and Disease. Proteins and Cell Regulation 5. Dordrecht: Springer. pp. 1–13. ISBN 978-1-4020-4709-1.
- ↑ Moustakas, Aristidis (2002-09-01). "Smad signalling network". Journal of Cell Science 115 (17): 3355–6. PMID 12154066. Arquivado dende o orixinal o 16 de setembro de 2019. Consultado o 15 de novembro de 2014.
- ↑ Wu, Jia-Wei; Hu, Min; Chai, Jijie; Seoane, Joan; Huse, Morgan; Li, Carey; Rigotti, Daniel J.; Kyin, Saw; Muir, Tom W.; Fairman, Robert; Massagué, Joan; Shi, Yigong (2001). "Crystal Structure of a Phosphorylated Smad2". Molecular Cell 8 (6): 1277–89. PMID 11779503. doi:10.1016/S1097-2765(01)00421-X.
- ↑ Pavletich, Nikola P.; Hata, Yigong; Lo, Akiko; Massagué, Roger S.; Pavletich, Joan (1997). "A structural basis for mutational inactivation of the tumour suppressor Smad4". Nature 388 (6637): 87–93. PMID 9214508. doi:10.1038/40431.
- ↑ Itoh, Fumiko; Asao, Hironobu; Sugamura, Kazuo; Heldin, Carl-Henrik; Ten Dijke, Peter; Itoh, Susumu (2001). "Promoting bone morphogenetic protein signaling through negative regulation of inhibitory Smads". The EMBO Journal 20 (15): 4132–42. PMC 149146. PMID 11483516. doi:10.1093/emboj/20.15.4132.
- ↑ Schmierer, Bernhard; Hill, Caroline S. (2007). "TGFβ–SMAD signal transduction: Molecular specificity and functional flexibility". Nature Reviews Molecular Cell Biology 8 (12): 970–82. PMID 18000526. doi:10.1038/nrm2297.
- ↑ Moustakas, Aristidis; Heldin, Carl-Henrik (2005). "Non-Smad TGF-β signals". Journal of Cell Science 118 (16): 3573–84. PMID 16105881. doi:10.1242/jcs.02554.
- ↑ Ohkawara, Bisei; Iemura, Shun-Ichiro; Ten Dijke, Peter; Ueno, Naoto (2002). "Action Range of BMP is Defined by Its N-Terminal Basic Amino Acid Core". Current Biology 12 (3): 205–9. PMID 11839272. doi:10.1016/S0960-9822(01)00684-4.
- ↑ Munir, Sadia; Xu, Guoxiong; Wu, Yaojiong; Yang, Burton; Lala, Peeyush K.; Peng, Chun (2004). "Nodal and ALK7 Inhibit Proliferation and Induce Apoptosis in Human Trophoblast Cells". Journal of Biological Chemistry 279 (30): 31277–86. PMID 15150278. doi:10.1074/jbc.M400641200.
- ↑ Duester, Gregg (September 2008). "Retinoic acid synthesis and signaling during early organogenesis". Cell 134 (6): 921–31. PMC 2632951. PMID 18805086. doi:10.1016/j.cell.2008.09.002.
- ↑ Jan A. Veenstra. Peptidergic paracrine and endocrine cells in the midgut of the fruit fly maggot. Cell and Tissue Research. May 2009, Volume 336, Issue 2, pp 309-323 Date: 25 Mar 2009. [1]
- ↑ Arthur J. Chu. Tissue Factor, Blood Coagulation, and Beyond: An Overview. International Journal of InflammationVolume 2011 (2011), Article ID 367284, 30 pages http://dx.doi.org/10.4061/2011/367284 [2] Arquivado 08 de novembro de 2014 en Wayback Machine.
Véxase tamén
editarOutros artigos
editarLigazóns externas
editar- Paracrine Signaling Medical Subject Headings (MeSH) na Biblioteca Nacional de Medicina dos EUA.