ADN mitocondrial
O ADN mitocondrial ou ADNmt (en inglés mtDNA) é o ADN que se encontra dentro das mitocondrias celulares, orgánulos eucariotas que fabrican a maior parte do ATP (molécula que almacena e cede enerxía na célula) a partir da enerxía obtida na oxidación dos alimentos. Porén, a maioría do ADN presente nas células eucarióticas está no núcleo celular.
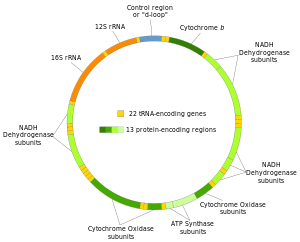
O ADN mitocondrial pode considerarse como o noso "cromosoma" máis pequeno, aínda que é bastante distinto dos verdadeiros cromosomas do núcleo. Foi a primeira parte do xenoma humano que foi secuenciada. Nos humanos e na maioría das especies hérdase só da nai.
Coñécese a secuencia do ADNmt de moitos organismos e individuos (incluíndo algúns extinguidos), e a súa comparación é moi importante en filoxenia. Permite tamén un exame das relacións entre poboacións humanas, moi interesante en antropoloxía.
Xenoma mitocondrial
editarEstrutura
editarNos humanos (e probablemente nos animais en xeral), están presentes en cada célula de 100-10.000 copias do ADNmt (o óvulo e o espermatozoide son excepcións). Nos mamíferos cada molécula de ADNmt circular de dobre cadea consta de 15.000-17.000 pares de bases. As dúas cadeas do ADNmt diferéncianse polo seu contido de nucleótidos, e unha das cadeas, chamada cadea pesada, é rica en guanina, e outra, a lixeira, é rica en citosina. A cadea pesada codifica 28 xenes, e a lixeira codifica 9, o que fai un total de 37 xenes. Deses 37 xenes, 13 codifican proteínas (polipéptidos), 22 codifican ARNt e 2 codifican o ARNr das subunidades maior e menor dos ribosomas mitocondriais. Este modelo é o que presentan tamén a maioría dos animais, aínda que nalgúns casos un ou máis dos 37 xenes pode estar ausente e o tamaño do ADNmt pode ser meirande. As variacións en tamaño e contido de xenes son aínda maiores en fungos e plantas, pero parece haber un conxunto básico de xenes que están presentes en todos os eucariotas (excepto aqueles poucos que non teñen mitocondrias en absoluto). Algunhas plantas teñen enormes ADNmt (de ata 2 500 000 pares de bases en cada molécula de ADNmt) pero, sorprendentemente, mesmo eses enormes ADNmt conteñen o mesmo número e tipo de xenes que en plantas de especies emparentadas filoxeneticamente con ADNmt moito menor.[2]
O xenoma da mitocondria do cogombro (Cucumis sativus) consta de tres cromosomas circulares distintos totalmente ou case autónomos de lonxitudes de 1556, 84 e 45 quilobases.[3]
Xenes
editarO contido do ADNmt varía dunha especie a outra. Os datos que se mostran aquí refírense ao xenoma mitocondrial humano.
Xenes da cadea de transporte electrrónico
editarO xenoma mitocondrial humano contén 13 xenes que codifican proteínas, que son os da táboa. Moitos destes xenes codifican proteínas da cadea de transporte electrónico, ou codifican a ATP-sintase.
| Categoría | Xenes |
| NADH deshidroxenase (complexo I) |
MT-ND1, MT-ND2, MT-ND3, MT-ND4, MT-ND4L, MT-ND5, MT-ND6 |
| Coencima Q-citocromo c redutase/Citocromo b (complexo III) |
MT-CYB |
| citocromo c oxidase (complexo IV) |
MT-CO1, MT-CO2, MT-CO3 |
| ATP sintase | MT-ATP6, MT-ATP8 |
Xenes de ARNr
editarO ARNr mitocondrial está codificado polos xenes MT-RNR1 (o de 12S) e MT-RNR2 (o de 16S).
Xenes de ARNt
editarOs seguintes xenes codifican o ARNt (indícase o aminoácido transportado polo ARNt):
| Aminoácido | abrev. de 3 letras | abrev. de 1 letra | ADNmt |
|---|---|---|---|
| Alanina | Ala | A | MT-TA |
| Arxinina | Arg | R | MT-TR |
| Asparaxina | Asn | N | MT-TN |
| Ácido aspártico | Asp | D | MT-TD |
| Cisteína | Cys | C | MT-TC |
| Ácido glutámico | Glu | E | MT-TE |
| Glutamina | Gln | Q | MT-TQ |
| Glicina | Gly | G | MT-TG |
| Histidina | His | H | MT-TH |
| Isoleucina | Ile | I | MT-TI |
| Leucina | Leu | L | MT-TL1, MT-TL2 |
| Lisina | Lys | K | MT-TK |
| Metionina | Met | M | MT-TM |
| Fenilalanina | Phe | F | MT-TF |
| Prolina | Pro | P | MT-TP |
| Serina | Ser | S | MT-TS1, MT-TS2 |
| Treonina | Thr | T | MT-TT |
| Triptófano | Trp | W | MT-TW |
| Tirosina | Tyr | Y | MT-TY |
| Valina | Val | V | MT-TV |
Replicación
editarA replicación do ADNmt faina o complexo γ da ADN polimerase, que está composto por unha ADN polimerase catalítica de 140 kDa codificada polo xene POLG, e unha subunidade accesoria de 55 kDa codificada polo xene POLG2. Durante a embrioxénese a replicación do ADNmt está estritamente regulada desde a fertilización do óvulo ata a preimplantación do embrión.[4] Na fase de blastocisto, o comezo da replicación do ADNmt é específico das células do trofoblasto.[4] Polo contrario, as células da masa celular interna restrinxen a replicación do ADNmt ata que reciben sinais para diferenciarse en tipos celulares específicos.[4]
Orixe
editarO ADN nuclear e o mitocondrial teñen unha orixe evolutiva diferente. O ADNmt deriva dos xenomas circulares das bacterias que foron incorporadas polos devanceiros dos eucariotas e viviron en endosimbiose con eles, acabando por converterse nun orgánulo da célula hospedadora. Esta teoría denomínase teoría endosimbiótica. Cada mitocondria estímase que contén de 2-10 copias do ADNmt.[5] Nas células dos organismos actuais, a gran maioría das proteínas presentes nas mitocondrias (ata 1500 diferentes en mamíferos) están codificadas polo ADN nuclear, pero os xenes dalgunhas delas, se non da maioría, pénsase que teñen orixe bacteriana, pero que foron despois transferidos ao núcleo eucariótico durante a evolución.
Herdanza do ADNmt
editarNa maioría dos seres pluricelulares (animais, plantas e fungos) o ADNmt hérdase só da nai. As razóns de que sexa así son: a simple dilución (porque o ovocito contén de 100.000 a 1 000 000 de moléculas de ADNmt, mentres que o espermatozoide só de 100 a 1.000), a degradación do ADNmt do espermatozoide no ovo fertilizado, e, nalgúns organismos, que o ADNmt do espermatozoide non consegue entrar no ovo. Nos mamíferos as mitocondrias do espermatozoide non chegan a entrar no ovo ou son marcadas con ubiquitina para a súa destrución posterior na fase embrionaria,[6] Algunhas técnicas de fertilización in vitro, especialmente inxectar o espermatozoide no ovocito, poden interferir con isto.
Herdanza materna
editarO feito de que o ADNmt sexa de herdanza materna permite aos investigadores trazar liñaxes matrilineais remontándose moito no tempo. (O ADN do cromosoma Y, de herdanza paterna, úsase analogamente para trazar a liñaxe por parte de pai). Isto faise nos humanos secuenciando unha ou máis das rexións control hipervariables (HVR1 ou HVR2) do ADNmt. A rexión HVR1 consta duns 440 pares de bases. Estes 440 pb son entón comparados coas rexións control doutros individuos (poden ser suxeitos específicos ou datos de suxeitos almacenados nunha base de datos) para determinar a liñaxe materna. O máis frecuente é comparar a chamada Secuencia Referencia de Cambridge revisada. Vilà et al. publicaron estudos nos que se trazaba a descencdencia matrilineal desde os cans aos lobos.[7] O concepto de Eva mitocondrial baséase no mesmo tipo de análises, cos que se trata de descubrir a orixe da humanidade rastreando a súa liñaxe no tempo.
Como a secuencia do ADNmt non está moi conservada e ten unha frecuencia rápida de mutación, é útil para estudar as relacións evolutivas (filoxenia) entre as distintas especies. Os biólogos poden determinar e despois comparar as secuencias do ADNmt entre diferentes especies e facer despois unha árbore evolutiva.
Como o ADNmt se transmite de nai a fillo/a, é útil tamén en estudos xenealóxicos para situar a unha persoa nunha liña materna determinada.
Herdanza paterna
editarHai informes de que as mitocondrias poden ter ocasionalmente herdanza paterna nunhas poucas especies, como os mexillóns.[8][9] Tamén se herdan por liña paterna nalgúns insectos como as moscas da froita,[10] abellas,[11] e carricantas.[12]
Tamén hai probas de raros casos de herdanza paterna en mamíferos, como o rato,[13][14] no que estas mitocondrias de liña paterna son despois eliminadas, e tamén en ovellas,[15] e en vacas clonadas.[16] Mesmo se atopou un único caso en humanos.[17]
Aínda que moitos deses casos referíanse a embrións clonados que despois eliminaban as mitocondrias paternas, hai outros casos nos que se documentou a súa herdanza in vivo e a persistencia posterior desas mitocondrias nas condicións de laboratorio.
Mutacións
editarSusceptibilidade
editarO ADNmt é especialmente susceptible ás especies reactivas do osíxeno xeradas pola cadea respiratoria debido á súa proximidade. Aínda que o ADNmt está empaquetado con proteínas e ten unha capacidade significativa de reparación, estas funcións protectoras son menos potentes que as que operan no ADN nuclear e, xa que logo, pénsase que isto contribúe a aumentar a susceptiobilidade ao dano oxidativo no ADNmt. Os resultados das mutacións no ADNmt poden ser a alteración na información codificada para a síntese dalgunhas proteínas,[18] que poden ter un efecto no metabolismo do organismo ou na súa capacidade de supervivencia.
Enfermidades xenéticas
editarAs mutacións no ADNmt poden orixinar varias doenzas como a intolerancia ao exercicio e a síndrome Kearns-Sayre, que causa que unha persoa perda completamente a función cardíaca, a do ollo, e os movementos musculares. Algunhas evidencias suxiren que estas mutacións poden ser unha importante contribución ao envellecemento e ás patoloxías asociadas coa idade.[19]
Uso no diagnóstico de enfermidades
editarRecentemente utilizouse unha mutación no ADNmt para axudar no diagnóstico do cáncer de próstata en pacientes con biopsias prostáticas negativas.[20][21]
Uso na identificación de persoas e estudo de relacións filoxenéticas
editarNo ser humano o ADNmt ten 16.569 pares de bases,[22] que representan só unha pequena fracción do ADN total da célula. A diferenza do ADN nuclear, que se herda dos dous proxenitores e que sofre o proceso da recombinación xenética, no ADNmt non hai normalmente cambios de pais (nais, en realidade) a fillos. O ADNmt tamén se recombina, pero só con copias de si mesmo dentro da mesma mitocondria. Por causa disto e de que a frecuencia de mutación do ADNmt animal é maior que a do nuclear,[23] o ADNmt é unha poderosa ferramenta para trazar liñaxes matrilineais, e foi usado para rastrear os antepasados de moitas especies desde centos de xeracións.
O ADNmt pode usarse tamén para identificar individuos.[24] Os laboratorios forenses usan ocasionalmente comparacións de ADNmt para identificar restos humanos, e especialmente para identificar restos esqueléticos antigos. Porén, a diferenza do ADN nuclear, o ADNmt non é específico dun individuo, pero pode usarse en combinación con outras evidencias (probas antropolóxicas, evidencias circunstanciais etc.) para establecer a identificación. O ADNmt tamén se usa para excluír posibles coincidencias entre persoas desaparecidas e restos identificados.[25] Moitos investigadores cren que o ADNmt é mellor para identificar esqueletos antigos que o ADN nuclear porque o gran número de copias existentes por cada célula incrementa a posibilidade de obter mostras útiles, e porque unha coincidencia cun parente vivo é posible mesmo se están ambos separados por numerosas xeracións maternas. Os restos do bandido norteamericano Jesse James foron identificados por medio dunha comparación entre o ADNmt extraído dos seus restos e o ADNmt dos fillos por liña materna da bisneta da súa irmá.[26] De xeito similar, puideron identificarse os restos da última emperatriz de Rusia, Alexandra Fiodorovna, e os seus fillos por comparación do seu ADNmt co do Príncipe Filipe, duque de Edimburgo, cuxa avoa materna foi a irmá de Alexandra, Vitoria de Hesse.[27] Tamén se identificaron os restos do último tsar de Rusia Nicolao II ao compararse o seu ADNmt co de James Carnegie, terceiro duque de Fife, cuxa bisavoa materna, a raíña Alexandra de Dinamarca, era irmá da nai de Nicolao II, Dagmar de Dinamarca (emperatriz María Fiodorovna).[28]
Os tamaños efectivos de poboación baixos e as rápidas frecuencias de mutación (en animais) fan ao ADNmt útil tamén para avaliar as relacións xenéticas de individuos ou grupos dunha mesma especie e tamén para identificar e cuantificar a filoxenia (relacións evolutivas) de diferentes especies, con tal de que non estean demasiado separadas no tempo. Para facelo, os biólogos determinan e despois comparan as secuencias de ADNmt de diferentes individuos ou especies. Os datos das comparacións úsanse para construír unha rede de relacións entre as secuencias, o cal proporciona unha estimación das relacións entre os individuos e as especies examinadas. Esta estratexia ten límites impostos pola frecuencia de cambio das secuencias. Por exemplo, en animais as altas taxas de mutación fan ao ADNmt máis útil para comparar individuos dunha especie e para comparar especies que teñen un parentesco próximo ou relativamene próximo, entre as cales o número de secuencias diferentes pode ser contado doadamente. Pero a medida que as especies teñen un parentesco máis afastado no tempo, o número de diferenzas nas secuencias faise máis grande, e os cambios empezan a acumularse ata que se fai imposible un reconto preciso.
Historia
editarO ADN mitocondrial foi descuberto na década de 1960 por Margit M. K. Nass e Sylvan Nass usando microscopio electrónico como unha fibra de ADN sensible ás ADNases situada no interior da mitocondria,[29] e por Ellen Haslbrunner, Hans Tuppy e Gottfried Schatz por medio de ensaios bioquímicos en fraccións mitocondriais moi purificadas.[30]
Notas
editar- ↑ Iborra FJ, Kimura H, Cook PR (2004). "The functional organization of mitochondrial genomes in human cells". BMC Biol. 2: 9. PMC 425603. PMID 15157274. doi:10.1186/1741-7007-2-9.
- ↑ Ward BL, Anderson RS, Bendich AJ (1981). "The mitochondrial genome is large and variable in a family of plants (cucurbitaceae)". Cell 25 (3): 793–803. PMID 6269758. doi:10.1016/0092-8674(81)90187-2. Consultado o 2010-08-09.
- ↑ Alverson AJ, Rice DW, Dickinson S, Barry K, Palmer JD (2011) Origins and Recombination of the Bacterial-Sized Multichromosomal Mitochondrial Genome of Cucumber. Plant Cell
- ↑ 4,0 4,1 4,2 John JC, Facucho-Oliveira J, Jiang Y, Kelly R, Salah R (2010). "Mitochondrial DNA transmission, replication and inheritance: a journey from the gamete through the embryo and into offspring and embryonic stem cells". Hum Reprod Update 16 (5): 488–509. PMID 20231166. doi:10.1093/humupd/dmq002.
- ↑ Wiesner RJ, Ruegg JC, Morano I (1992). "Counting target molecules by exponential polymerase chain reaction, copy number of mitochondrial DNA in rat tissues". Biochim Biophys Acta. 183 (2): 553–559. PMID 1550563.
- ↑ Sutovsky, P.; et al. (Nov. 25, 1999). "Ubiquitin tag for sperm mitochondria". Nature 402 (6760): 371–372. PMID 10586873. doi:10.1038/46466. Discussed in [1].
- ↑ Vilà C, Savolainen P, Maldonado JE, and Amorin IR (13 June 1997). "Multiple and Ancient Origins of the Domestic Dog". Science 276 (5319): 1687–1689. ISSN 0036-8075. PMID 9180076. doi:10.1126/science.276.5319.1687.
- ↑ Hoeh WR, Blakley KH, Brown WM (1991). "Heteroplasmy suggests limited biparental inheritance of Mytilus mitochondrial DNA". Science 251 (5000): 1488–1490. PMID 1672472. doi:10.1126/science.1672472.
- ↑ Penman, Danny (23 August 2002). "Mitochondria can be inherited from both parents". NewScientist.com. Arquivado dende o orixinal o 11 de outubro de 2008. Consultado o 2008-02-05.
- ↑ Kondo R, Matsuura ET, Chigusa SI (1992). "Further observation of paternal transmission of Drosophila mitochondrial DNA by PCR selective amplification method,". Genet. Res. 59 (2): 81–4. PMID 1628820. doi:10.1017/S0016672300030287.
- ↑ Meusel MS, Moritz RF (1993). "Transfer of paternal mitochondrial DNA during fertilization of honeybee (Apis mellifera L.) eggs". Curr. Genet. 24 (6): 539–43. PMID 8299176. doi:10.1007/BF00351719.
- ↑ Fontaine, KM, Cooley, JR, Simon, C (2007). "Evidence for paternal leakage in hybrid periodical cicadas (Hemiptera: Magicicada spp.)". PLoS One. 9 (9): e892. PMC 1963320. PMID 17849021. doi:10.1371/journal.pone.0000892.
- ↑ Gyllensten U, Wharton D, Josefsson A, Wilson AC (1991). "Paternal inheritance of mitochondrial DNA in mice". Nature 352 (6332): 255–7. PMID 1857422. doi:10.1038/352255a0.
- ↑ Shitara H, Hayashi JI, Takahama S, Kaneda H, Yonekawa H (1998). "Maternal inheritance of mouse mtDNA in interspecific hybrids: segregation of the leaked paternal mtDNA followed by the prevention of subsequent paternal leakage". Genetics 148 (2): 851–7. PMC 1459812. PMID 9504930.
- ↑ Zhao X, Li N, Guo W; et al. (2004). "Further evidence for paternal inheritance of mitochondrial DNA in the sheep (Ovis aries)". Heredity 93 (4): 399–403. PMID 15266295. doi:10.1038/sj.hdy.6800516.
- ↑ Steinborn R, Zakhartchenko V, Jelyazkov J; et al. (1998). "Composition of parental mitochondrial DNA in cloned bovine embryos". FEBS Lett. 426 (3): 352–6. PMID 9600265. doi:10.1016/S0014-5793(98)00350-0.
- ↑ Schwartz M, Vissing J (2002). "Paternal inheritance of mitochondrial DNA". N. Engl. J. Med. 347 (8): 576–80. PMID 12192017. doi:10.1056/NEJMoa020350.
- ↑ C.Michael Hogan. 2010. Mutation. ed. E.Monosson and C.J.Cleveland. Encyclopedia of Earth. National Council for Science and the Environment. Washington DC
- ↑ Alexeyev, Mikhail F.; LeDoux, Susan P.; Wilson, Glenn L. (July 2004). "Mitochondrial DNA and aging" (PDF). Clinical Science 107 (4): 355–364. PMID 15279618. doi:10.1042/CS20040148.
- ↑ Reguly B, Jakupciak JP, Parr RL. (2010). "3.4 kb mitochondrial genome deletion serves as a surrogate predictive biomarker for prostate cancer in histopathologically benign biopsy cores". Can Urol Assoc J. 4 (5): E118–22. PMID 20944788.
- ↑ Robinson K, Creed J, Reguly B, Powell C, Wittock R, Klein D, Maggrah A, Klotz L, Parr RL, Dakubo GD. (2010). "Accurate prediction of repeat prostate biopsy outcomes by a mitochondrial DNA deletion assay". Prostate Cancer Prostatic Dis. 13 (2): 126–31. PMID 20084081.
- ↑ "Copia arquivada". Arquivado dende o orixinal o 13 de agosto de 2011. Consultado o 13 de xullo de 2011.
- ↑ Brown WM, George M Jr., Wilson AC (1979). "Rapid evolution of mitochondrial DNA". Proc Natl Acad Sci USA 76 (4): 1967–1971. PMC 383514. PMID 109836. doi:10.1073/pnas.76.4.1967.
- ↑ Brown WM (1980). "Polymorphism in mitochondrial DNA of humans as revealed by restriction endonuclease analysis". Proc Natl Acad Sci USA 77 (6): 3605–3609. PMC 349666. PMID 6251473. doi:10.1073/pnas.77.6.3605.
- ↑ "Paleo-DNA Laboratory - Forensic Services". Arquivado dende o orixinal o 13 de marzo de 2012. Consultado o 13 de xullo de 2011.
- ↑ Stone AC, Starrs JE, Stoneking M (2001). "Mitochondrial DNA analysis of the presumptive remains of Jesse James" (PDF). J. Forensic Sci. 46 (1): 173–6. PMID 11210907. Arquivado dende o orixinal (PDF) o 22 de febreiro de 2006. Consultado o 13 de xullo de 2011.
- ↑ Gill P, Ivanov PL, Kimpton C; et al. (1994). "Identification of the remains of the Romanov family by DNA analysis". Nat. Genet. 6 (2): 130–5. PMID 8162066. doi:10.1038/ng0294-130.
- ↑ Os detalles das probas publicáronse en Gil et al., Identification of the Remains. O duque de Fife foi oficialmente nomeado como a fonte da mostra de comparación do ADNmt en Ivanov, Mitochondrial DNA, p. 419.
- ↑ NASS MM, NASS S (1963). "INTRAMITOCHONDRIAL FIBERS WITH DNA CHARACTERISTICS. I. FIXATION AND ELECTRON STAINING REACTIONS" (PDF) 19: 593–611. PMC 2106331. PMID 14086138.
- ↑ Ellen Haslbrunner, Hans Tuppy and Gottfried Schatz (1964 no Instituto de Bioquímic na facultade de Medicina da Universidade de Viena en Viena, Austria): "Deoxyribonucleic Acid Associated with Yeast Mitochondria" (PDF) Biochem. Biophys. Res. Commun. 15, 127 - 132.
Véxase tamén
editar| Wikimedia Commons ten máis contidos multimedia na categoría: ADN mitocondrial |
Outros artigos
editar- Eva mitocondrial
- Migracións humanas prehistóricas
- Evidencias xenéticas da orixe asiática dos xitanos
- Xuetas
- Análises moleculares de ADN
- Historia xenética da Península Ibérica
- Historia xenética de África
- Historia xenética de Europa
- Historia xenética dos pobos indíxenas de América
- Cinetoplasto
- ADN cloroplástico