Coencima Q-citocromo c redutase
A coencima Q-citocromo c redutase, tamén chamada ubiquinol-citocromo c redutase, complexo do citocromo bc1 ou Complexo III respiratorio (número EC 1.10.2.2, ver máis nomes máis abaixo) é o complexo encimático que forma o terceiro complexo da cadea de transporte de electróns respiratoria, que é fundamental para que funcione o transporte de electróns que posibilita a xeración posterior de ATP (molécula que almacena enerxía) durante a fosforilación oxidativa. O complexo consta de moitas subunidades proteicas transmembrana. O Complexo III está presente na mitocondria de todos os animais e todos os eucariotas aeróbicos e na membrana plasmática da maioría das eubacterias. As mutacións no Complexo III causan intolerancia ao exercicio e trastornos multisistema. O complexo do bc1 consta de 11 subunidades, 3 subunidades respiratorias (citocromo B, citocromo C1, proteína de Rieske), 2 proteínas do core e 6 proteínas de baixo peso molecular.
| Coencima Q-citocromo c redutase | |||||||||
|---|---|---|---|---|---|---|---|---|---|
 Estrutura cristalina do complexo do citocromo bc1 mitocondrial unido á ubiquinona.[1] | |||||||||
| Identificadores | |||||||||
| Símbolo | UCR_TM | ||||||||
| Pfam | PF02921 | ||||||||
| InterPro | IPR004192 | ||||||||
| SCOPe | 1be3 / SUPFAM | ||||||||
| TCDB | 3.D.3 | ||||||||
| OPM superfamily | 345 | ||||||||
| OPM protein | 3cx5 | ||||||||
| |||||||||
| ubiquinol—citocromo-c redutase | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| Identificadores | |||||||||
| Número EC | 1.10.2.2 | ||||||||
| Número CAS | 9027-03-6 | ||||||||
| Bases de datos | |||||||||
| IntEnz | vista de IntEnz | ||||||||
| BRENDA | entrada de BRENDA | ||||||||
| ExPASy | vista de NiceZyme | ||||||||
| KEGG | entrada de KEGG | ||||||||
| MetaCyc | vía metabólica | ||||||||
| PRIAM | perfil | ||||||||
| Estruturas PDB | RCSB PDB PDBe PDBj PDBsum | ||||||||
| Gene Ontology | AmiGO / EGO | ||||||||
| |||||||||
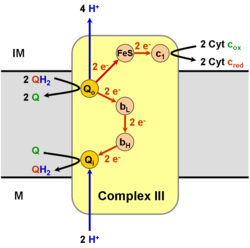

A ubiquinol—citocromo c redutase cataliza a seguinte reacción química:
- QH2 + 2 ferricitocromo c Q + 2 ferrocitocromo c + 2 H+
Os dous substratos deste encima son a dihidroquinona (QH2) e o ferri- (Fe3+) citocromo c, e os seus tres produtos son: quinona (Q), ferro- (Fe2+) citocromo c, e H+.
Este encima pertence á familia das oxidorredutases, concretamente ás que actúan sobre os difenois e substrancias relacionadas como doantes cun citocromo como aceptor. Este encima forma parte da cadea de transporte electrónico, polo que participa na fosforilación oxidativa. Ten catro cofactores, que son: citocromo C1, citocromo b-562, citocromo b-566 e unha ferredoxina con dous átomos de ferro.
Nomenclatura
editarO nome sistemático desta clase de encimas é ubiquinol:ferricitocromo-c oxidorredutase. Entre os outros nomes que se lle teñen dado están:
|
|
Xenes humanos
editarO Complexo III é un complexo proteico de moitas subunidades codificadas tanto no xenoma mitocondrial (caso do seu compoñente o citocromo b) coma no xenoma nuclear (o resto das subunidades). Os xenes humanos que as codifican son:
MTCYB: citocromo b codificado no ADNmt; mutacións asociadas coa intolerancia ao exercicio
CYC1: citocromo c1
CYCS: citocromo c
UQCRFS1: proteína ferro-sulfurada de Rieske
UQCRB: proteína de unión á ubiquinona, mutación ligada coa deficiencia do Complexo III mitocondrial tipo nuclear 3
UQCRH: proteína bisagra
UQCRC2: Core 2 (proteína central 2), mutacións ligadas coa deficiencia do Complexo III mitocondrial, tipo nucler 5
UQCRC1: Core 1
UQCR: subunidade de 6,4KD
UQCR10: subunidade de 7,2KD
TTC19: nova subunidade identificada; mutacións ligadas coa deficiencia do Complexo III, tipo nuclear 2.
Estrutura
editarComparado con outras subunidades que bombean protóns da cadea de transporte de electróns, o número de subunidades encontradas pode ser nalgunhas especies pequeno, de tan só tres cadeas de polipéptidos, pero este número aumenta nos animais superiores, nos que se encontran 11 subunidades.[2] Tres das subunidades teñen grupos prostéticos. A subunidade do citocromo b ten dous hemos B (bL e bH), a subunidade do citocromo c ten un hemo C (c1), e a subunidade da proteína ferro-sulfurada de Rieske (ISP) ten un cluster de ferro-xofre con dous átomos de ferro e dous de xofre (2Fe•2S).
Reacción
editarCataliza a redución de citocromo c por oxidación do coencima Q (CoQ) e o bombeo simultáneo de 4 protóns desde a matriz mitocondrial ao espazo intermembrana:
- QH2 + 2 cytocromo c (FeIII) + 2 H+entran → Q + 2 citocromo c (FeII) + 4 H+saen
No proceso chamado ciclo Q,[3][4] consómense dous protóns da matriz, libéranse catro protóns ao espazo intermembrana, e pasan dous electróns ao citocromo c.
Mecanismo de reacción
editarO mecanismo de reacción do Complexo III (citocromo bc1, coencima Q-citocromo C redutase) coñécese como o ciclo da ubiquinona ou ciclo Q. Neste ciclo son liberados catro protóns no lado "P" positivo (espazo intermembrana), pero só se captan dous protóns desde o lado "N" negativo (matriz). Como resultado créase un gradiente de protóns a través da membrana. Na reacción global, oxídanse dúas moléculas de ubiquinol a ubiquinona e redúcese unha ubiquinona a ubiquinol. No mecanismo completo, transfírense dous electróns desde o ubiquinol á ubiquinona, por medio de dous intermediatos citocromo c.
Reacción global:
- 2 x QH2 oxidadas a Q
- 1 x Q reducida a QH2
- 2 x Cyt c1 reducidos
- 4 x H+ liberados no espazo intermembrana
- 2 x H+ recollidos da matriz
A reacción precede seguindo as seguintes etapas:
Rolda 1:
- Ao citocromo b únense un ubiquinol e unha ubiquinona.
- O centro 2Fe/2S e o hemo bL atraen cada un un electrón procedente do ubiquinol unido, liberando dous ións hidróxeno no espazo intermembrana.
- Transfírese un electrón ao citocromo c1 desde o centro 2Fe/2S, mentres outro é transferido desde o hemo bL ao hemo bH.
- O citocromo c1 transfire o seu electrón ao citocromo c (non confundilo co citocromo c1), e o hemo bH transfire o seu electrón a unha ubiquinona próxima, orixinando a formación dunha ubisemiquinona.
- O citocromo c difunde. Libérase o primeiro ubiquinol (agora oxidado a ubiquinona), e a semiquinona permanece unida.
Rolda 2:
- Únese un segundo ubiquinol ao citocromo b.
- O centro 2Fe/2S e o hemo bL atraen cada un un electrón do ubiquinol unido, liberando dous ión hidróxeno ao espazo intermembrana.
- Transfírese un electrón ao citocromo c1 desde o centro 2Fe/2S, e transfírese outro máis desde o hemo bL ao hemo bH.
- O citrocromo c1 transfire despois o seu electrón ao citocromo c, mentres que a semiquinoma adxacente recolle un segundo electrón procedente do hemo bH, xunto con dous protóns da matriz.
- Libéranse o segundo ubiquinol (agora oxidado a ubiquinona), xunto co ubiquinol acabado de formarse.[5]
Inhibidores do complexo III
editarHai tres grupos distintos de inhibidores do Complexo III.
- A Antimicina A únese ao sitio Qi e inhibe a transferencia de electróns no Complexo III desde o hemo bH á Q oxida (inhibidor do sitio Qi).
- O mixotiazol e a estigmatelina únense ao sitio Qo e inhiben a transferencia de electróns desde a QH2 (reducida) á proteína ferro-sulfurada de Rieske. O mixotiazol e a estigmatelina únense a distintos petos do sitio Qo.
- O mixotiazol únese moi preto do citocromo bL (polo que se chama inhibidor "proximal").
- A estigmatelina únese preto da proteína ferro-sulfurada de Rieske, coa cal interacciona fortemente.
Algúns inhibidores foron comercializados como funxicidas (os derivados da estrobilurina, o máis coñecido dos cales é a azoxistrobina; inhibidores QoI) e como axentes antimaláricos (atovaquone).
Ademais, a propilhexedrina inhibe a citocromo c redutase.[6]
Radicais libres do oxíxeno
editarUnha pequena fracción dos electróns saen da cadea de transporte de electróns antes de chegaren ao complexo IV. Unha fuga prematura de electróns cara ao oxíxeno dá lugar á formación de superóxidos. A relevancia desta reacción colateral menor é que o superóxido e outras especies reactivas do oxíxeno son moi tóxicas e crese que xogan un papel en varias patoloxías, e no envellecemento (teoría do envellecemento por radicais libres).[7] A fuga de electróns ocorre principalmente no sitio Qo e é estimulada pola antimicina A. A antimicina A pecha os hemos B no seu estado reducido ao impedir a súa reoxidación no sitio Qi, o cal, á súa vez, causa que aumenten as concentracións no estado estable de semiquinona Qo, e esta última reacciona co oxíxeno para formar superóxido. O efecto dos altos potenciais de membrana pénsase que son similares.[8] O superóxido producido no sitio Qo pode ser liberado na matriz mitocondrial[9][10] e no espazo intermembrana (desde onde pode pasar ao citosol).[9][11] Isto pode explicarse polo feito de que o Complexo III podería producir superóxido como HOO• permeable nas membranas en vez de O2-. para o cal as membranas son impermeable.[10]
Mutacións nos xenes do Complexo III en enfermidades humanas
editarAs mutacións nos xenes relacionados co Complexo III maniféstanse tipicamente como unha intolerancia ao exercicio.[12][13] Outras mutacións nestes xenes causan displasia septo-óptica[14] e trastornos multisistema.[15] Porén, as mutacións en BCS1L, un xene responsable da correcta maduración do Complexo III, poden orixinar a síndrome de Björnstad e a síndrome GRACILE, as cales en neonatos son condicións mortais que teñen manifestacións multisistema e neurolóxicas que tipifican trastornos mitocondriais graves. A patoxenicidade de varias mutacións foi verificada en sistemas modelo como os lévedos.[16]
No se coñece o grao en que estas diversas patoloxías son debidas a déficit bioenerxético ou a sobreprodución de superóxido.
Notas
editar- ↑ PDB 1ntz; Gao X, Wen X, Esser L, Quinn B, Yu L, Yu CA, Xia D (2003). "Structural basis for the quinone reduction in the bc1 complex: a comparative analysis of crystal structures of mitochondrial cytochrome bc1 with bound substrate and inhibitors at the Qi site". Biochemistry 42 (30): 9067–80. PMID 12885240. doi:10.1021/bi0341814.
- ↑ Iwata S, Lee JW, Okada K, Lee JK, Iwata M, Rasmussen B, Link TA, Ramaswamy S, Jap BK (1998). "Complete structure of the 11-subunit bovine mitochondrial cytochrome bc1 complex". Science 281 (5373): 64–71. PMID 9651245. doi:10.1126/science.281.5373.64.
- ↑ Kramer DM, Roberts AG, Muller F, Cape J, Bowman MK (2004). "Q-cycle bypass reactions at the Qo site of the cytochrome bc1 (and related) complexes". Meth. Enzymol. Methods in Enzymology 382: 21–45. ISBN 978-0-12-182786-1. PMID 15047094. doi:10.1016/S0076-6879(04)82002-0.
- ↑ Crofts AR (2004). "The cytochrome bc1 complex: function in the context of structure". Annu. Rev. Physiol. 66: 689–733. PMID 14977419. doi:10.1146/annurev.physiol.66.032102.150251.
- ↑ Ferguson SJ, Nicholls D, Ferguson S (2002). Bioenergetics (3rd ed.). San Diego: Academic. pp. 114–117. ISBN 0-12-518121-3.
- ↑ Holmes JH, Sapeika N, Zwarenstein H. Inhibitory effect of anti-obesity drugs on NADH dehydrogenase of mouse heart homogenates. Res Commun Chem Pathol Pharmacol. 1975 Aug;11(4):645-6. PMID 241101.[1]
- ↑ Muller, F. L., Lustgarten, M. S., Jang, Y., Richardson, A. and Van Remmen, H. (2007). "Trends in oxidative aging theories". Free Radic. Biol. Med. 43 (4): 477–503. PMID 17640558. doi:10.1016/j.freeradbiomed.2007.03.034.
- ↑ Skulachev VP (1996). "Role of uncoupled and non-coupled oxidations in maintenance of safely low levels of oxygen and its one-electron reductants". Q. Rev. Biophys. 29 (2): 169–202. PMID 8870073.
- ↑ 9,0 9,1 Muller F (2000). ""The nature and mechanism of superoxide production by the electron transport chain: Its relevance to aging"". AGE 23 (4): 227–253. doi:10.1007/s11357-000-0022-9.
- ↑ 10,0 10,1 Muller FL, Liu Y, Van Remmen H (2004). "Complex III releases superoxide to both sides of the inner mitochondrial membrane". J. Biol. Chem. 279 (47): 49064–73. PMID 15317809. doi:10.1074/jbc.M407715200.
- ↑ Han D, Williams E, Cadenas E (2001). "Mitochondrial respiratory chain-dependent generation of superoxide anion and its release into the intermembrane space". Biochem. J. 353 (Pt 2): 411–6. PMC 1221585. PMID 11139407. doi:10.1042/0264-6021:3530411.
- ↑ DiMauro S (2006). "Mitochondrial myopathies". Curr Opin Rheumatol 18 (6): 636–41. PMID 17053512. doi:10.1097/01.bor.0000245729.17759.f2.
- ↑ DiMauro S (2007). "Mitochondrial DNA medicine". Biosci. Rep. 27 (1–3): 5–9. PMID 17484047. doi:10.1007/s10540-007-9032-5.
- ↑ Schuelke M, Krude H, Finckh B, Mayatepek E, Janssen A, Schmelz M, Trefz F, Trijbels F, Smeitink J (2002). "Septo-optic dysplasia associated with a new mitochondrial cytochrome b mutation". Ann. Neurol. 51 (3): 388–92. PMID 11891837. doi:10.1002/ana.10151.
- ↑ Wibrand F, Ravn K, Schwartz M, Rosenberg T, Horn N, Vissing J (2001). "Multisystem disorder associated with a missense mutation in the mitochondrial cytochrome b gene". Ann. Neurol. 50 (4): 540–3. PMID 11601507. doi:10.1002/ana.1224.
- ↑ Fisher N, Castleden CK, Bourges I, Brasseur G, Dujardin G, Meunier B (2004). "Human disease-related mutations in cytochrome b studied in yeast". J. Biol. Chem. 279 (13): 12951–8. PMID 14718526. doi:10.1074/jbc.M313866200.
Véxase tamén
editarOutros artigos
editarBibliografía
editar- Marres CM, Slater EC (1977). "Polypeptide composition of purified QH2:cytochrome c oxidoreductase from beef-heart mitochondria". Biochim. Biophys. Acta. 462 (3): 531–548. PMID 597492. doi:10.1016/0005-2728(77)90099-8.
- Rieske JS (1976). "Composition, structure, and function of complex III of the respiratory chain". Biochim. Biophys. Acta. 456 (2): 195–247. PMID 788795.
- Wikstrom M, Krab K, Saraste M (1981). "Proton-translocating cytochrome complexes". Annu. Rev. Biochem. 50: 623–655. PMID 6267990. doi:10.1146/annurev.bi.50.070181.003203.
Ligazóns externas
editar- cytochrome bc1 complex site (Edward A. Berry) en lbl.gov
- cytochrome bc1 complex site (Antony R. Crofts) Arquivado 17 de setembro de 2007 en Wayback Machine. en uiuc.edu
- PROMISE Database: cytochrome bc1 complex en scripps.edu
- Interactive Molecular Model of Complex III (Requires MDL Chime)
- UMichOPM families superfamily 3 - Posicións calculadas do bc1 e complexos relacionados nas membranas
- Coenzyme Q-Cytochrome-c Reductase Medical Subject Headings (MeSH) na Biblioteca Nacional de Medicina dos EUA.
