Serina protease
As serina proteases ou serina endopeptidases (tamén serín proteases) son encimas que rompen enlaces peptídicos de proteínas, nas cales o aminoácido serina funciona como nucleófilo no sitio activo do encima.[1] Encóntranse amplamente distribuídas en eucariotas e procariotas. As serina proteases divídense en dúas clases segundo a súa estrutura: de tipo quimotripsina (ou tipo tripsina) ou de tipo subtilisina.[2] Nos humanos, son responsables de coordinar varias funcións fisiolóxicas, como a dixestión, resposta inmune, coagulación do sangue e reprodución.[1]
| Serina endopeptidases | |||||||||
|---|---|---|---|---|---|---|---|---|---|

| |||||||||
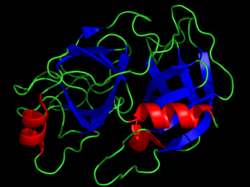
| Estrutura cristalina da quimotripsina bovina. Os residuos catalíticos móstranse en amarelo. De PDB 1CBW. | |||||||||
| Identificadores | |||||||||
| Número EC | 3.4.21. | ||||||||
| Bases de datos | |||||||||
| IntEnz | vista de IntEnz | ||||||||
| BRENDA | entrada de BRENDA | ||||||||
| ExPASy | vista de NiceZyme | ||||||||
| KEGG | entrada de KEGG | ||||||||
| MetaCyc | vía metabólica | ||||||||
| PRIAM | perfil | ||||||||
| Estruturas PDB | RCSB PDB PDBe PDBj PDBsum | ||||||||
| |||||||||
Clasificación
editarA clasificación das proteases de MEROPS comprende 16 superfamilias (en 2013) cada unha das cales contén moitas familias. Todas as superfamilias utilizan unha díade ou tríade catalítica cun diferente pregamento proteico, o que representa un caso de evolución converxente do mecanismo catalítico. A maioría pertencen á familia S1 do clan PA (superfamilia) de proteases.
Na táboa, para as superfamilias, P = superfamilia que contén unha mestura de familias de clases de nucleófilos, S = puramente serina proteases. Dentro de cada superfamilia, as familias desígnanse polo seu nucleófilo catalítico (S = serina proteases).
| Superfamilia | Familia | Exemplos |
|---|---|---|
| PA | S1, S3, S6, S7, S29, S30, S31, S32, S39, S46, S55, S64, S65, S75 | quimotripsina A (Bos taurus) |
| PB | S45, S63 | precursor da penicilina G acilase (Escherichia coli) |
| PC | S51 | dipeptidase E (Escherichia coli) |
| PE | P1 | DmpA aminopeptidase (Ochrobactrum anthropi) |
| SB | S8, S53 | subtilisina (Bacillus licheniformis) |
| SC | S9, S10, S15, S28, S33, S37 | prolyl oligopeptidase (Sus scrofa) |
| SE | S11, S12, S13 | D-Ala-D-Ala peptidase C (Escherichia coli) |
| SF | S24, S26 | sinal peptidase I (Escherichia coli) |
| SH | S21, S73, S77, S78, S80 | asemblina de citomegalovirus (herpesvirus humano 5) |
| SJ | S16, S50, S69 | Lon-A peptidase (Escherichia coli) |
| SK | S14, S41, S49 | peptidase Clp (Escherichia coli) |
| SO | S74 | endosialidase do fago K1F de Escherichia coli proteína autoclivante CIMCD (fago K1F de enterobacterias) |
| SP | S59 | nucleoporina 145 (Homo sapiens) |
| SR | S60 | lactoferrina (Homo sapiens) |
| SS | S66 | mureína tetrapeptidase LD-carboxipeptidase (Pseudomonas aeruginosa) |
| ST | S54 | romboide-1 (Drosophila melanogaster) |
| non asignadas | S48, S62, S68, S71, S72, S79, S81 |
Especificidade de substrato
editarAs serina proteases caracterízanse por ter unha estrutura distintiva que consta de dous dominios barrís beta que converxen no sitio activo catalítico. Estes encimas poden ser clasificados ademais pola súa especificidade de substrato en de tipo tripsina, tipo quimiotripsina ou tipo elastase.[3] Un grupo á parte é o das subtilisinas procarióticas.
Tipo tripsina
editarAs proteases de tipo tripsina rompen os enlaces peptídicos situados despois dun residuo aminoácido lisina ou arxinina cargado positivamente.[4] Esta especificidade está determinada polo residuo que se sitúa na base do peto S1 do encima (xeralmente un residuo de ácido aspártico ou ácido glutámico cargado positivamente).
Tipo quimotripsina
editarO peto S1 dos encimas de tipo quimotripsina é máis hidrofóbico que o das proteases de tipo tripsina. Isto ten como resultado unha especificidade por residuos hidrofóbicos de tamaño medio ou grande, como a tirosina, fenilalanina e triptófano.
Tipo elastase
editarAs proteases de tipo elastase teñen unha fenda S1 moito menor que a das proteases de tipo tripsina ou quimotripsina. En consecuencia, tenden a preferir residuos como a alanina, glicina e valina.
Tipo subtilisina
editarA subtilisina é unha serina protease de procariotas. A subtilisina non está relacionada evolutivamente co clan da quimotripsina, mais comparte o mesmo mecanismo catalítico utilizando unha tríade catalítica, para crear unha serina nucleofílica. Este é o exemplo clásico que ilustra a evolución converxente, xa que o mesmo mecanismo evolucionou dúas veces durante a evolución.
Mecanismo catalítico
editar- Véxase tamén: Tríade catalítica.
O principal protagonista no mecanismo catalítico dos encimas do clan da quimotripsina e a subtilisina mencionados antes é a tríade catalítica. A tríade está localizada no sitio activo do encima, onde ten lugar a catálise, e está preservado en todas as serina proteases. A tríade é unha estrutura coordinada que consta dos tres aminoácidos seguintes: histidina (His 57), serina (Ser 195, polo cal reciben o nome de serina proteases) e ácido aspártico (Asp 102). Estes tres aminoácidos clave xogan todos un papel esencial na capacidade de romper enlaces que teñen as proteases. Mentres que os aminoácidos membros da tríade están localizados lonxe uns dos outros na secuencia da proteína, pero debido ao pregamento, quedan moi próximos no centro do encima. A xeometría particular dos membros da tríade é moi característica das súas funcións específicas: a posición de só catro puntos da tríade caracteriza a función do encima que a contén.[5]
No momento da catálise, ocorre un mecanismo ordenado no cal se xeran varios intermediarios. A catálise da clivaxe do péptido pode considerarse que é unha catálise ping-pong, na cal un substrato se une (neste caso o polipéptido que vai ser clivado), libérase un produto (a "metade" N-terminal do péptido), únese outro substrato (neste caso a auga), e libérase outro produto (a "metade" C-terminal do péptido).
Cada aminoácido da tríade realiza unha tarefa específica neste proceso:
- A serina ten un grupo OH que pode actuar como nucleófilo, atrae o carbono do carbonilo do enlace peptídico que se vai romper do substrato.
- Un par de electróns do nitróxeno da histidina ten a capacidade de aceptar o hidróxeno do grupo OH da serina, coordinando así o ataque ao enlace peptídico.
- O grupo carboxilo do ácido aspártico á súa vez establece un enlace de hidróxeno coa histidina, facendo que o átomo de nitróxeno mencionado antes sexa moito máis electronegativo.
A reacción completa pode resumirse así:
- O polipéptido substrato únese á superficie do encima serina protease de modo que o enlace a romper se insira no sitio activo do encima, co carbono do carbonilo deste enlace situado preto da serina nucleofílica.
- O grupo OH da serina ataca o carbono carbonílico, e o nitróxeno da histidina acepta o hidróxeno do OH da serina e móvense un par de electróns desde o dobre enlace do oxíxeno carbonílico ao oxíxeno. Como resultado, xérase un intermediario tetraédrico.
- Agora rompe o enlace que une o nitróxeno e o carbono no enlace peptídico. Os electróns covalentes que forman este enlace móvense para atacar o hidróxeno da histidina, rompendo a conexión. Os electróns que previamente se moveron desde o dobre enlace do oxíxeno carbonílico móvense de novo desde o oxíxeno negativo para volver a formar o enlace, xerando un intermediario acil-encima.
- Seguidamente, entra na reacción a auga. A auga substitúe o N-terminal do péptido cortado, e ataca o carbono carbonílico. Unha vez máis, os electróns do dobre enlace móvense cara ao oxíxeno, facéndoo negativo, á vez que se forma o enlace entre o oxíxeno da auga e o carbono. Isto está coordinado polo nitróxeno da histidina, o cal acepta un protón da auga. En conxunto, isto xera outro intermediario tetraédrico.
- Nunha reacción final, o enlace formado no primeiro paso entre a serina e o carbono carbonílico móvese para atacar o hidróxeno que a histidina acaba de captar. O agora o carbono carbonílico deficiente en electróns refai o dobre enlace co oxíxeno. Como resultado, o C-terminal do péptido é liberado.
Efectos estabilizantes adicionais
editarDescubriuse que outros dous aminoácidos adicionais da protease, a Gly 193 e a Ser 195, están implicados en crear o que se denomina un oco oxianión. Tanto a Gly 193 coma a Ser 195 poden doar hidróxenos do eixe carbonado da molécula para establecer enlaces de hidróxeno. Cando se xeran os intermediarios tetraédricos dos pasos 1 e 3, o ión oxíxeno negativo, que aceptou os electróns do dobre enlace do carbonilo encaixa perfectamente no oco oxianión. De feito, as serina proteases únense preferentemente ao estado de transición e a estrutura global é favorecida, rebaixando a enerxía de activación da reacción. Esta "unión preferente" é responsable de moita da eficiencia catalítica do encima.
Regulación da actividade das serina proteases
editarOs organismos que conteñen estes encimas deben asegurarse que a actividade das serina proteases está ben regulada. Isto conséguese ao ser necesaria unha activación inicial da protease, e pola secreción de inhibidores.
Activación de cimóxenos
editarOs cimóxenos (ou zimóxenos) son os precursores de maior tamaño xeralmente inactivos dun encima, como moitos encimas dixestivos. Se os encimas dixestivos fosen activos desde o momentos en que se sintetizan, empezarían inmediatamente a dixerir os órganos e tecidos que os sintetizaron. A pancreatite aguda é un exemplo de doenza na cal hai unha activación prematura dos encimas dixestivos no páncreas, e como resultado prodúcese autodixestión (autólise). Isto tamén complica as investigacións postmortem, xa que o páncreas a miúido se autodixire antes de que poida ser avaliado visualmente.
Os cimóxenos son grandes estruturas proteicas inactivas, que teñen a capacidade de perder unha parte por clivaxe ou cambiar formando un encima máis pequeno activado. Só despois da activación o sitio activo ten a conformación axeitada para que teña lugar a proteólise.
| Cimóxeno | Encima | Notas |
| Tripsinóxeno | tripsina | Cando o tripsinóxeno entra no intestino delgado procedente do páncreas, unha enteropeptidase segregada pola mucosa duodenal cliva o enlace peptídico entre a lisina 15 e a isoleucina 16 do cimóxeno. Como resultado, o cimóxeno tripsinóxeno rompe dando lugar a tripsina. Como a propia tripsina pode tamén romper enlaces peptídicos nos que intervén a lisina, unha vez que se formou unha pequena cantidade inicial de tripsina, ela mesma pode participar na clivaxe do seu propio cimóxeno e xerar máis tripsina. O proceso da activación da tripsina pode, xa que logo, considerarse autocatalítico. |
| Quimotripsionóxeno | quimotripsina | Unha vez que a tripsina rompe o enlace peptídico entre a arxinina 15 e a isoleucina 16 do cimóxeno quimotripsinóxeno, a nova estrutura xerada chamada pi-quimotripsina sofre autólise, xerando quimotripsina activa. |
| Proelastase | elastase | É activada ao ser clivada pola tripsina. |
Como pode verse, a activación do tripsinóxeno a tripsina é esencial, porque esta activa a súa propia reacción, e tamén a reacción da quimotripsina e a elastase. Por tanto, é esencial que esta activación non ocorra prematuramente. Hai varias medidas de protección no organismo para previr a autodixestión, como son:
- A activación do tripsinóxeno pola tripsina é relativamente lenta.
- Os cimóxenos son almacenados en gránulos de cimóxeno, que son vesículas con paredes que se cre son resistentes á proteólise.
Inhibición
editarHai algúns inhibidores que lembran o intermediario tetraédrico, e poden ocupar o sitio activo do encima, impedindo que este poida funcionar axeitadamente.
As serina proteases son inhibidas por diversos inhibidores, entre os que están inhibidores químicos sintéticos para a investigación ou propósitos terapéuticos, e inhibidores proteicos naturais. Unha familia de inhibidores naturais é a das chamadas "serpinas" (abreviación de inhibidores das serina proteases), que poden formar un enlace covalente coas serina proteases, inhibindo a súa función. As serpinas mellor estudadas son a antitrombina e a alfa 1-antitripsina, estudadas polo seu papel na coagulación/trombose e enfisema/A1AT, respectivamente. Moléculas pequenas artificiais que son inhibidores irreversibles son AEBSF e PMSF.
Identificouse unha familia de inhibidores de serina proteases de artrópodos, chamadas pacifastinas, en insectos e crustáceos, que poden funcionar no sistema inmunitario deses animais.[6]
Papel en enfermidades
editarAs mutacións nestes encimas poden orixinar un incremento ou diminución da súa actividade. Isto pode ter diferentes consecuencias, dependendo da función normal das serina proteases. Por exemplo, as mutacións na proteína C poden causar deficiencia en proteína C e predispoñer á trombose.
Uso en diagnósticos
editarA determinación dos niveis de serina proteases pode ser útil no contexto de determinadas enfermidades.
- Pode pedirse a medida dos factores de coagulación para o diagnóstico de condicións hemorráxicas ou trombóticas.
- A elastase fecal emprégase para determinar a actividade exócrina do páncreas, por exemplo, na fibrose quística ou na pancreatite crónica.
- A medida dos niveis de antíxeno prostático específico sérico utilízase en casos de cancro de próstata.
- As serina proteases están relacionadas cos mastocitos, e son un marcador de diagnóstico importante para as reaccións de hipersensibilidade de tipo 1 (por exemplo a anafilaxe). É máis útil que, por exemplo, a histamina debido á súa maior vida media, o que significa que permanece no sistema durante un período de tempo útil clinicamente.
Notas
editar- ↑ 1,0 1,1 Hedstrom, L. (Dec 2002). "Serine protease mechanism and specificity.". Chem Rev 102 (12): 4501–24. PMID 12475199. doi:10.1021/cr000033x.
- ↑ Madala PK, Tyndall JD, Nall T, Fairlie DP. (Jun 2010). "Update 1 of: Proteases universally recognize beta strands in their active sites". Chem Rev 110 (6): PR1–31. PMID 20377171. doi:10.1021/cr900368a.
- ↑ Ovaere P, Lippens S, Vandenabeele P, Declercq W. (Aug 2009). "The emerging roles of serine protease cascades in the epidermis". Trends Biochem Sci 34 (9): 453–63. PMID 19726197. doi:10.1016/j.tibs.2009.08.001.
- ↑ Evnin, Luke B.; Vásquez, John R.; Craik, Charles S. (1990). "Substrate specificity of trypsin investigated by using a genetic selection". Proceedings of the National Academy of Sciences of the United States of America 87 (17): 6659–63. JSTOR 2355359. PMC 54596. PMID 2204062. doi:10.1073/pnas.87.17.6659.
- ↑ Iván, Gábor.; Szabadka, Zoltán; Ordög, Rafael; Grolmusz, Vince; Náray-Szabó, Gábor (2009). "Four Spatial Points That Define Enzyme Families". Biochemical and Biophysical Research Communications 383 (4): 417–420. PMID 19364497. doi:10.1016/j.bbrc.2009.04.022.
- ↑ Breugelmans B, Simonet G, van Hoef V, Van Soest S, Vanden Broeck J (2009). "Pacifastin-related peptides: structural and functional characteristics of a family of serine peptidase inhibitors.". Peptides 30 (3): 622–32. PMID 18775459. doi:10.1016/j.peptides.2008.07.026.
Véxase tamén
editarOutros artigos
editarLigazóns externas
editar- Base de datos en liña MEROPS para as peptidases e os seus inhibidores: Serine Peptidase Arquivado 04 de abril de 2017 en Wayback Machine.
- Serine Proteases da Saint Louis University (SLU)
- Serine proteases Medical Subject Headings (MeSH) na Biblioteca Nacional de Medicina dos EUA.