Subtilisina
A subtilisina é un encima protease non específico que se obtivo inicialmente do Bacillus subtilis (de aí o seu nome), pero tamén aparecen subtilisinas noutras especies bacterianas.[2][3][4][5][6][7]
| subtilisina | |||||||||
|---|---|---|---|---|---|---|---|---|---|

| |||||||||
| Identificadores | |||||||||
| Número EC | 3.4.21.62 | ||||||||
| Número CAS | 9014-01-1 | ||||||||
| Bases de datos | |||||||||
| IntEnz | vista de IntEnz | ||||||||
| BRENDA | entrada de BRENDA | ||||||||
| ExPASy | vista de NiceZyme | ||||||||
| KEGG | entrada de KEGG | ||||||||
| MetaCyc | vía metabólica | ||||||||
| PRIAM | perfil | ||||||||
| Estruturas PDB | RCSB PDB PDBe PDBj PDBsum | ||||||||
| Gene Ontology | AmiGO / EGO | ||||||||
| |||||||||
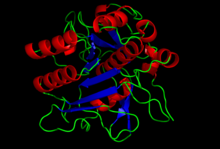
| |
| Estrutura cristalográfica da subtilisina.[1] | |
Subtilisina BPN'
| |
| Identificadores | |
| Símbolo | apr |
| Entrez | 5712479 |
| PDB | 1st2 Máis estruturas |
| UniProt | P00782 |
| Outros datos | |
| Número EC | 3.4.21.62 |
As subtilisinas pertencen ás subtilases, un grupo de serina proteases que, igual que as outras serina proteases, inician o ataque nucleofílico ao enlace peptídico por medio dun residuo de serina situado no sitio activo. As subtilisinas teñen xeralmente pesos moleculares de aproximadamente entre 20.000 e 45.000 daltons. Poden obterse de certos tipos de bacterias do solo, como Bacillus amyloliquefaciens que as segregan en grandes cantidades.
Nomenclatura
editarOutros nomes que se lle deron ás subtilisinas son: alcalase, alcalase 0.6L, alcalase 2.5L, encima ALK, bacilopeptidase A, bacilopeptidase B, proteinase alcalina bioprase de Bacillus subtilis, bioprase AL 15, bioprase APL 30, colistinase, subtilisina J, subtilisina S41, subtilisina Sendai, subtilisina GX, subtilisina E, subtilisina BL, xenenase I, esperase, maxatase, termoase PC 10, protease XXVII, termoase, superase, subtilisina DY, subtilopeptidase, SP 266, savinase 8.0L, savinase 4.0T, kazusase, protease VIII, opticlean, proteinase alcalina de Bacillus subtilis, protina A 3L, savinase, savinase 16.0L, savinase 32.0 L EX, orientase 10B, protease S.
Estrutura
editarA estrutura da subtilisina foi determinada por cristalografía de raios X. É unha proteína globular de 275 residuos con varias hélices alfa, e unha grande folla beta. Non está estruturalmente relacionada co clan da quimotripsina de serina proteases, pero utiliza o mesmo tipo de tríade catalítica no seu sitio activo. Este é un exemplo clásico de evolución converxente.
Mecanismo de catálise
editarO sitio activo presenta unha rede de relevo de carga na que interveñen os residuos Asp-32, His-64, e a Ser-221 do sitio activo dispostos nunha tríade catalítica. A rede de relevo de carga funciona do seguinte xeito: A cadea lateral carboxilato do Asp-32 forma enlaces de hidróxeno co protón unido a nitróxeno do anel de imidazol da His-64. Isto é posible porque o Asp está cargado negativamente a pH fisiolóxico. O outro nitróxeno da His-64 forma enlaces de hidróxeno co protón do O-H da Ser-221. Esta última interacción ten como resultado unha separación de carga de O-H, e o átomo de oxíxeno faise máis nucleofílico. Isto permite que o átomo de oxíxeno da Ser-221 ataque aos substratos entrantes (é dicir, corte os enlaces peptídicos dos péptidos entrantes), axudado pola cadea lateral carboxiamida veciña da Asn-155.
Aínda que os residuos Asp-32, His-64, e Ser-221 están lonxe na secuencia proteica, están xuntos no sitio activo cando a proteína se prega.
Para resumir as interaccións antes descritas, a Ser-221 actúa como un nucleófilo e rompe o enlace peptídico co seu átomo de oxíxeno parcialmente negativo. Isto é posible debido á natureza de relevo de carga do sitio activo da subtilisina.
Aplicacións
editarFerramenta de laboratorio
editarEn bioloxía molecular, cando se usa como organismo modelo a bacteria B. subtilis, o xene que codifica a subtilisina (aprE) é a miúdo o segundo xene que se prefire utilizar, despois do xene amyE, para facer construtos reporteiros integrados, debido á súa prescindibilidade.
Comerciais
editarAs subtilisinas transformadas por enxeñaría xenética utilízanse moito en produtos comerciais (o encima nativo é doadamente inactivado por deterxentes e temperaturas altas) para limpeza en lavandarías[8] deterxentes de lavalouzas, cosméticos, procesamento de alimentos,[9] pomadas para o coidado da pel,[10] limpeza de lentes de contacto, e en investigación en química orgánica sintética.
Notas
editar- ↑ PDB 1st2; Bott R, Ultsch M, Kossiakoff A, Graycar T, Katz B, Power S (June 1988). "The three-dimensional structure of Bacillus amyloliquefaciens subtilisin at 1.8 A and an analysis of the structural consequences of peroxide inactivation". J. Biol. Chem. 263 (16): 7895–906. PMID 3286644.
- ↑ Ottesen Mm, Svendsen I (1970). "The subtilisins". Methods Enzymol. 19: 199–215. doi:10.1016/0076-6879(70)19014-8.
- ↑ Markland FS, Smith EL (1971). "Subtilisins: primary structure, chemical and physical properties". En Boyer PD. The Enzymes 3 (3rd ed.). New York: Academic Press. pp. 561–608.
- ↑ Philipp M, Bender ML (1983). "Kinetics of subtilisin and thiolsubtilisin". Mol. Cell. Biochem. 51 (1): 5–32. PMID 6343835. doi:10.1007/bf00215583.
- ↑ Nedkov P, Oberthür W, Braunitzer G (April 1985). "Determination of the complete amino-acid sequence of subtilisin DY and its comparison with the primary structures of the subtilisins BPN', Carlsberg and amylosacchariticus". Biol. Chem. Hoppe-Seyler 366 (4): 421–30. PMID 3927935. doi:10.1515/bchm3.1985.366.1.421.
- ↑ Ikemura H, Takagi H, Inouye M (June 1987). "Requirement of pro-sequence for the production of active subtilisin E in Escherichia coli". J. Biol. Chem. 262 (16): 7859–64. PMID 3108260.
- ↑ Polgár L (1987). "Structure and function of serine proteases". En Brocklehurst K, Neuberger A. Hydrolytic enzymes. Amsterdam: Elsevier. ISBN 0-444-80886-8.
- ↑ Spar Washing Detergent contents http://www.5010358128009.detergent-info.com/ Arquivado 23 de setembro de 2020 en Wayback Machine.
- ↑ "Copia arquivada". Arquivado dende o orixinal o 26 de setembro de 2008. Consultado o 24 de setembro de 2014.
- ↑ "Copia arquivada". Arquivado dende o orixinal o 03 de febreiro de 2008. Consultado o 24 de setembro de 2014.