Centrómero
En bioloxía o centrómero é o estreitamento ou constrición primaria que separa os brazos dos cromosomas, e que, utilizando tinguiduras tradicionais, aparece menos tinguido ca o resto do cromosoma. É a zona pola que o cromosoma interacciona coas fibras do fuso acromático desde a profase á anafase, tanto na mitose coma na meiose, e é responsable de realizar e regular os movementos cromosómicos que teñen lugar durante estas fases. As estruturas centroméricas que interaccionan coas fibras do fuso denomínanse cinetocoros. Ademais, o centrómero contribúe á nucleación da unión das cromátides irmás. Na estrutura do centrómero interveñen tanto o ADN centromérico coma proteínas centroméricas.
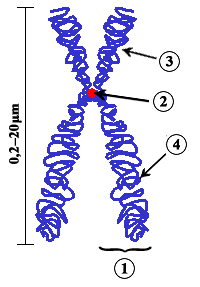
No lévedo (Saccharomyces cerevisiae) o ADN centromérico consta unicamente de 125 pares de bases e está conservado entre os diferentes cromosomas.[1] Porén, o ADN centromérico en metazoos pode constar de megabases, e non contén secuencias consenso doadamente identificables (ver a revisión de Choo en 1997[2]). Malia as diferenzas entre o ADN centromérico de lévedos e metazoos, o cinetocoro ensámblase en ambos os casos sobre nucleosomas centroméricos que conteñen unha forma especializada de histona H3 (Cse4p en lévedos[3] ou o seu homólogo CENP-A en metazoos).
O ADN centromérico organízase en forma de heterocromatina constitutiva, que permanece condensada en case todas as células somáticas dun organismo. Estas rexións son pobres en xenes e poden inducir a represión da expresión xénica das rexións adxacentes de maneira epixenética. Este fenómeno denomínase "variegación por efecto de posición" (en inglés PEV, por Position Effect Variegation).[4] A aparición ocasional de centrómeros de novo (neocentrómeros) suxire que máis que a secuencia do ADN per se, a característica primaria dos centrómeros é a organización estrutural dos dominios centroméricos. A selección do centrómero pode ser tamén o resultado dun complexo número de parámetros, como o momento da súa replicación, a posición dentro do núcleo celular, así como outras características herdables da estrutura da cromatina.
O centrómero ten un comportamento diferente durante a anafase mitótica e a anafase I da meiose, de maneira que durante a anafase mitótica as cromátides irmás se separan e van a polos opostos da célula (segregación anfitélica) entanto que na anafase-I da meiose o que se separa a polos opostos son os cromosomas homólogos completos, cada un constituído por dúas cromátides (segregación sintélica).
Posición do centrómero
editarCada cromosoma posúe dous brazos, un longo (chamado q) e outro curto (chamado p) separados polo centrómero, os cales se conectan de forma metacéntrica, submetacéntrica, acrocéntrica, holocéntrica ou telocéntrica. Esta característica serve para clasificar os cromosomas atendendo á posición do centrómero nos seguintes tipos:
- Metacéntrico. Cromosoma co centrómero situado na metade do cromosoma, dando lugar a brazos de igual lonxitude. No cariotipo humano catro pares de cromosomas son metacéntricos, concretamente o 1, o 3, o 19 e o 20.
- Submetacéntrico. Cromosoma co centrómero algo desprazado do centro, de modo que un dos seus brazos é lixeiramente máis curto ca o outro. A maior parte dos cromosomas humanos son submetacéntricos, en concreto os seguintes pares: 2, 4, 5, 6, 7, 8, 9, 10, 11, 12, 16, 17 e 18 e mais o cromosoma X. Ademais, o cromosoma Y ás veces considérase submetacéntrico tamén, pero outros descríbeno como acrocéntrico sen satélite.
- Acrocéntrico. Cromosoma co centrómero moi próximo a un dos telómeros, cun brazo moi curto (p) e o outro longo (q). Son deste tipo os pares de cromosomas humanos 13, 14, 15, 21 e o 22 e actúan como organizadores nucleolares.
- Subtelocéntrico (e telocéntrico). Aínda que o concepto de cromosoma telocéntrico (centrómero no extremo) é amplamente aceptado e utilizado entre a comunidade científica[5], realmente, un cromosoma telocéntrico como tal non existe. Supostamente neste tipo de cromosomas o centrómero está localizado nun extremo do mesmo, mais a rexión telocéntrica non permite que molecularmente haxa outra estrutura ao final do cromosoma, polo que o telómero sempre está ao final. De feito, o acurtamento do telómero ou a súa ausencia total causa inestabilidade nos cromosomas e unha translocación robertsoniana.[6] Xa que logo, o termo telocéntrico é incorrecto e debe considerarse mellor o termo subtelocéntrico, o cal implica que o telómero (e non o centrómero) se sitúa ao final, aínda que non sexa visible, e que o centrómero está xusto despois. Ningún dos cromosomas humanos presenta esta característica; pero, por exemplo, os 40 cromosomas do rato común son subtelocéntricos.[7][8]
Ademais dos tipos anteriores, existen cromosomas onde non se observa un centrómero ben definido, que son do seguinte tipo:
- Holocéntrico. Nos cromosomas holocéntricos toda a lonxitude do cromosoma actúa como centrómero, xa que non teñen constrición primaria nin un lugar concreto para a ensamblaxe do cinetocoro, o cal se ensambla de forma difusa por toda a lonxitude do cromosoma. Exemplos deste tipo de centrómero non se atopan no cariotipo humano, pero poden atoparse en diversas especies animais e vexetais.[9] O exemplo mellor coñecido é o do verme nematodo Caenorhabditis elegans.
A secuencia centromérica
editarHai dous tipos de centrómero: rexionais e puntuais.[10]
Os centrómeros rexionais son de gran lonxitude. Neles as secuencias de ADN contribúen á súa función pero non a determinan. Os centrómero rexionais conteñen grandes cantidades de ADN e están en xeral empaquetados formando heterocromatina. Na maioría dos eucariotas este centrómero non ten unha secuencia definida de ADN. Consiste tipicamente en grandes seccións de ADN repetitivo (por exemplo, ADN satélite) nos que a secuencia observada nos elementos repetidos individuais é similar mais non idéntica. Nos seres humanos, a unidade de repetición centromérica primaria chámase α-satélite (ou alfoide), aínda que se atoparon tamén outros tipos de secuencias nesta rexión.
Os centrómeros puntuais son máis pequenos e máis compactos. Neste caso as secuencias de ADN son necesarias e suficientes para especificar a identidade e a función do centrómero nos organismos que os teñen. Nos lévedos de xemación a rexión do centrómero é relativamente pequena (duns 125 pares de bases) e contén dúas secuencias de ADN moi conservadas que serven como sitios de unión para proteínas cinetocóricas esenciais.
Herdanza
editarDado que a secuencia do ADN non é o determinante na especificación dos centrómeros en metazoos, pénsase que a herdanza epixenética xoga un papel esencial na especificación do centrómero.[11] Os cromosomas fillos forman os centrómeros no mesmo lugar cós cromosomas parentais, independentemente da secuencia. Suxírese que a histona H3 na súa variante CENP-A (Proteína do Centrómero A) é a marca epixenética dos centrómeros.[12] A pregunta a responder é se hai algunha determinada vía pola cal se especifica o centrómero, mesmo se é ademais herdado epixeneticamente. Se o centrómero se herda epixeneticamente dunha xeración á seguinte, o problema hai que retrotraelo á orixe dos primeiros metazoos.
Estrutura do centrómero
editarO ADN centromérico está normalmente en estado de heterocromatina, o cal é esencial para o recrutamento do complexo proteico da cohesina, que media no establecemento da cohesión entre as cromátides irmás despois da replicación do ADN, e na coordinación da separación das cromátides irmás durante a anafase. Nesta cromatina a histona normal H3 foi substituída por unha variante súa específica do centrómero, a CENP-A no caso humano.[13] Crese que a presenza da CENP-A é importante para a ensamblaxe do cinetocoro no centrómero. Demostrouse que a CENP-C se localiza case exclusivamente nas rexións da cromatina asociadas á CENP-A. Nas células humanas encontrouse que as histonas están máis enriquecidas en H4K20me3 e H3K9me3,[14] o cal se coñece como modificacións heterocromáticas.
No lévedo Schizosaccharomyces pombe (e probablemente noutros eucariotas), a formación de heterocromatina centromérica está relacionada co RNAi.[15] En nematodos como Caenorhabditis elegans, nalgunhas plantas e nalgúns ordes de insectos como Lepidópteros e Hemípteros, os cromosomas son "holocentricos", o que significa que non hai un sitio primario para a unión dos microtúbulos nin unha constrición primaria, e ao longo de toda a lonxitude do cromosoma vaise formar un cinetocoro "difuso".
Aberracións centroméricas
editarNos humanos, en raros casos, poden formarse neocentrómeros en lugares novos do cromosoma. Identificáronse ata agora uns 90 neocentrómeros humanos en 20 cromosomas distintos.[16][17] A formación dun neocentrómero debe estar acompañada ou seguida ou precedida pola inactivación do centrómero, xa que un cromosoma con dous centrómeros funcionais (cromosoma dicéntrico) sufriría unha rotura durante a mitose. Nalgúns casos raros observouse que os neocentrómeros humanos se formaban espontaneamente sobre cromosomas fragmentados. Algunhas destas novas posicións dos centrómeros eran orixinalmente eucromáticas e á vez carecían do ADN satélite alfa.
As proteínas do centrómero son tamén a diana autoantixénica para algúns anticorpos antinucleares, como os anticorpos anticentrómero.
Centrómeros en metazoos
editarA determinación dos centrómeros de metazoos constitúe unha tarefa difícil. En animais e plantas, os centrómeros están incluídos en rexións de ADN satélite altamente repetido, que é difícil analizar mesmo cos métodos de mapeo máis potentes. Estas rexións de ADN satélite están incluídas en rexións de heterocromatina constitutiva, que se mantén silenciada na maior parte das células somáticas dun organismo. A ausencia maioritaria de xenes activos nas rexións centroméricas é unha característica que parece que se adquiriu progresivamente a través da evolución.[18]
ADN satélite
editarEn Drosophila melanogaster a secuencia AATAACATAG está repetida en tándem en rexións próximas ao centrómero. Dado que estas secuencias curtas de 10 pb non son representativas do xenoma dunha especie, adoita suceder que o seu contido en G+C é diferente ao contido en G+C do resto do xenoma. Isto fai que cando o ADN dunha especie eucariota se centrifuga en gradiente de densidade de cloruro de cesio, apareza unha banda principal que contén a maior parte do ADN da especie e unha banda satélite (minoritaria) que está formada por unha secuencia curta de ADN repetida en tándem. O ADN satélite nalgunhas especies ten maior densidade que o ADN principal (maior contido en G+C) e noutras especies ten menor densidade e, xa que logo, menor contido en G+C que o ADN principal. Cando o ADN satélite de rato se marca radioactivamente e se realiza unha hibridación in situ co ADN de cromosomas metafásicos mitóticos, obsérvase que a marcaxe radioactiva (hibridación) se produce en rexións próximas ao centrómero. O ADN satélite tamén se denomina α-satélite e é un dos compoñentes do xenoma eucariótico que evoluciona máis rapidamente.[19]
Aínda que na maior parte dos casos non se detectaron motivos especialmente definidos, unha secuencia candidata no ADN satélite tería que estar repetida en centos de quilobases (kb), xa que este é o tamaño mínimo dun centrómero funcional, identificado en diferentes organismos. Por exemplo, en Drosophila a unidade mínima necesaria de repeticións en tándem é de 420 kb, no millo necesítanse 500 kb e en humanos a unidade mínima consta de 100 kb. Unha característica interesante da maior parte do ADN satélite é a súa unidade de lonxitude, xa que aínda que non se detectaron secuencias con motivos conservados, a lonxitude da unidade que se repite é moi parecida entre organismos. En primates, por exemplo, a unidade básica que se repite ten 171 pares de bases (pb), no peixe Sparus aurata (dourada) a repetición centromérica ten 186 pb, no insecto Chironomus pallidivittatus ten 155 pb, en Arabidopsis thaliana e no millo ten 180 pb, e no arroz 168 pb.[21] A estreita variación en lonxitude da unidade que se repite no ADN satélite corresponde aproximadamente ao rango de lonxitude do ADN que rodea a un nucleosoma, e repeticións máis longas, como as que se encontran nos centrómeros do porco (340 pares de bases) corresponden aproximadamente á lonxitude dos nucleosomas. Hai excepcións notables, como as repeticións pentaméricas que se atopan en Drosophila melanogaster. En xeral, a selección da lonxitude dun nucleosoma podería limitar a evolución do ADN centromérico, de acordo coa súa función estrutural no xenoma.
Estas unidades mínimas (denominadas monómeros) encóntranse normalmente asociadas de forma cabeza-cola. Nas rexións centroméricas do núcleo funcional, o ADN satélite organízase nunha unidade repetida que consta de múltiples monómeros. A unidade multimonomérica repítese á súa vez moitas veces, xerando un vector (array) de nivel superior. Os vectores de nivel superior de ADN satélite son a organización típica das rexións centroméricas humanas e esténdense a través de megabases de ADN que non se encontran maioritariamente interrompidas por ningún tipo de inserción ou mutación. Por tanto, animais e plantas presentan centrómeros "rexionais", fronte aos centrómeros "puntuais" típicos de lévedos.
Porén, malia o feito de que o ADN satélite se atopa nos centrómeros humanos nativos, detectáronse centrómeros humanos xerados de novo (neocentrómeros) que carecen de α-satélite ou outras repeticións en tándem,[22] o que indica que o ADN satélite non é fundamental para definir un centrómero funcional.
Proteínas centroméricas
editar- Artigo principal: Cinetocoro.
Unha característica conservada e herdable dos centrómeros é a presenza nos seus nucleosomas dunha variante especial da histona H3, que se encontra unicamente no núcleo da rexión centromérica. Esta histona específica dos centrómeros denomínase CENP-A en mamíferos (centromeric protein A), Cid (centromere identifier) en Drosophila, Cse4 en Saccharomyces cerevisiae e Cnp1 en Schizosaccharomyces pombe (revisado por Choo en 2001[23]). A presenza desta variante da histona H3 parece ser fundamental para a ensamblaxe do cinetocoro e distingue a placa interna do cinetocoro da heterocromatina pericéntrica, que contén a histona H3 normal. CENP-A presenta algunhas características que a diferencian da histona H3 normal, como unha cola NH2-terminal non canónica, un pregamiento diverxente e unha rexión lazo 1 máis longa.[24] Aínda que a histona H3 está sometida a unha forte selección evolutiva, as histonas centroméricas son sorprendentemente diverxentes. Esta diferenza podería deberse á necesidade que ten H3 de interaccionar con todo o xenoma, mentres que a variante centromérica só necesita interaccionar co ADN centromérico correspondente. Este ADN está formado por ADN satélite, que é un dos compoñentes do xenoma eucariótico que evoluciona máis rapidamente.[19] Propúxose que a interacción entre a histona centromérica e o ADN centromérico é responsable da lonxitude similar á que rodea un nucleosoma das repeticións do ADN satélite.[21]
Un estudo realizado en Drosophila identificou que as rexións centrais dos centrómeros se replican como dominios illados en estadios temperáns da fase S, antes da replicación da heterocromatina pericéntrica, que se replica de forma tardía.[25] Se no momento en que se replican os centrómeros, a rexión do núcleo na que se localizan os centrómeros exclúe a histona H3 pero secuestra a histona centromérica, a compartimentalización aseguraría que só CENP-A estea dispoñible para a ensamblaxe da cromatina centromérica.[21] Este modelo apóiase en varias liñas de evidencias en diferentes organismos.
Ademais de CENP-A, identificáronse outros compoñentes constitutivos no centrómero. Un deles é CENP-C, que está conservado evolutivamente, aínda que só comparte un motivo de 20 aminoácidos co seu homólogo en S. cerevisiae, Mif2. Tamén se atoparon homólogos noutras especies, como HCP-4 en C. elegans. A localización centromérica de CENP-C depende de CENP-A e suxeriuse que CENP-C podería interaccionar coa estrutura da cromatina alterada por CENP-A. Porén, aínda que a zona de unión de CENP-C ao ADN se mapeou na zona central da proteína, non se identificaou unha secuencia específica de unión. Parece ademais que CENP-C presenta a capacidade de unirse a ARN de forma específica,[26] aínda que a contribución destas capacidades á localización de CENP-C non está clara.[20]
CENP-B é a única proteína centromérica que se une a unha secuencia de ADN específica de 17 pares de bases (a chamada "caixa CENP-B"), que se encontra nun subconxunto de monómeros de α-satélite. A función de CENP-B nos centrómeros non está clara, xa que a diferenza con CENP-A ou CENP-C, CENP-B non é esencial para a función mitótica (de feito, o rato knockout para CENP-B é viable). CENP-B pode estar presente tanto en centrómeros activos coma inactivos, o que suxire que non está asociada simplemente á función centromérica.[20] Ademais, algúns centrómeros funcionais carecen de caixas CENP-B (como o centrómero do cromosoma Y, por exemplo). Porén, está demostrado que a unión de CENP-B aumenta a eficiencia da unión de CENP-A en cromosomas humanos artificiais. A pesar de non ser esencial en humanos, CENP-B está conservada a través de varios filos e S. pombe presenta tres homólogos que si son esenciais para a viabilidade celular.
Estas tres proteínas centroméricas están organizadas de forma diferente no centrómero humano. CENP-B está presente en todo o vector de nivel superior, mentres que CENP-A e CENP-C se encontran só nalgunos bloques de unidades repetidas, intercalados con bloques que conteñen nucleosomas canónicos (que inclúen a histona H3) e con modificacións de histonas máis características de eucromatina que de heterocromatina. Crese que eses bloques de CENP-A se auto-organizan para presentar unha "superficie" combinada que organiza o resto das proteínas do cinetocoro, que servirá como sitio de ancoraxe dos microtúbulos.[20] Considérase ademais que os monómeros pericéntricos que están a cada lado das repeticións centroméricas están xeralmente desprovistos de proteínas centroméricas, e están empaquetados en nucleosomas canónicos que posúen modificacións de histonas características de heterocromatina, e unidos a proteínas específicas de heterocromatina como HP1. Esta heterocromatina pericéntrica é importante tanto para definir os límites dos dominios centroméricos coma para recrutar as cohesinas que manterán unidas as cromátides irmás ata a anafase durante o ciclo celular.
ADN centromérico e evolución
editarO proceso de segregación cromosómica está sometido a unha forte presión evolutiva, dado que perder ou gañar cromosomas (unha situación denominada aneuploidía) pode producir importantes alteracións fenotípicas, como a síndrome de Down en humanos, por exemplo. Por esa razón a maquinaria encargada de distribuír os cromosomas entre as células fillas durante a división celular presenta unha gran sofisticación e está sometida a un estrito control. Os centrómeros son as rexións cromosómicas sobre as que se ensamblan os cinetocoros, que son as estruturas proteicas responsables da ancoraxe dos cromosomas ao fuso mitótico, e, por tanto, a zona responsable do movemento cromosómico e a súa regulación. Porén, a pesar de todo as secuencias de ADN que definen as secuencias centroméricas están moi pouco conservadas e evolucionan rapidamente mesmo entre especies moi relacionadas. Isto non quere dicir que as secuencias de ADN do centrómero son hipermutables, senón que as variantes das secuencias se fixan por expansión e contracción, e poden aparecer de novo en sitios novos (neocentrómeros). Estes cambios no ADN centromérico teñen lugar debido á existencia de diferentes procesos mutacionais, como erros na replicación do ADN, intercambio desequilibrado, transposición e excisión. As proteínas centroméricas tamén presentan signos inesperados dunha rápida evolución. En consecuencia, tense suxerido que no núcleo desta rápida evolución existe un conflito xenético en funcionamento.[21]
Parece ser que a arquitectura nun vector (array) de nivel superior que se observa nos centrómeros de humanos puido aparecer recentemente nun centrómero na evolución dos primates (arrededor da separación gorila-orangután) e estendeuse aos outros cromosomas vía transposición.[27] Posteriormente, os intercambios desiguaies ou conversións xénicas amplificaron os vectores de nivel superior, dando lugar á arquitectura en vectores centroméricos de nivel superior que é específica da especie humana e que se observa en diferentes cromosomas humanos. Ademais, orixináronse algunhas variantes por mutación que se fixaron nalgúns centrómeros.[20] A comparación de unidades monoméricas e unidades vectoriais de nivel superior que se encontran nos centrómeros de cromosomas ortólogos (por exemplo, entre chimpancés e humanos) levou ao descubrimento sorprendente de que os vectores centroméricos de diferentes especies son máis diverxentes entre si do que as unidades pericéntricas.[28] Esta observación é anti-intuitiva, porque o vector de ADN satélite centromérico é o centrómero funcional e está sometido a unha forte presión selectiva, entanto que as rexións de heterocromatina pericéntrica non o están. Por tanto, a observación é paradoxal: as unidades de ADN satélite que están fortemente limitadas dentro dunha especie evolucionaron rapidamente entre especies.
Este paradoxo levou a pensar que algunha forza selectiva debe dirixir a rápida fixación das mutacións nos vectores centroméricos, impoñendo unha presión para manter as mutacións, incrementando desta forma as taxas de mutación do vector completo. Suxeriuse que esta forza selectiva pode ser a vantaxe conferida aos centrómeros durante a meiose feminina, ou "deriva-centromérica":[21] novas variacións na secuencia de α-satélite, unha nova organización ou simplemente un incremento na cantidade de α-satélite fornece unha maior oportunidade de incorporación da proteína histónica CENP-A (que intervén na ensamblaxe do cinetocoro) e, por tanto, unha maior capacidade para a unión de microtúbulos. A asimetría da tétrade meiótica feminina proporciona unha oportunidade para os cromosomas de competir por ser incluído no núcleo do ovocito por medio dunha orientación favorable durante a meiose. Os centrómeros que aproveitan esta oportunidade na meiose I "gañan", e unha lixeira vantaxe en cada meiose feminina é de abondo para fixar unha variación centromérica favorable.[29]
Como contrapartida, ben que a deriva centromérica pode xerar unha vantaxe selectiva na meiose feminina, pode producir defectos na meiose masculina, pois neste caso un centrómero mutado se emparellará con outro normal, xerándose unha diferenza de tensión que pode activar o punto de control da mitose, provocando a morte celular e unha diminución da fertilidade masculina. Unha forma de contrarrestar este efecto na meiose masculina sería a aparición de mutacións nas proteínas centroméricas con alteración na súa capacidade de unión ao ADN e que equilibraran a tensión centromérica. A proteína candidata máis probable é CENP-A.
Se este proceso ten lugar en dúas poboacións illadas da mesma especie, as configuracións do ADN satélite e CENP-A diverxirán rapidamente. En cada poboación, CENP-A evolucionará para suprimir os efectos deletéreos da evolución do ADN satélite. Desta forma, as novas variantes de CENP-A resultarán incompatibles co ADN satélite da outra poboación. Cruces entre ambas as poboacións orixinarán defectos nos híbridos. Por tanto, o proceso evolutivo entre CENP-A e o ADN satélite dá lugar ao comezo do illamento reprodutivo entre as dúas poboacións. Isto quere dicir que a evolución centromérica ten como consecuencia inevitable a especiación.[21]
Notas
editar- ↑ Fitzgerald-hayes M., Clarke L., Carbon J. (1982). "Nucleotide sequence comparisons and functional analysis of yeast centromere DNAs". Cell (1 ed.) 29: 235–44. doi:10.1016/0092-8674(82)90108-8.
- ↑ Choo K.H.A. (1997). "The centromere".
- ↑ Meluh P.B., Yang P., Glowczewski L., Koshland D., Smith M.M. (1998). "Cse 4 P is a Component of the Core Centromere of Saccharomyces cerevisiae". Cell(Cambridge) (5 ed.) 94: 607–613. Arquivado dende o orixinal o 09 de xullo de 2009. Consultado o 22 de maio de 2011.
- ↑ Perrod S., Gasser S.M. (2003). "Long-range silencing and position effects at telomeres and centromeres: parallels and differences" (PDF). Cellular and Molecular Life Sciences (CMLS) (11 ed.) 60: 2303–2318. doi:10.1007/s00018-003-3246-x. Arquivado dende o orixinal (PDF) o 05 de febreiro de 2009. Consultado o 22 de maio de 2011.
- ↑ "On the origin of telocentric chromosomes in mammals" Original Research Article. Hirotami T. Imai. Journal of Theoretical Biology, Volume 71, Issue 4, 20 de abril de 1978, páxinas 619-637
- ↑ "Telomere length and telomere-centromere relationships?" Original Research Article. P. Slijepcevic. Mutation Research/Fundamental and Molecular Mechanisms of Mutagenesis, Volume 404, Issues 1-2, 3 August 1998, Pages 215-220
- ↑ Silver, Lee M. (1995). "Karyotypes, Chromosomes, and Translocations". Mouse Genetics: Concepts and Applications. Oxford: Oxford University Press. pp. 83–92. ISBN 978-0-19-507554-0.
- ↑ Chinwalla, Asif T.; Cook, Lisa L.; Delehaunty, Kimberly D.; Fewell, Ginger A.; Fulton, Lucinda A.; Fulton, Robert S.; Graves, Tina A.; Hillier, Ladeana W.; et al. (2002). "Initial sequencing and comparative analysis of the mouse genome". Nature 420 (6915): 520–62. PMID 12466850. doi:10.1038/nature01262.
- ↑ Dernburg, A. F. (2001). "Here, There, and Everywhere: Kinetochore Function on Holocentric Chromosomes". The Journal of Cell Biology 153 (6): F33–8. PMC 2192025. PMID 11402076. doi:10.1083/jcb.153.6.F33.
- ↑ Pluta, A. F.; MacKay, A. M.; Ainsztein, A. M.; Goldberg, I. G.; Earnshaw, W. C. (1995). "The Centromere: Hub of Chromosomal Activities". Science 270 (5242): 1591–4. PMID 7502067. doi:10.1126/science.270.5242.1591.
- ↑ Dalal, Yamini (2009). "Epigenetic specification of centromeres". Biochemistry and Cell Biology 87 (1): 273–82. PMID 19234541. doi:10.1139/O08-135.
- ↑ Bernad, Rafael; Sánchez, Patricia; Losada, Ana (2009). "Epigenetic specification of centromeres by CENP-A". Experimental Cell Research 315 (19): 3233–41. PMID 19660450. doi:10.1016/j.yexcr.2009.07.023.
- ↑ Chueh, A. C.; Wong, LH; Wong, N; Choo, KH (2004). "Variable and hierarchical size distribution of L1-retroelement-enriched CENP-A clusters within a functional human neocentromere". Human Molecular Genetics 14 (1): 85–93. PMID 15537667. doi:10.1093/hmg/ddi008.
- ↑ Rosenfeld, Jeffrey A; Wang, Zhibin; Schones, Dustin E; Zhao, Keji; Desalle, Rob; Zhang, Michael Q (2009). "Determination of enriched histone modifications in non-genic portions of the human genome". BMC Genomics 10: 143. PMC 2667539. PMID 19335899. doi:10.1186/1471-2164-10-143.
- ↑ Volpe, T. A.; Kidner, C; Hall, IM; Teng, G; Grewal, SI; Martienssen, RA (2002). "Regulation of Heterochromatic Silencing and Histone H3 Lysine-9 Methylation by RNAi". Science 297 (5588): 1833–7. PMID 12193640. doi:10.1126/science.1074973.
- ↑ Marshall, Owen J.; Chueh, Anderly C.; Wong, Lee H.; Choo, K.H. Andy (2008). "Neocentromeres: New Insights into Centromere Structure, Disease Development, and Karyotype Evolution". The American Journal of Human Genetics 82 (2): 261–82. doi:10.1016/j.ajhg.2007.11.009.
- ↑ Warburton, Peter E. (2004). "Chromosomal dynamics of human neocentromere formation". Chromosome Research 12 (6): 617–26. PMID 15289667. doi:10.1023/B:CHRO.0000036585.44138.4b.
- ↑ Nagaki = K., Cheng Z., Ouyang S., Talbert P.B., Kim M., Jones K.M., Henikoff S., Buell C.R., Jiang J. (2004). "Sequencing of a rice centromere uncovers active genes". Nature Genetics 36: 138–145. doi:10.1038/ng1289.
- ↑ 19,0 19,1 Csink A.K., Henikoff S. (1998). "Something from nothing: the evolution and utility of satellite repeats". Trends in Genetics (5 ed.) 14: 200–204. doi:10.1016/S0168-9525(98)01444-9.
- ↑ 20,0 20,1 20,2 20,3 20,4 Bayes, Joshua J.; Malik, Harmit S. (abril de 2008). "The Evolution of Centromeric DNA Sequences". En John Wiley & Sons, Ltd: Chichester. Encyclopedia of Life Sciences (ELS). [1] Arquivado 09 de decembro de 2008 en Wayback Machine. DOI: 10.1002/9780470015902.a0020827
- ↑ 21,0 21,1 21,2 21,3 21,4 21,5 Henikoff Steven, Ahmad Kami, Malik Harmit S. (2001). "The Centromere Paradox: Stable Inheritance with Rapidly Evolving DNA" (PDF). Science (5532 ed.) 293: 1098–1102. PMID 11498581. doi:10.1126/science.1062939.
- ↑ Lo A.W.I., Craig J.M., Saffery R., Kalitsis P., Irvine D.V., Earle E., Magliano D.J., Choo K.H.A. (2001). "A 330 kb CENP-A binding domain and altered replication timing at a human neocentromere". The EMBO Journal 20: 2087–2096. doi:10.1093/emboj/20.8.2087. Arquivado dende o orixinal o 06/03/2019. Consultado o 02/03/2019.
- ↑ Choo K.H.A. (2001). "Domain Organization at the Centromere and Neocentromere". Developmental Cell (2 ed.) 1: 165–177. doi:10.1016/S1534-5807(01)00028-4.
- ↑ Malik H.S., Henikoff S. (2001). "Adaptive Evolution of Cid, a Centromere-Specific Histone in Drosophila". Genetics (3 ed.) 157: 1293–1298.
- ↑ Ahmad K., Henikoff S. (2001). "Centromeres Are Specialized Replication Domains in Heterochromatin". The Journal of Cell Biology (1 ed.) 153: 101–110.
- ↑ Wong L.H., Brettingham-moore K.H., Chan L., Quach J.M., Anderson M.A., Northrop E.L., Hannan R., Saffery R., Shaw M.L., Williams E., Others (2007). "Centromere RNA is a key component for the assembly of nucleoproteins at the nucleolus and centromere". Genome Research (8 ed.) 17: 1146. doi:10.1101/gr.6022807.
- ↑ Schueler M.G., Sullivan B.A. (2006). "Structural and Functional Dynamics of Human Centromeric Chromatin" (PDF). Annual Reviews of Genomics and Human Genetics 77: 301–313. Arquivado dende o orixinal (PDF) o 13 de xuño de 2010. Consultado o 22 de maio de 2011.
- ↑ Rudd M.K., Wray G.A., Willard H.F. (2006). "The evolutionary dynamics of α-satellite" (PDF). Genome Res 16: 88–96. doi:10.1101/gr.3810906.
- ↑ Fishman L., Saunders A. (2008). "Centromere-Associated Female Meiotic Drive Entails Male Fitness Costs in Monkeyflowers". Science 322 (5907). 1559 - 1562. [2]
Véxase tamén
editarBibliografía
editar- Alberts, Bruce, et al. Molecular Biology of the Cell, 4th ed. Nova York: Garland Publishing, 2000.
- Atherly, Alan G., Jack R. Girton, e John F. McDonald. The Science of Genetics. Philadelphia, PA: Saunders College Publishing, 1998.
- Cooper, D. N. Human Gene Mutation. Bios Scientific Publishers Ltd., 1997.
- Harvey Lodish et al Lodish, Harvey; Berk, Arnold; Kaiser, Chris A.; Krieger, Monty; Scott, Matthew P.; Bretscher, Anthony; Ploegh, Hiddle; Matsudaira, Paul (2008). Molecular Cell Biology (6th ed.). New York: W.H. Freeman. ISBN 978-0-7167-7601-7.
Outros artigos
editarLigazóns externas
editar- Departamento de Genética. Facultad de Genética. Universidad Complutense de Madrid. Centrómero. [3] Arquivado 16 de marzo de 2011 en Wayback Machine.
- Nagaki, Kiyotaka; Cheng, Zhukuan; Ouyang, Shu; Talbert, Paul B; Kim, Mary; Jones, Kristine M; Henikoff, Steven; Buell, C Robin; Jiang, Jiming (2004). "Sequencing of a rice centromere uncovers active genes". Nature Genetics 36 (2): 138–45. PMID 14716315. doi:10.1038/ng1289. Resumo divulgativo – Science Daily (13 de xaneiro de 2004).