Cromatina
A cromatina é o conxunto formado polo ADN, histonas e proteínas non histónicas que se encontra no núcleo das células eucariotas, visible ao microscopio electrónico como fibras finas enredadas durante a interfase do ciclo celular, e que durante o período de división celular constitúe o cromosoma eucariótico. Segundo o seu grao de empaquetamento a cromatina pode aparecer en tres estados, sucesivamente máis empaquetados, que son:
- A fibra de 10 nm de grosor ou "colar de doas".
- A fibra de 30 nm ou "solenoide".
- Os cromosomas mitóticos.
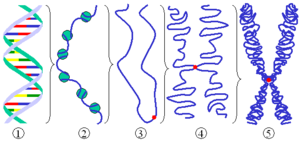

Hai algunhas excepcións a este esquema, xa que os espermatozoides e os glóbuos vermellos das aves presentan unha cromatina moito máis densamente enmpaquetada ca a da maioría das células eucariotas e os protozoos tripanosomátidos non condensan a cromatina a cromosomas durante a mitose.
As unidades básicas da cromatina son os nucleosomas. Estes están formados por ADN de aproximadamente 146 pares de bases de lonxitude (o número depende do organismo), asociado a un complexo específico de 8 histonas nucleosómicas (octámero de histonas). Toda a fibra de cromatina é unha sucesión de nucleosomas. Cada nucleosoma ten unha forma de disco, cun diámetro de 10-11 nm e contén dúas copias de cada unha das 4 histonas H3, H4, H2A e H2B. Este octámero forma un núcleo proteico arrededor do que se enrola a hélice de ADN (dá aproximadamente 1,8 voltas). Entre cada unha das asociacións de ADN e histonas existe un ADN libre chamado ADN "espazador", de lonxitude variable entre 0 e 80 pares de nucleótidos que lle dá flexibilidade á fibra de cromatina. Este tipo de organización, permite unha primeira compactación do material xenético, e dá lugar a unha estrutura semellante a un "colar de doas".
Posteriormente, un segundo nivel de organización de orde superior constitúeo a "fibra de 30nm" composta por grupos de nucleosomas empaquetados uns sobre outros adoptando disposicións regulares grazas á acción da histona H1.
Finalmente, se a célula se vai dividir, continúa o incremento do empaquetamiento do ADN ata obter os cromosomas que observamos na mitose, que acadan o seu maior grosor na metafase, momento no que se chega ao máximo nivel de condensación do ADN.
Estados
editarFibra cromatínica de 10 nm: o "colar de doas"
editarO elemento básico que se repite na cromatina é o nucleosoma. Os nucleosomas conéctanse uns a outros por seccións de ADN espazador (linker), formando unha estrutura moito máis curta e máis grosa ca a do ADN puro en solución.
Ademais das histonas do octámero, existen tamén as histonas de unión ou H1, que fixan os puntos de saída e entrada das fibras do ADN no nucleosoma. O conxunto formado polo octámero ou core do nucleosoma e a histona H1, recibe o nome de cromatosoma. Os nucleosomas, xunto cuns 20 a 60 pares de bases de ADN espazador, poden formar, en condicións non fisiolóxicas, unha fibra de aproximadmente 10 nm de grosor, coñecida como "colar de doas" ou "colar de perlas".
Os nucleosomas únense ao ADN non especificamente. Hai, non obstante, longas secuencias de ADN que determinan de forma preferente a posición dos nucleosomas. Isto débese primordialmente ás variadas propiedades físicas que presentan as diferentes secuencias de ADN: Por exemplo, a adenina e a timina poden ser máis facilmente comprimidas no suco menor do ADN, e o ADN rota a intervalos determinados para maximizar o número de adeninas e timinas que se situarán no suco menor interno do ADN.
Fibra cromatínica de 30 nm: o "solenoide"
editarEsquerda: 1 estrutura en "solenoide".
Dereita: 2 estrutura coa hélice máis solta.
Nota: as histonas non se representaron neste esquema ; só se mostra o ADN.
Cando se engade a histona H1 a estrutura en "colar de doas" retórcese sobre si mesma orixinando unha hélice, que é a fibra ou filamento de 30 nm de diámetro. A estrutura precisa da fibra de cromatina na célula non se coñece en detalle, e aínda hai algunha discusión sobre o asunto.
Este nivel estrutural da cromatina pénsase que é o que ten a eucromatina, a cal contén xenes activos. Diversos estudos de microscopía electrónica demostraron que a fibra de 30 nm é moi dinámica, polo que se pode desenrolar dando a fibra de 10 nm (o "colar de doas") cando unha ARN polimerase está facendo a transcrición.
Debuxouse o ADN espazador en amarelo e o ADN do nucleosoma en rosa.
Os modelos que se teñen elaborado aceptan xeralmente que os nucleosomas están dispostos perpendicularmente ao eixe da fibra, coas histonas H1 situadas internamente. A estabilidade da fibra de 30 nm depende da colocación regular dos nucleosomas ao longo do ADN. O ADN espazador é relativamente resistente ao pregamento e á rotación. Isto fai que a lonxitude do ADN espazador sexa fundamental para a estabilidade da fibra, xa que se require que os nucleosomas estean separados por tramos cuxa lonxitude permita a rotación e pregamento na correcta orientación e sen causar excesiva tensión no ADN. Deste modo, ADNs espazadores de distintas lonxitudes producirían diferentes topoloxías de pregamento na fibra cromatínica. Algúns traballos teóricos recentes, baseados en imaxes de microscopía electrónica[1] de fibras reconstituídas apoian esta idea.[2]
Tipos de cromatina: eucromatina e heterocromatina
editarA fibra cromatínica pode ser de 10 ou de 30 nm pero estas fibras poden atoparse no núcleo en dúas formas: máis condensada (heterocromatina) ou menos condensada (eucromatina).
- Eucromatina. Está espallada polo núcleo en zonas de aspecto máis claro ca as ocupadas pola heterocromatina, debido á súa menor condensación. Tínguese feblemente cos colorantes (a súa maior intensidade obsérvase na mitose e non é visible co microscopio óptico). Representa a forma activa da cromatina na que se está transcribindo o material xenético das moléculas de ADN a moléculas de ARNm, polo que é aquí onde se encontran a maioría dos xenes activos. A eucromatina en transcrición está formada por unha fibra dun diámetro que corresponde ao do nucleosoma. Na eucromatina inactiva (que non transcribe), esta fibra enrólase sobre si mesma grazas ás histonas H1 para formar o solenoide ou fibra de 30 nm. Pode pasarse da fibra de 10 á de 30 e viceversa. A interacción con outras proteínas non histonas (topoisomerase II, proteínas de armazón, lamininas…) pode provocar maiores graos de organización.
- Heterocromatina. É unha forma inactiva condensada localizada sobre todo na periferia do núcleo, que se tingue fortemente cos colorantes. En 1928 Emil Heitz, baseándose en observacións histolóxicas, definiu a heterocromatina (HC) como os segmentos cromosómicos que aparecían moi condensados e escuros no núcleo en interfase. De feito, a cromatina está formada por un enguedello de fibras de diámetro variable segundo o momento do ciclo celular e a rexión do cromosoma observada. A fibra que constitúe a heterocromatina encóntrase máis condensada ca a da eucromatina e a miúdo aparece formada por agregados. A súa formación require numerosas proteínas adicionais, que inclúen as proteínas HP1 (Heterochromatin Protein 1 ou proteína da heterocromatina 1). A riqueza en ADN satélite determina tanto a natureza permanente ou reversible da heterocromatina, como o seu polimorfismo e propiedades tintoriais.
Aínda que a heterocromatina non transcribe xenes, exerce algunhas importantes funcións, como son: (1). Organiza os dominios nucleares. A cromatina está situada no núcleo de forma preferente nunha posición máis periférica, preto da membraana nuclear, e deixa a zona central do núcleo á eucromatina, facilitando a tanscrición. (2). Xoga un papel na función do centrómero, xa que parece que é necesaria para a cohesión das cromátides irmás e a correcta disxunción dos cromosomas meióticos. (3). Realiza un control transcricional, por medio da formación de complexos locais de transcrición ou pola súa influencia en zonas extensas adxacentes. Os xenes da eucromatina poden ser silenciados cando están situados preto dun dominio de heterocromatina.
As propiedades da cromatina poden resumirse así: (1). A heterocromatina está condensada. Isto faina moi cromofílica e bastante inaccesible a ADNases e encimas de restrición. (2). O ADN da heterocromatina replícase máis tarde ca a eucromatina. (3). O ADN da heterocromatina está metilado (con metil-citosinas), especialmente na heterocromatina constitutiva. (4). Na heterocromatina as histonas están hipoacetiladas, mentres que na eucromatina están hiperacetiladas. (5). As histonas da heterocromatina están metiladas na lisina 9. (6). A heterocromatina é transcricionalmente inactiva, seguramente pola súa escasz en xenes. (7). A heterocromatina non participa na recombinación xenética.
Aínda que toda a heterocromatina comparte as anteriores propiedades, a heterocromatina pode ser de dous tipos diferentes: constitutiva ou facultativa.
- A heterocromatina constitutiva, idéntica para todas as células do organismo e que carece de información xenética, inclúe os telómeros e centrómeros do cromosoma que non expresan o seu ADN. A heterocromatina constitutiva contén un tipo particular de ADN denominado ADN satélite, formado por gran número de secuencias curtas repetidas en tándem. Os tipos principais deste ADN son o ADN satélite α, e os ADN satélite I, II e III. Estas secuencias de ADN satélite poden dobrarse sobre si mesmas e poden ter un papel importante na formación da estrutura altamente compacta da heterocromatina constitutiva. A heterocromatina constitutiva é estable e conserva as súas propiedades heterocromáticas durante todas as etapas do desenvolvemento e en todos os tecidos. A heterocromatina constitutiva é altamente polimórfica, probablemente debido á inestabilidade do ADN satélite. Este polimorfismo pode afectar, non soamente ao seu tamaño senón tamén á localización da heterocromatina, e aparentemente non ten un efecto fenotípico. A heterocromatina constitutiva aparece intensamente tinguida na técnica de bandas C, o que é o resultado dunha renaturalización moi rápida do ADN satélite tras a desnaturalización.
- A heteromatina facultativa, diferente nos distintos tipos celulares, contén todos aqueles xenes que non se expresan ou que poden expresarse só nalgún momento. Inclúe o ADN satélite e o corpo ou corpúsculo de Barr. A heterocromatina facultativa caracterízase pola presenza de secuencias de nucleótidos repetidas tipo LINE. Estas secuencias, espalladas ao longo do xenoma, poderían promover a propagación dunha estrutura de cromatina condensada. A heterocromatina facultativa é reversible, e o seu estado heterocromático depende da etapa do desenvolvemento e do tipo celular. Dous exemplos deste tipo de heterocromatina son o cromosoma X inactivo (corpo de Barr) das células somáticas femininas e a vesícula sexual inactiva na etapa do paquiteno das meioses masculinas. A heterocromatina facultativa non é particularmente rica en ADN satélite, e por esa razón non é polimórfica. A heterocromatina facultativa nunca queda tinguida na técnica de bandas C.
Tense observado que na formación de heterocromatina frecuentemente participa o fenómeno do ARN interferente. Por exemplo, en Schizosaccharomyces pombe, a heterocromatina fórmase no centrómero, telómeros e no loci mating-type.[3] A formación da heterocromatina no centrómero depende do mecanismo da interferencia de ARN (RNAi). Prodúcense ARN de dobre cadea complementarios de secuencias repetidas localizadas no centrómero, que inducen ARNi e seguidamente a metilación da lisina 9 da histona 3 e enlazamento de Swi6 (proteína estrutural da heterocromatina, a cal é homóloga a HP1 en mamíferos).[4]
Outras organizacións da cromatina
editarDurante a espermatoxénese animal, a cromatina das espermátidas remodélase orixinando unha estrutura máis ampla e case cristalina. Este proceso está asociado co cesamento da transcrición e implica cambios nas proteínas nucleares. As histonas son en grande medida desprazadas e substituídas por protaminas (pequenas proteínas ricas en arxinina).
Papel da cromatina na expresión xénica
editarA estrutura local da cromatina durante a interfase depende dos xenes presentes na zona do ADN implicada, o ADN que codifica xenes que se transcriben moi activamente está máis laxamente empaquetado e asociado con ARN polimerases (denomínase eucromatina), mentres que o ADN que codifica xenes inactivos está asociado con proteínas estruturais e está máis densamente empaquetado (denomínase heterocromatina).[5][6] Por tanto, a cromatina é unha estrutura dinámica que adapta o seu estado de compactación e empaquetamento para optimizar os procesos de replicación, transcrición e reparación do ADN, e ten un papel regulatorio fundamental na expresión xénica. Os distintos estados de compactación poden asociarse (aínda que non exclusivamente) ao grao de transcrición que mostren os xenes que se encontran nesas zonas. A cromatina é, en principio, fortemente represiva para a transcrición, xa que a asociación do ADN coas distintas proteínas dificulta a acción das distintas ARN polimerases. Por tanto, existe unha variada cantidade de procesos remodeladores da cromatina e modificadores de histonas.
Na regulación da expresión xénica funciona o que se coñece como "código de histonas". As distintas histonas poden sufrir modificacións postraducionais, como poden ser a metilación, acetilación, fosforilación, xeralmente en residuos lisina ou arxinina.[7] A acetilación está asociada coa activación da trascrición, xa que ao acetilarse unha lisina, diminúe a carga positiva global da histona polo cal ten unha menor afinidade polo ADN (que está cargado negativamente). En consecuencia, o ADN pode estar unido menos fortemente, o que permite o acceso da maquinaria transcricional. Polo contrario, a metilación está asociada coa represión transcricional e unha unión ADN-histona máis forte (pero isto non sempre se cumpre). Por exemplo, no lévedo Schizosaccharomyces pombe, a metilación no residuo de lisina 9 da histona 3 está asociada coa represión da transcrición na heterocromatina, mentres que a metilación no residuo de lisina 4 promove a expresión de xenes.[4]
Os encimas que levan a cabo as funcións de modificación de histonas son as acetilases e desacetilases de histonas, e as metilases e desmetilases de histonas, que forman distintas familias de encimas cuxos integrantes se encargan de modificar un residuo en particular da longa cadea das histonas.
Ademais das modificacións das histonas, existen tamén maquinarias remodeladoras da cromatina, como por exemplo SAGA, que se encargan de reposicionar nucleosomas, xa sexa movéndoos, rotándoos, ou mesmo desensamblándoos parcialmente, retirando algunhas das histonas constituíntes do nucleosoma, e logo volvéndoos a colocar. En xeral, as maquinarias remodeladoras da cromatina son esenciais para o proceso de transcrición en eucariotas, xa que permiten o acceso e a acción das polimerases.
Outra forma de marcación da cromatina como "inactiva" pode darse a nivel da metilación do ADN, en citosinas que pertenzan a dinucleótidos CpG. En xeral a metilación do ADN e da cromatina son procesos sinérxicos, xa que, por exemplo, ao metilarse o ADN, existen encimas metiladores de histonas que poden recoñecer citosinas metiladas, e metilan histonas próximas. Do mesmo modo, encimas que metilan o ADN poden recoñecer histonas metiladas, e así seguir coa metilación a nivel de ADN. O grupo polycomb de proteínas xoga tamén un papel na regulación dos xenes por medio da modulación da estrutura da cromatina.[8]
Todas estas modificacións forman parte do que se chama modificacións epixenéticas.
Notas
editar- ↑ Robinson DJ, Fairall L, Huynh VA, Rhodes D. (2006). "EM measurements define the dimensions of the "30-mm" chromatin fiber: evidence for a compact, interdigitated structure". PNAS 103 (17): 6506–11. PMC 1436021. PMID 16617109. doi:10.1073/pnas.0601212103.
- ↑ Wong H, Victor JM, Mozziconacci J. (2007). "An all-atom model of the chromatin fiber containing linker histones reveals a versatile structure tuned by the nucleosomal repeat leαngth". PLoS ONE 2 (9): e877. PMC 1963316. PMID 17849006. doi:10.1371/journal.pone.0000877.
- ↑ Lippman Z. y Martienssen R. (2004). Nature 431: 364-370
- ↑ 4,0 4,1 Volpe, T. y colaboradores. Science 297: 1833-1837
- ↑ "Chromatin Network Home Page.". Consultado o 2008-11-18.
- ↑ Dame, R.T. (2005). "The role of nucleoid-associated proteins in the organization and compaction of bacterial chromatin". Molecular Microbiology 56 (4): 858–70. PMID 15853876. doi:10.1111/j.1365-2958.2005.04598.x.
- ↑ Bernstein, B.E., T.S. Mikkelsen, X. Xie, M. Kamal, D.J. Huebert, J. Cuff, B. Fry, A. Meissner, M. Wernig, K. Plath, R. Jaenisch, A. Wagschal, R. Feil, S.L. Schreiber & E.S. Lander (2006). "A bivalent chromatin structure marks key developmental genes in embryonic stem cells". Cell 125 (2): 315–26. ISSN 0092-8674. PMID 16630819. doi:10.1016/j.cell.2006.02.041.
- ↑ Portoso M and Cavalli G (2008). "The Role of RNAi and Noncoding RNAs in Polycomb Mediated Control of Gene Expression and Genomic Programming". RNA and the Regulation of Gene Expression: A Hidden Layer of Complexity. Caister Academic Press. ISBN 978-1-904455-25-7.
Véxase tamén
editarBibliografía
editar- Alberts, Bruce; et al. (1996). Biología Molecular de la célula. Barcelona: Ediciones Omega. ISBN 84-282-1011-X.
- Cooper, Geoffrey M. 2000. The Cell, 2nd edition, A Molecular Approach. Chapter 4.2 Chromosomes and Chromatin.
- Corces, V. G. 1995. Chromatin insulators. Keeping enhancers under control. Nature 376:462-463.
- Cremer, T. 1985. Von der Zellenlehre zur Chromosomentheorie: Naturwissenschaftliche Erkenntnis und Theorienwechsel in der frühen Zell- und Vererbungsforschung, Veröffentlichungen aus der Forschungsstelle für Theoretische Pathologie der Heidelberger Akademie der Wissenschaften. Springer-Vlg., Berlín, Heidelberg.
- Elgin, S. C. R. (ed.). 1995. Chromatin Structure and Gene Expression, vol. 9. IRL Press, Oxford, Nova York, Tokyo.
- Gerasimova, T. I., e V. G. Corces. 1996. Boundary and insulator elements in chromosomes. Current Op. Genet. and Dev. 6:185-192.
- Gerasimova, T. I., e V. G. Corces. 1998. Polycomb and Trithorax group proteins mediate the function of a chromatin insulator. Cell 92:511-521.
- Gerasimova, T. I., e V. G. Corces. 2001. CHROMATIN INSULATORS AND BOUNDARIES: Effects on Transcription and Nuclear Organization. Annu Rev Genet 35:193-208.
- Gerasimova, T. I., K. Byrd, e V. G. Corces. 2000. A chromatin insulator determines the nuclear localization of DNA [In Process Citation]. Mol Cell 6:1025-35.
- Ha, S. C., K. Lowenhaupt, A. Rich, Y. G. Kim, e K. K. Kim. 2005. Crystal structure of a junction between B-DNA and Z-DNA reveals two extruded bases. Nature 437:1183-6.
- Pollard, T., e W. Earnshaw. 2002. Cell Biology. Saunders.
- Saumweber, H. 1987. Arrangement of Chromosomes in Interphase Cell Nuclei, p. 223-234. In W. Hennig (ed.), Structure and Function of Eucaryotic Chromosomes, vol. 14. Springer-Verlag, Berlín, Heidelberg.
- Sinden, R. R. 2005. Molecular biology: DNA twists and flips. Nature 437:1097-8.
- Van Holde KE. 1989. Chromatin. Nova York: Springer-Verlag. ISBN 0-387-96694-3.
- Van Holde, K., J. Zlatanova, G. Arents, e E. Moudrianakis. 1995. Elements of chromatin structure: histones, nucleosomes, and fibres, p. 1-26. In S. C. R. Elgin (ed.), Chromatin structure and gene expression. IRL Press at Oxford University Press, Oxford.
Outros artigos
editar- Cromatina sexual ou corpúsculo de Barr
- Eucromatina
- Heterocromatina
- Nucleosoma
- Histona
Ligazóns externas
editar- Recent chromatin publications and news
- Chromatin, Histones & Cathepsin; PMAP O mapa da proteólise - animación.
- Protocol for in vitro Chromatin Assembly