Succinato deshidroxenase
A succinato deshidroxenase ou succinato-coencima Q redutase (SQR) ou Complexo II respiratorio é un complexo encimático, unido á membrana mitocondrial interna das mitocondrias de mamíferos e moitas células bacterianas. É o único encima que participa tanto no ciclo de ácido cítrico coma na cadea de transporte electrónico.[1]
| succinato deshidroxenase (ubiquinona) | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| |||||||||
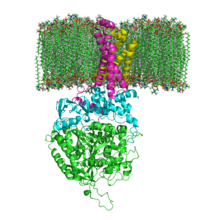
| Estrutura da SQR na membrana fosfolipídica. SdhA, SdhB, SdhC e SdhD | |||||||||
| Identificadores | |||||||||
| Número EC | 1.3.5.1 | ||||||||
| Número CAS | 9028-11-9 | ||||||||
| Bases de datos | |||||||||
| IntEnz | vista de IntEnz | ||||||||
| BRENDA | entrada de BRENDA | ||||||||
| ExPASy | vista de NiceZyme | ||||||||
| KEGG | entrada de KEGG | ||||||||
| MetaCyc | vía metabólica | ||||||||
| PRIAM | perfil | ||||||||
| Estruturas PDB | RCSB PDB PDBe PDBj PDBsum | ||||||||
| Gene Ontology | AmiGO / EGO | ||||||||
| |||||||||
| Succinato deshidroxenase | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| Identificadores | |||||||||
| Símbolo | Fumarate_red_C | ||||||||
| Pfam | PF02300 | ||||||||
| InterPro | IPR003510 | ||||||||
| OPM superfamily | 3 | ||||||||
| OPM protein | 1nek | ||||||||
| |||||||||
No paso 8 do ciclo do ácido cítrico, a SQR cataliza a oxidación do succinato a fumarato á vez que se reduce a ubiquinona a ubiquinol. Isto ten lugar na membrana mitocondrial interna ao acoplárense as dúas reaccións.
Estrutura
editarSubunidades
editarOs monómeros da SQR de mamíferos, mitocondriais, e moitas bacterias están compostos por catro subunidades: dúas hidrofílicas e dúas hidrofóbicas. As dúas primeiras subunidades, que son unha flavoproteína (SdhA) e unha proteína ferro-sulfurada (SdhB), son hidrofílicas. A SdhA contén unha molécula do cofactor flavín adenín dinucleótido (FAD) unida covalentemente (grupo prostético) e o sitio de unión para o succinato. Ademais, a SdhB contén tres clusters de ferro-xofre: [2Fe-2S], [4Fe-4S], e [3Fe-4S]. As outras dúas subunidades son subunidades áncora de membrana hidrofóbicas denominadas SdhC e SdhD. As mitocondrias humanas conteñen dúas isoformas distintas de SdhA (subunidades Fp tipo I e tipo II), estas isoformas tamén se encontran nos vermes Ascaris suum e Caenorhabditis elegans.[2] As subunidades forman un complexo citocromo b unido á membrana con seis hélices transmembrana que conteñen un grupo hemo B e un sitio de unión para a ubiquinona. Nas subunidades SdhC e SdhD tamén se encontran dúas moléculas de fosfolípidos, unha de cardiolipina e unha de fosfatidiletanolamina, as cales serven para ocupar o espazo hidrofóbico situado baixo o hemo B. Arredor da SdhC e SdhD está a membrana fosfolipídica co espazo intermembrana na parte superior da primeira e segunda imaxes do artigo.[3]
Sitio de unión da ubiquinona
editarO sitio de unión da ubiquinona está localizado nun oco formado pola SdhB, SdhC, e SdhD. A ubiquinona está estabilizada polas cadeas laterais da His207 da subunidade B, a Ser27 e a Arg31 da subunidade C, e a Tyr83 da subunidade D. O anel quinona está rodeado pola Ile28 da subunidade C e a Pro160 da subunidade B. Estes residuos, xunto coa Ile209, o Trp163, e o Trp164 da subunidade B, e a Ser27 da subunidade C, forman o ambiente hidrofóbico do peto de unión da quinona.[4]
Sitio de unión do succinato
editarA SdhA proporciona o sitio de unión para a oxidación do succinato. As cadeas laterais de Thr254, His354, e Arg399 da subunidade A estabiliza a molécula mentres que o FAD oxida e transporta os electróns ao primeiro cluster de ferro-xofre, o [2Fe-2S].[5]
Centros redox
editarO centro de unión do succinato e o centro de unión da ubiquinona están conectados por unha cadea de centros redox que inclúen o FAD e os clusters de ferro-xofre. Esta cadea esténdese no monómero ao longo dunha lonxitde duns 40 Å. Todas as distancias de bordo a bordo entre os centros son menores que o límite de 14 Å suxerido para a transferencia electrónica fisiolóxica.[3]
Mecanismo
editarOxidación do succinato
editarSábese pouco sobre o mecanismo exacto de oxidación do succinato. Porén, a estrutura cristalina obtida do complexo mostra que o FAD, Glu255, Arg286, e His242 da subunidade A son bos candidatos para a reacción inicial de desprotonación. Hai dous posibles mecanismos de eliminación, chamados E2 ou E1cb. Na eliminación E2, o mecanismo é coordinado. O residuo básico ou cofactor desprotona o carbono alfa, e o FAD acepta o hidruro procedente do carbono beta, oxidando o succinato unido, que se converte en fumarato. No mecanismo E1cb, fórmase un intermediario enolato, antes de que o FAD acepte o hidruro. Requírense máis investigacións para determinar que mecanismo de eliminación afecta ao succinato na succinato deshidroxenase. O fumarato oxidado, que agora está unido pouco firmemente ao centro activo, é libre de saír da proteína e pasar á matriz da mitocondria.
Tunelado de electróns
editarDespois de que os electróns son derivados da oxidación do succinato por medio do FAD, son tunelados ao longo do centros de ferro-xofre ata que chegan ao cluster [3Fe-4S] final. Estes electróns son transferidos seguidamente a unha molécula de ubiquinona no centro activo. O sistema de tunelado de electróns de ferro-xofre móstrase na última imaxe.
Redución da ubiquinona
editarO oxíxeno do carbonilo O1 da ubiquinona é orientado no sitio activo por medio de interaccións por ponte de hidróxeno coa Tyr83 da subunidade D. A presenza de electróns no cluster de ferro-xofre [3Fe-4S] induce o movemento da ubiquinona a unha segunda orientación. Isto facilita unha segunda interacción por ponte de hidróxeno entre o grupo carbonilo O4 da ubiquinona e a Ser27 da subunidade C. Despois do primeiro paso de redución dun só electrón, fórmase un radical semiquinona. O segundo electrón chega desde o cluster [3Fe-4S] e produce a redución completa da ubiquinona a ubiquinol. Este mecanismo da redución da ubiquinona móstrase na imaxe correspondente.
Grupo prostético hemo
editarAínda que a funcionalidade do hemo na succinato deshidroxenase aínda hai que investigala máis, algúns estudos concluíron que o primeiro electrón que pasa á ubiquinona desde o [3Fe-4S] pode ser tunelado cara a adiante e atrás entre o hemo e o intermediato ubiquinona. Deste modo, o cofactor hemo actúa como un sumidoiro de electróns. O seu papel é impedir a interacción do intermediato co oxíxeno molecular, o que produciría especies reactivas do oxíxeno.
Propúxose que existe un mecanismo de peche que pode servir para impedir que os electróns tunelen directamente ao hemo desde o cluster [3Fe-4S]. Un candidato potencial é o residuo His207, que se encontra entre o cluster e o hemo. A His207 da subunidade B está en directa proximidade ao cluster [3Fe-4S], á ubiquinona unida, e ao hemo; e podería modular o fluxo de electróns entre estes centros redox.[6]
Transferencia de protóns
editarPara reducir completamente a quinona na SQR cómpren dous electróns e dous protóns. Propúxose que unha molécula de auga (HOH39) chega ao sitio activo e é coordinada pola His207 da subunidade B, a Arg31 da subunidade C, e o Asp82 da subunidade D. A semiquinona é protonada por protóns procedentes da HOH39, completando a redución da ubiquinona a ubiquinol. A His207 e o Asp82 probablemente facilitan este proceso. Outros estudos consideran que a Tyr83 da subunidade D está coordinada a unha histidina próxima e ao oxíxeno carbonilo O1 da ubiquinona. O residuo de histidina fai diminuír o pKa da tirosina, facendo que lle sexa máis fácil doar o seu protón ao intermediato ubiquinona reducido.
Inhibidores
editarHai dúas clases de inhibidores do Complexo II: os que se unen no peto do sucinato e os que se unen no peto da ubiquinona. Entre os inhibidores de tipo ubiquinona están a carboxina e a thenoiltrifluoroacetona. Os inhibidores análogos do succinato son, por exempo, o composto sintético malonato e os intermediatos do ciclo do ácido cítrico malato e oxalacetato. O oxalacetato é un dos inhibidores máis potentes do Complexo II. Non se comprende totalmente como un intermediato común do ciclo do ácido cítrico pode inhibir ao Complexo II, pero podería exercer un papel protector para minimizar a produción de superóxidos mediada por transferencia inversa de electróns polo Complexo I.[7]
Os inhibidores de tipo ubiquinona téñense utilizado como funxicidas na agricultura desde a década de 1960s. A carboxina utilizouse principalmente para controlar as enfermidades producidas nas plantas por fungos basidiomicetos. Máis recentemente, desenvolvéronse outros compostos cun espectro de acción máis amplo contra un conxunto de patóxenos de plantas, como boscalid, penthiopyrad e fluopyram.[8] Algúns fungos que son importantes como pragas agrícolas non son sensibles aos inhibidores de tipo ubiquinona de nova xeración.[9]
Enfermidades
editarO papel fundamental da succinato-coencima Q redutase na cadea de transporte de electróns da mitocondria fai que sexa vital para a maioría dos organismos pluricelulres, e a eliminación deste encima do xenoma viuse que é letal para os ratos na fase embrionaria.
- As mutacións na SdhA poden orixinar a síndrome de Leigh, encefalopatía mitocondrial, e atrofia óptica.
- As mutacións SdhB poden causar tumoroxénese nas células cromafíns, orixinando paraganglioma hereditario e feocromocitoma hereditario. Os tumores adoitan a ser malignos. Poden tamén diminuír a éxpectativa de vida e incrementar a produción de ións superóxido.
- As mutacións na SdhC poden producir unha diminución na duración da vida, incrementar a produción de ións superóxido, e causar paraganglioma e feocromocitoma hereditarios. Neste caso, os tumores adoitan a ser benignos. Estas mutacións son pouco comúns.
- As mutacións na SdhD poden causar tamén paraganglioma e feocromocitoma hereditarios. Os tumores tenden a ser benignos, e aparecen xeralmente na cabeza e pescozo. Estas mutacións poden tamén facer decrecer a duración da vida e incrementar a produción de ións superóxido.
A succinato deshidroxenase de mamíferos funciona non só na xeración de enerxía mitocondrial, senón que tamén ten un papel na detección do oxíxeno e a supresión de tumores; e, xa que logo, é o obxecto de diversas investigacións que están en marcha.
Notas
editar- ↑ Oyedotun KS, Lemire BD (2004). "The quaternary structure of the Saccharomyces cerevisiae succinate dehydrogenase. Homology modeling, cofactor docking, and molecular dynamics simulation studies". J. Biol. Chem. 279 (10): 9424–31. PMID 14672929. doi:10.1074/jbc.M311876200.
- ↑ Tomitsuka E, Hirawake H, Goto Y, Taiwaki M, Harada S, Kita K (2003). "Direct evidence for two distinct forms of the flavoprotein subunit of human mitochondrial complex II (succinate-ubiquinone reductase)". J. Biochem 134 (2): 191–5. PMID 12966066. doi:10.1093/jb/mvg144.
- ↑ 3,0 3,1 Yankovskaya V, Horsefield R, Törnroth S; et al. (2003). "Architecture of succinate dehydrogenase and reactive oxygen species generation". Science 299 (5607): 700–4. PMID 12560550. doi:10.1126/science.1079605.
- ↑ Horsefield R, Yankovskaya V, Sexton G; et al. (2006). "Structural and computational analysis of the quinone-binding site of complex II (succinate-ubiquinone oxidoreductase): a mechanism of electron transfer and proton conduction during ubiquinone reduction". J. Biol. Chem. 281 (11): 7309–16. PMID 16407191. doi:10.1074/jbc.M508173200.
- ↑ Kenney WC (1975). "The reaction of N-ethylmaleimide at the active site of succinate dehydrogenase". J. Biol. Chem. 250 (8): 3089–94. PMID 235539.
- ↑ Tran QM, Rothery RA, Maklashina E, Cecchini G, Weiner JH (2006). "The quinone binding site in Escherichia coli succinate dehydrogenase is required for electron transfer to the heme b". J. Biol. Chem. 281 (43): 32310–7. PMID 16950775. doi:10.1074/jbc.M607476200.
- ↑ Muller FL, Liu Y, Abdul-Ghani MA; et al. (2008). "High rates of superoxide production in skeletal-muscle mitochondria respiring on both complex I- and complex II-linked substrates". Biochem. J. 409 (2): 491–9. PMID 17916065. doi:10.1042/BJ20071162.
- ↑ Avenot, H. F.; Michailides, T. J. (2010). "Progress in understanding molecular mechanisms and evolution of resistance to succinate dehydrogenase inhibiting (SDHI) fungicides in phytopathogenic fungi". Crop Protection 29 (7): 643. doi:10.1016/j.cropro.2010.02.019.
- ↑ Dubos, T.; Pasquali, M.; Pogoda, F.; Casanova, A. L.; Hoffmann, L.; Beyer, M. (2013). "Differences between the succinate dehydrogenase sequences of isopyrazam sensitive Zymoseptoria tritici and insensitive Fusarium graminearum strains". Pesticide Biochemistry and Physiology 105: 28. [ http://www.sciencedirect.com/science/article/pii/S0048357512001769 doi 10.1016/j.pestbp.2012.11.004].