Poliovirus
| Poliovirus | |||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
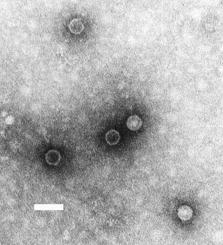 Micrografía de microscopio electrónico de transmisión de virións de poliovirus. Barra de escala, 50 nm. | |||||||||||||||||
| Clasificación científica | |||||||||||||||||
| |||||||||||||||||
| Serotipos | |||||||||||||||||
| |||||||||||||||||
O poliovirus é o axente vírico causante da poliomielite (ou polio). Actualmente non se considera unha especie de virus senón un serotipo da especie Enterovirus C, da familia Picornaviridae.[1]
O poliovirus está composto dun xenoma de ARN e unha cápside proteica e sen envoltura. O xenoma é unha molécula de ARN monocatenario de sentido positivo duns 7 500 nucleótidos de longo.[2] A partícula viral é duns 30 nm de diámetro con simetría icosaédrica. Debido ao seu curto xenoma e a súa composición simple (só un ARN e as proteínas da cápside), o poliovirus é considerado como o virus importante máis simple.[3]
O poliovirus illouse por primeira vez en 1909 por Karl Landsteiner e Erwin Popper.[4] A estrutura do virus foi dilucidada primeiro usando difracción de raios X por un equipo do Birkbeck College liderado por Rosalind Franklin,[5][6] mostrando que o virus da polio tiña unha simetría icosaédrica.[7]
En 1981, publicouse o xenoma do poliovirus grazas aos traballos de dous equipos: o de Vincent Racaniello e David Baltimore no MIT[8] e o de Naomi Kitamura e Eckard Wimmer na Universidade de Stony Brook.[9] O poliovirus é un dos virus mellor caracterizados, e converteuse nun sistema modelo útil para a comprensión da bioloxía dos virus de ARN.
Ciclo de replicación editar
O poliovirus infecta as células humanas ao unirse a un receptor de tipo inmunoglobulina, o CD155 (tamén coñecido como receptor do poliovirus ou PVR)[11][12] situado na superficie celular.[13] A interacción do poliovirus co CD155 facilita un cambio conformacional irreversible da partícula viral necesario para a entrada viral.[14][15] Despois de adherirse á membrana plasmática da célula hóspede, crese que a entrada do ácido nucleico viral se produce por unha destas dúas vías: por medio da formación dun poro na membrana plasmática a través do cal se “inxecta” o ARN no citoplasma, ou por captación do virus por endocitose mediada por receptor.[16] Hai probas experimentais recentes que apoian esta última hipótese e suxiren que o poliovirus se une ao CD155 e despois entra por endocitose. Inmediatamente despois da internalización da partícula, libérase o ARN viral.[17]
O poliovirus é un virus de ARN de febra positiva. O seu xenoma está encerrado na partícula viral e pode utilizarse como ARN mensaxeiro e ser inmediatamente traducido pola célula hóspede. Ao entrar, o virus aprópiase da maquinaria de tradución da célula, causando a inhibición da síntese proteica celular en favor da produción de proteínas específicas do virus.[18] A diferenza dos ARNm da célula hóspede, o extremo 5' do ARN do poliovirus é extremadamente longo (duns 700 nucleótidos) e está moi estruturado. Esta rexión do xenoma viral denomínase sitio de entrada ao ribosoma interno (IRES, do inglés internal ribosome entry site). Esta rexión consta de moitas estruturas secundarias e 3 ou 4 dominios. O dominio máis importante do IRES é o dominio 3 (parte de iniciación da tradución). O dominio 3 é un elemento do ARN de autopregamento que contén motivos estruturais conservados en varios talos-bucle estables enlazados por dúas unións de catro vías. Un IRES consta de moitos dominios e estes dominios tamén constan de moitos bucles que contribúen a unha tradución modificada sen a caparuza do extremo 5’ ao apropiarse do ribosoma celular que, a diferenza da tradución convencional, non empeza desde o primeiro paso senón desde os últimos pasos. O bucle de interacción do dominio 3 é un tetrabucle GNRA. Os residuos de adenosinas A180 e A181 no tetrabucle GUAA forman enlaces de hidróxeno por interaccións de apareamento de bases non canónico cos pares de bases dos receptores C230/G242 e G231/C241, respectivamente.[19] As mutacións xenéticas nesta rexión impiden a produción de proteínas virais.[20][21][22] O primeiro IRES que se descubriu atopouse no ARN do poliovirus.[23]
O ARNm do poliovirus tradúcese formando un único polipéptido longo. Este polipéptido é despois autoclivado por proteases internas orixinando 10 proteínas virais separadas. Non todas as clivaxes ou cortes ocorren coa mesma eficiencia, polo que a cantidade de proteínas producidas pola clivaxe dos polipéptidos varía: por exemplo, prodúcense cantidades menores da proteína 3Dpol que das proteínas da cápside, VP1–4.[24][25] Estas proteínas virais separadas son:[3][26]
- 3Dpol, unha ARN polimerase dependennte de ARN cuxa función é facer múltiples copias do xenoma do ARN viral.
- 2Apro e 3Cpro/3CDpro, proteases que clivan o polipéptido viral.
- VPg (3B), unha pequena proteína que se une ao ARN viral e é necesaria para a síntese do ARN de febra positiva e negativa.
- 2BC, 2B, 2C (unha ATPase).[27], 3AB, 3A, 3B, proteínas que forman o complexo proteico necesario para a replicación do virus.
- VP0, que é despois clivada orixinando VP2 e VP4, VP1 e VP3, que son as proteínas da cápside viral.
Despois da tradución, realízanse a transcrición e replicación do xenoma, que implican un só proceso (síntesde do ARN (+)). Para que se replique o ARN (+), deben transcribirse múltiples copias de ARN (−) que despois se utilizan como moldes para a síntese de ARN (+). Os intermediarios replicativos (RIs), que están asociados con moléculas de ARN constan dun molde de ARN e varios ARN en crecemento de distintas lonxitudes, que se ven tanto nos complexos de replicación de ARN (−) coma de ARN (+). Para a síntese de cada ARN de febra positiva e negativa, as proteínas VPg do poliovirus funcionan como un cebador. A ARN polimerase dependente de ARN do poliovirus engade dous nucleótidos de uracilo (UU) á proteína VPg que utiliza a cola poli(A) no extremo 3′ do xenoma de ARN monocatenario positivo como padrón, para a síntese de ARN antixenómico de febra negativa. Para iniciar esta síntese de ARN monocatenario negativo, é necesario o hidroxilo da tirosina da VPg. Pero para a iniciación da síntese do ARN de febra positiva, cómpre a uridilización da VPg dependente de CRE (polo que é unha proteína con U unidos). Iso significa que se utiliza unha vez máis a VPg como cebador, pero esta vez engádense os dous uridín trifosfato usando como molde un elemento de replicación que actúa en cis (ou CRE, do inglés cis-acting replication element).[28][29]. O CRE do poliovirus identifícase no ARN como un talo no que non se produciu apareamento de bases e un bucle final de 61 nucleótidos. O CRE encóntrase en enterovirus. É un elemento estrutural secundario do ARN altamente conservado incrustado na rexión do xenoma codificante da poliproteína. O complexo pode ser translocado á rexión 5' do xenoma que non ten actividade codificante, polo menos a unha distancia de 3,7 kb da localización inicial. Este proceso pode ocorrer sen influír negativamente na actividade. As copias do CRE non inflúen na replicación negativamente. O proceso de uridilización da VPg que ten lugar no CRE necesita a presenza de 3CDpro, que é unha proteína que se une ao ARN. Esta únese ao CRE de frorma directa e específica. Debido á súa presenza, a VPg pode unirse correctamente ao CRE e realízase a produción primaria sen problemas.[30]
Algunhas das moléculas de ARN (+) utilízanse como moldes para facer máis síntese de ARN (−), algúns funcionan como ARNm, e outros están destinados a ser os xenomas dos virións da proxenie.[24]
Na ensamblaxe das novas partículas do virus (é dicir, o empaquetamento do xenoma da proxenie nunha procápside que poida sobrevivir fóra da célula hóspede) ocorre o seguinte:[31]
- Ensámblanse cinco copias de cada de VP0, VP3 e VP1, cuxa parte N-terminal e a VP4 forman a superfice interna da cápside, ensámblanse formando un ‘pentámero’ e 12 destes pentámeros forman unha procápside. (A superficie externa da cápside consta de VP1, VP2, VP3; mentres que a parte C-terminal de VP1 e VP3 forman os canóns que hai arredor de cada vértice; nese momento, clívanse as 60 copias de VP0 orixinando VP4 e VP2.)
- Cada procápside adquire unha copia do xenoma do virus, coa VPg aínda unida ao extremo 5'.
O poliovirus completamente ensamblado abandona a célula hóspede cando ten lugar a súa lise,[32] que se produce de 4 a 6 horas despois da iniciación da infección en células de mamífero cultivadas.[33] O mecanismo da liberación viral da célula non está claro,[2] pero cada célula que morre pode liberar ata 10 000 virións do poliovirus.[33]
Drake demostrou que o poliovirus pode sufrir unha reactivación de multiplicidade.[34] Isto significa que cando os poliovirus son irradiados con luz UV e se lles permite que realicen múltiples infeccións de células hóspede, pode formarse proxenie viable incluso cando as doses de UV inactivarían o virus nunha soa infección. O poliovirus pode sufrir recombinación xenética cando están presentes polo menos dous xenomas virais na mesma célula hóspede. Kirkegaard e Baltimore[35] presentaron probas de que a ARN polimerase dependente de ARN (RdRP) cataliza a recombinación por un mecanismo de elección de copia no cal a RdRP cambia entre moldes de ARN monocatenario (+) durante a síntese de febras negativas. A recombinación en virus de ARN parece ser un mecanismo adaptativo para reparar danos no xenoma.[36][37]
Orixe e serotipos editar
O poliovirus é estruturalmente similar a outros enterovirus humanos (coxsackievirus, ecovirus e rinovirus), que tamén usan moléculas de tipo inmunoglobulina para recoñecer e entrar nas células hóspede.[12] A análise filoxenética das secuencias de ARN e proteínas do poliovirus indica que probablemente evolucionou a partir dun antepasado que era un virus Coxsackie A do grupo C, que se orixinou por unha mutación na cápside.[38] A especiación do poliovirus seguramente ocorreu como resultado dun cambio na especificidade polo receptor celular desde a molécula de adhesión intercelular 1 (ICAM-1), utilizada polos virus Coxsackie A do grupo C, ao CD155; orixinando un cambio na patoxenicidade, e permitindo que o virus infecte o tecido nervioso.
A taxa de mutación do virus é relativamente alta incluso para un virus de ARN, cunha taxa de substitución sinónima de 1,0 x 10−2 substitucións/sitio/ano e unha taxa de substitución non sinónima de 3,0 x 10−4 substitucións/sitio/ano.[39] A distribución de bases no xenoma non é aleatoria, e a adenosina é menos común do esperado no extremo 5' e maior do esperado no extremo 3'.[40] O uso dos codóns non é aleatorio, e os codóns que acaban en adenosina están favorecidos e os que acaban en citosina ou guanina tenden a ser evitados. O uso de codóns difire entre os tres xenotipos e parece que é impulsado pola mutación e non pola selección.[41]
Os tres serotipos de poliovirus, PV-1, PV-2 e PV-3, teñen proteínas da cápside lixeiramente diferentes. As proteínas da cápside definen a especificidade de receptor celular e a antixenicidade do virus. A PV-1 é a forma máis común que se encontra na natureza, pero as tres formas son extremadamente infecciosas.[4] En marzo de 2020, a PV-1 salvaxe ou silvestre estaba moi localizada en rexións de Paquistán e Afganistán. A PV-2 salvaxe foi declarada erradicada en setembro de 2015 despois de ser detectada por última vez en 1999,[42] mentres que a PV-3 salvaxe foi declarada erradicada en 2019 despois de detectarse por última vez en 2012.[43]
As cepas específicas de cada serotipo utilízanse para preparar as vacinas contra a polio. A vacina da polio inactiva prepárase por inactivación con formaldehido das tres cepas salvaxes virulentas de referencia, que son: Mahoney ou Brunenders (PV-1), MEF-1/Lansing (PV-2) e Saukett/Leon (PV-3). A vacina da polio oral contén cepas vivas atenuadas (debilitadas) dos tres serotipos de poliovirus. O paso das cepas do virus por células epiteliais de ril de mono introduce mutacións no IRES viral e dificultra (ou atenúa) a capacidade do virus de infectar o tecido nervioso.[33]
Taxonomía editar
Os poliovirus eran antes clasificados como unha especie distinta pertencente ao xénero Enterovirus da familia Picornaviridae. En 2008, a antiga especie Poliovirus foi eliminada e os tres serotipos foron asignados á especie Enterovirus C humano (que posteriormente foi renomeada como Enterovirus C), do xénero Enterovirus da familia Picornaviridae. A especie tipo do xénero Enterovirus foi cambiada de Poliovirus a Enterovirus C (humano).[44]
Patoxénese editar
- Artigo principal: Poliomielite.
O principal determinante da infección por calquera virus é a súa capacidade de entrar na célula e producir partículas máis infecciosas. Pénsase que a presenza do CD155 define os animais e os tecidos que poden ser infectados polo poliovirus. O CD155 atópase (fora dos laboratorios) só nas células humanas, de primates superiores e de monos do Vello Mundo. Porén, o poliovirus é estritramente un patóxeno humano e non infecta de forma natural a ningunha outra especie (aínda que o chimpancé común e os monos do Vello Mundo poden ser infectados experimentalmente).[45]
O xene CD155 parece que estivo sometido a selección positiva.[46] A súa proteína ten varios dominios, dos cales o dominio D1 contén o sitio de unión para o virus da polio. Dentro deste dominio, hai 37 aminoácidos que son os responsables da unión do virus.
O poliovirus é un enterovirus. A infección ocorre pola ruta fecal-oral, o que significa que se inxire o virus e a replicación viral ten lugar no tracto gastrointestinal.[47] O virus atópase nas feces dos individuos infectados. No 95% dos casos só se produce unha presenza primaria transitoria de virus no sangue (viremia), e a infección por poliovirus é asintomática. En aproximadamente o 5% dos casos, o virus espállase e replícase noutras partes do corpo, como na graxa marrón, tecido reticuloendotelial e músculo. A replicación viral sostida causa unha viremia secundaria e orixina o desenvolvemento de síntomas menores, como febre, dor de cabeza e de gorxa.[48] A poliomielite paralítica prodúcese en menos do 1% das infeccións por poliovirus. A enfermidade paralítica ocorre cando o virus entra no sistema nervioso central (SNC) e replícase en motoneuronas da medula espiñal, tronco cerebral ou córtex motor, causando a destrución selectiva de neuronas motoras que levan a unha parálise temporal ou permanente. Isto é algo moi raro en bebés, que aínda teñen anticorpos antipoliovirus recibidos das súas nais.[49] En raros casos, a poliomielite paralítica causa parada respiratoria e morte. En casos de enfermidade paralítica, obsérvanse con frecuencia dor muscular e espasmos antes do comezo da debilidade e parálise. A parálise persiste tipicamente durante días a semanas antes de recuperación.[50]
En moitos aspectos a fase neurolóxica da infección pénsase que é unha desviación accidental da infección gastrointestinal normal.[16] O mecanismo polo cal o poliovirus entra no SNC non se coñece ben. Presentáronse tres hipóteses non mutuamente excluíntes para explicar esta entrada. Todas elas requiren unha viremia primaria. A primeira hipótese predí que os virións pasan directamente desde o sangue ao sistema nervioso central cruzando a barreira hematoencefálica independente de CD155.[51] Unha segunda hipótese propón que os virións son transportados desde os tecidos periféricos que foron bañados polo sangue virémico, por exemplo o tecido muscular, ata a medula espiñal a través de nervios por transporte axonal retrógrado.[52][53][54] A terceira hipótese é que o virus é importado ao SNC por medio de monocitos ou macrófagos infectados.[10]
A poliomielite é unha doenza do sistema nervioso central. Porén, o CD155 crese que está presente na superficie da maioría ou de todas as células humanas. Por tanto, a expresión do receptor non explica por que o poliovirus infecta preferencialmente certos tecidos. Isto suxire que o tropismo de tecido está determinado pola infección celular. Recentes traballos suxeriron que a resposta do interferón tipo I (especificamente a dos interferóns alfa e beta) é un factor importante que define os tipos de células que poden soster a replicación dos poliovirus.[55] En ratos que expresan o CD155 (por medio de enxeñaría xenética) pero que carecen do receptor do interferón tipo I, o poliovirus non só se replica nun repertorio ampliado de tipos de tecidos, senón que estes ratos poden tamén ser infectados oralmente co virus.[56]
Evitación do sistema inmunitario editar
O poliovirus usa dous mecanismos clave para evadirse da acción do sistema inmunitario. Primeiro, pode sobrevivir ás condicións moi ácidas do estómago, o que lle permite infectar as células hóspede e espallarse polo corpo a través do sistema linfático.[3] Segundo, como pode replicarse moi rapidamente, o virus invade os órganos hóspede antes de que a resposta inmune poida poñerse en funcionamento.[57] Os poliovirus con canóns na superficie do virión teñen sitios de adhesión localizados en petos na base dos canóns. Os canóns son demasiado estreitos como para que accedan a eles os anticorpos, polo que os sitios de adhesión do virus están protexidos da vixilancia inmunitaria do hóspede, mentres que o resto da superficie do virión pode mutar para evitar a resposta inmune do hóspede.[58]
Os individuos que foron expostos ao poliovirus, sexa por infección sexa por inmunización coa vacina da polio, desenvolven inmunidade. Nos individuos inmunes, os anticorpos contra o poliovirus esán presentes nas amígdalas e tracto gastrointestinal (concretamente os anticorpos IgA) e poden bloquear a replicación do poliovirus; os anticorpos IgG e IgM contra o poliovirus poden previr o espallamento do virus a motoneuronas do sistema nervioso central.[33] A infección cun serotipo de poliovirus non proporciona inmunidade contra os outros serotipos; porén, un segundo ataque contra un mesmo individuo é extremadamente raro. Non obstante, as vacinas fanse utilizando todos ou case todos os serotipos.[59]
Rato transxénico PVR editar
Aínda que os humanos son os únicos hóspedes naturais coñecidos do poliovirus, os monos poden ser infectados experimentalmente e foron usados desde hai moito tempo para estudar o poliovirus. En 1990–91, dous laboratorios desenvolveron un pequeno modelo animal de poliomielite. Uns ratos foron modificados por enxeñaría xenética para que expresasen o receptor humano do poliovirus (hPVR).[60][61]
A diferenza dos ratos normais, os ratos con receptor do poliovirus transxénico (TgPVR) son susceptibles ao poliovirus inxectado intravenosa ou intramuscularmente e cando se inxectan directamente na medula espiñal ou no cerebro.[62] Despois da infección, os ratos TgPVR mostran signos de parálise que lembran os da poliomielite en humanos e monos, e os sistemas nerviosos centrais dos ratos paralizados son similares histocitoquimicamente aos de humanos e monos. Este modelo de ratos da infección por poliovirus humana demostrou ser unha ferramenta moi valiosa para entendermos a bioloxía do poliovirus e a súa patoxenicidade.[63]
Estudáronse ben tres tipos de ratos TgPVR:[64]
- Nos ratos TgPVR1 o transxene que codifica o PVR humano foi incorporado ao cromosoma 4 do rato. Estes ratos expresan altos niveis do transxene e as maiores sensibilidades para o poliovirus. Os ratos TgPVR1 son susceptibles ao poliovirus polas vías intraespiñal, intracerebral, intramuscular e intravenosa, pero non pola ruta oral.
- Os ratos TgPVR21 incorporaron o PVR humano no seu cromosoma 13. Estes ratos son menos susceptibles á infección por poliovirus a través da ruta intracerebral, posiblemente porque expresan menores niveis de hPVR. Os ratos TgPVR21 son susceptibles á infección por poliovirus por inoculación intranasal e poden ser útiles como un modelo de infección na mucosa.[65]
- Nos ratos TgPVR5 o transxene humano está localizado no seu cromosoma 12. Estes ratos presentan os niveis mais baixos de expresión de hPVR e son os menos susceptibles á infección por poliovirus.
Recentemente, desenvolveuse un cuarto modelo de rato TgPVR. Estes ratos "cPVR" portan ADNc hPVR, dirixido por un promotor de β-actina, e demostraron ser susceptibles ao poliovirus polas rutas intracerebral, intramuscular e intranasal. Ademais, estes ratos poden desenvolver a forma bulbar da polio por inoculación intranasal.[65]
O desenvolvemento do rato TgPVR tivo un profundo efecto na produción da vacina oral da polio (OPV). Previamente, a monitorización da seguridade da vacina oral da polio tiña que realizarse en monos, porque só os primates son susceptibles ao virus. En 1999, a Organización Mundial da Saúde aprobou o uso dos ratos TgPV como un método alternativo de avaliar a efectividade da vacina contra o poliovirus tipo 3. En 2000, o modelo de ratos foi aprobado para tests de vacinas contra os poliovirus tipo 1 e 2.[66]
Clonación e síntese editar
En 1981, Racaniello e Baltimore usaron a tecnoloxía do ADN recombinante para xerar o primeiro clon infeccioso dun virus de ARN animal, o poliovirus. Introduciuse un ADN que codificaba o xenoma de ARN de poliovirus en células de mamífero cultivadas e orixinou un poliovirus infeccioso.[67] A creación de clons infecciosos impulsou o coñecemento da bioloxía do poliovirus e converteuse nunha tecnoloxía estándar usada no estudo de moitos outros virus.
En 2002, o grupo de Eckard Wimmer na Universidade de Stony Brook (Nova York) tivo éxito en sintetizar o poliovirus a partir do seu código químico, producindo o primeiro virus sintético.[68] Os científicos converteron primeiro a secunecia de ARN de poliovirus publicada, de 7 741 bases de longo, nunha secuencia de ADN, xa que o ADN era máis fácil de sintetizar. Conseguíronse fragmentos curtos desta secuencia de ADN por envío postal por catálogo e ensambláronse. O xenoma viral completo foi despois ensamblado por unha compañía de síntese de xenes. Incorporáronse dezanove marcadores ao ADN sintetizado, para que se puidese distinguir do poliovirus natural. Utilizáronse encimas para converter o ADN de novo en ARN, que é o seu estado natural. Outoros encimas utilizáronse para traducir o ARN a un polipéptido, producindo unha partícula viral funcional. Este proceso meticuloso completo levoulles dous anos. O novo virus sintético acuñado foi inxectado nun rato transxénico PVR para determinar se a versión sintética podía causar a enfermidade. O virus sintético puido replicarse, infectar, e causar parálise ou morte nos ratos. Porén, a versión sintética era entre 1 000 e 10 000 veces máis débil que o virus orixinal, probablemente debido a un dos marcadores engadidos.[69]
Modificación para terapias editar
Unha modificación do poliovirus chamada PVSRIPO foi testada en ensaios clínicos iniciais en 2014 como posible tratamento para o cancro.[70]
Notas editar
- ↑ Ryan KJ, Ray CG, eds. (2004). Sherris Medical Microbiology (4th ed.). McGraw Hill. ISBN 978-0-8385-8529-0.
- ↑ 2,0 2,1 Hogle JM (2002). "Poliovirus cell entry: common structural themes in viral cell entry pathways". Annual Review of Microbiology 56: 677–702. PMC 1500891. PMID 12142481. doi:10.1146/annurev.micro.56.012302.160757.
- ↑ 3,0 3,1 3,2 Goodsell DS (1998). The machinery of life. New York: Copernicus. ISBN 978-0-387-98273-1.
- ↑ 4,0 4,1 Paul JR (1971). A History of Poliomyelitis. (Yale studies in the history of science and medicine). New Haven, Conn: Yale University Press. ISBN 978-0-300-01324-5.
- ↑ "Behind the picture: Rosalind Franklin and the polio model". Medical Research Council. 2019-03-14. Arquivado dende o orixinal o 30 de outubro de 2018. Consultado o 4 September 2019.
- ↑ Maddox B (2003). Rosalind Franklin: The Dark Lady of DNA. London: Harper Collins. p. 296. ISBN 0-00-655211-0.
- ↑ Brown A (2007). J.D. Bernal: The Sage of Science. New York: Oxford University Press. pp. 359–361. ISBN 978-0-19-920565-3.
- ↑ Racaniello VR, Baltimore D (August 1981). "Molecular cloning of poliovirus cDNA and determination of the complete nucleotide sequence of the viral genome". Proceedings of the National Academy of Sciences of the United States of America 78 (8): 4887–91. Bibcode:1981PNAS...78.4887R. PMC 320284. PMID 6272282. doi:10.1073/pnas.78.8.4887.
- ↑ Kitamura N, Semler BL, Rothberg PG, Larsen GR, Adler CJ, Dorner AJ, et al. (June 1981). "Primary structure, gene organization and polypeptide expression of poliovirus RNA". Nature 291 (5816): 547–53. Bibcode:1981Natur.291..547K. PMID 6264310. doi:10.1038/291547a0.
- ↑ 10,0 10,1 10,2 De Jesus NH (July 2007). "Epidemics to eradication: the modern history of poliomyelitis". Virology Journal 4 (1): 70. PMC 1947962. PMID 17623069. doi:10.1186/1743-422X-4-70.
- ↑ Mendelsohn CL, Wimmer E, Racaniello VR (March 1989). "Cellular receptor for poliovirus: molecular cloning, nucleotide sequence, and expression of a new member of the immunoglobulin superfamily". Cell 56 (5): 855–65. PMID 2538245. doi:10.1016/0092-8674(89)90690-9.
- ↑ 12,0 12,1 He Y, Mueller S, Chipman PR, Bator CM, Peng X, Bowman VD, et al. (April 2003). "Complexes of poliovirus serotypes with their common cellular receptor, CD155". Journal of Virology 77 (8): 4827–35. PMC 152153. PMID 12663789. doi:10.1128/JVI.77.8.4827-4835.2003.
- ↑ Dunnebacke TH, Levinthal JD, Williams RC (October 1969). "Entry and release of poliovirus as observed by electron microscopy of cultured cells". Journal of Virology 4 (4): 505–13. PMC 375900. PMID 4309884. doi:10.1128/JVI.4.4.505-513.1969.
- ↑ Kaplan G, Freistadt MS, Racaniello VR (October 1990). "Neutralization of poliovirus by cell receptors expressed in insect cells". Journal of Virology 64 (10): 4697–702. PMC 247955. PMID 2168959. doi:10.1128/JVI.64.10.4697-4702.1990.
- ↑ Gómez Yafal A, Kaplan G, Racaniello VR, Hogle JM (November 1993). "Characterization of poliovirus conformational alteration mediated by soluble cell receptors". Virology 197 (1): 501–5. PMID 8212594. doi:10.1006/viro.1993.1621.
- ↑ 16,0 16,1 Mueller S, Wimmer E, Cello J (August 2005). "Poliovirus and poliomyelitis: a tale of guts, brains, and an accidental event". Virus Research 111 (2): 175–93. PMID 15885840. doi:10.1016/j.virusres.2005.04.008.
- ↑ Brandenburg B, Lee LY, Lakadamyali M, Rust MJ, Zhuang X, Hogle JM (July 2007). "Imaging poliovirus entry in live cells". PLOS Biology 5 (7): e183. PMC 1914398. PMID 17622193. doi:10.1371/journal.pbio.0050183.
- ↑ Attardi G, Smith J (1962). "Virus specific protein and a ribo-nucleic acid associated with ribosomes in poliovirus infected HeLa cells". Cold Spring Harbor Symposia on Quantitative Biology 27: 271–92. PMID 13965389. doi:10.1101/SQB.1962.027.001.026.
- ↑ Lozano G, Fernandez N, Martinez-Salas E (February 2016). "Modeling Three-Dimensional Structural Motifs of Viral IRES". Journal of Molecular Biology 428 (5 Pt A): 767–776. PMID 26778619. doi:10.1016/j.jmb.2016.01.005.
- ↑ Chen CY, Sarnow P (April 1995). "Initiation of protein synthesis by the eukaryotic translational apparatus on circular RNAs". Science 268 (5209): 415–7. Bibcode:1995Sci...268..415C. PMID 7536344. doi:10.1126/science.7536344.
- ↑ Pelletier J, Sonenberg N (July 1988). "Internal initiation of translation of eukaryotic mRNA directed by a sequence derived from poliovirus RNA". Nature 334 (6180): 320–5. Bibcode:1988Natur.334..320P. PMID 2839775. doi:10.1038/334320a0.
- ↑ Jang SK, Kräusslich HG, Nicklin MJ, Duke GM, Palmenberg AC, Wimmer E (August 1988). "A segment of the 5' nontranslated region of encephalomyocarditis virus RNA directs internal entry of ribosomes during in vitro translation". Journal of Virology 62 (8): 2636–43. PMC 253694. PMID 2839690. doi:10.1128/JVI.62.8.2636-2643.1988.
- ↑ Carter J, Saunders VA (2007). Virology: Principles and Applications. John Wiley & Sons. p. 4. ISBN 978-0-470-02386-0.
- ↑ 24,0 24,1 Carter J, Saunders VA (2007). Virology: Principles and Applications. John Wiley & Sons. p. 165. ISBN 978-0-470-02386-0.
- ↑ Harper DR (2012). Viruses: Biology, Applications, and Control. The United States of America: Garland Science. ISBN 978-0-8153-4150-5.
- ↑ "Poliomyelitis". Brown University. Arquivado dende o orixinal o 22 de febreiro de 2007. Consultado o 07 de agosto de 2020.
- ↑ Carter J, Saunders VA (2007). Virology: Principles and Applications. John Wiley & Sons. p. 164. ISBN 978-0-470-02386-0.
- ↑ Louten J (2016). "Poliovirus". Essential Human Virology. Elsevier. pp. 257–271. ISBN 978-0-12-800947-5. doi:10.1016/b978-0-12-800947-5.00014-4.
- ↑ Murray KE, Barton DJ (April 2003). "Poliovirus CRE-dependent VPg uridylylation is required for positive-strand RNA synthesis but not for negative-strand RNA synthesis". Journal of Virology 77 (8): 4739–50. PMC 152113. PMID 12663781. doi:10.1128/JVI.77.8.4739-4750.2003.
- ↑ Goodfellow IG, Kerrigan D, Evans DJ (January 2003). "Structure and function analysis of the poliovirus cis-acting replication element (CRE)". RNA 9 (1): 124–37. PMC 1370376. PMID 12554882. doi:10.1261/rna.2950603.
- ↑ Carter J, Saunders VA (2007). Virology: Principles and Applications. John Wiley & Sons. pp. 161, 165. ISBN 978-0-470-02386-0.
- ↑ Carter J, Saunders VA (2007). Virology: Principles and Applications. John Wiley & Sons. p. 166. ISBN 978-0-470-02386-0.
- ↑ 33,0 33,1 33,2 33,3 Kew OM, Sutter RW, de Gourville EM, Dowdle WR, Pallansch MA (2005). "Vaccine-derived polioviruses and the endgame strategy for global polio eradication". Annual Review of Microbiology 59: 587–635. PMID 16153180. doi:10.1146/annurev.micro.58.030603.123625.
- ↑ Drake JW (August 1958). "Interference and multiplicity reactivation in polioviruses". Virology 6 (1): 244–64. PMID 13581529. doi:10.1016/0042-6822(58)90073-4.
- ↑ Kirkegaard K, Baltimore D (November 1986). "The mechanism of RNA recombination in poliovirus". Cell 47 (3): 433–43. PMC 7133339. PMID 3021340. doi:10.1016/0092-8674(86)90600-8.
- ↑ Barr JN, Fearns R (June 2010). "How RNA viruses maintain their genome integrity". The Journal of General Virology 91 (Pt 6): 1373–87. PMID 20335491. doi:10.1099/vir.0.020818-0.
- ↑ Bernstein H, Bernstein C, Michod RE (January 2018). "Sex in microbial pathogens". Infection, Genetics and Evolution 57: 8–25. PMID 29111273. doi:10.1016/j.meegid.2017.10.024.
- ↑ Jiang P, Faase JA, Toyoda H, Paul A, Wimmer E, Gorbalenya AE (May 2007). "Evidence for emergence of diverse polioviruses from C-cluster coxsackie A viruses and implications for global poliovirus eradication". Proceedings of the National Academy of Sciences of the United States of America 104 (22): 9457–62. Bibcode:2007PNAS..104.9457J. PMC 1874223. PMID 17517601. doi:10.1073/pnas.0700451104.
- ↑ Jorba J, Campagnoli R, De L, Kew O (May 2008). "Calibration of multiple poliovirus molecular clocks covering an extended evolutionary range". Journal of Virology 82 (9): 4429–40. PMC 2293050. PMID 18287242. doi:10.1128/JVI.02354-07.
- ↑ Rothberg PG, Wimmer E (December 1981). "Mononucleotide and dinucleotide frequencies, and codon usage in poliovirion RNA". Nucleic Acids Research 9 (23): 6221–9. PMC 327599. PMID 6275352. doi:10.1093/nar/9.23.6221.
- ↑ Zhang J, Wang M, Liu WQ, Zhou JH, Chen HT, Ma LN, et al. (March 2011). "Analysis of codon usage and nucleotide composition bias in polioviruses". Virology Journal 8: 146. PMC 3079669. PMID 21450075. doi:10.1186/1743-422X-8-146.
- ↑ "Global eradication of wild poliovirus type 2 declared". Global Polio Eradication Initiative. 2015-09-20. Consultado o 2015-09-30.
- ↑ "GPEI-Two out of three wild poliovirus strains eradicated" (en inglés). Consultado o 2019-11-02.
- ↑ Carstens EB, Ball LA (July 2009). "Ratification vote on taxonomic proposals to the International Committee on Taxonomy of Viruses (2008)". Archives of Virology 154 (7): 1181–8. PMC 7086627. PMID 19495937. doi:10.1007/s00705-009-0400-2.
- ↑ Mueller S, Wimmer E (August 2003). "Recruitment of nectin-3 to cell-cell junctions through trans-heterophilic interaction with CD155, a vitronectin and poliovirus receptor that localizes to alpha(v)beta3 integrin-containing membrane microdomains". The Journal of Biological Chemistry 278 (33): 31251–60. PMID 12759359. doi:10.1074/jbc.M304166200.
- ↑ Suzuki Y (May 2006). "Ancient positive selection on CD155 as a possible cause for susceptibility to poliovirus infection in simians". Gene 373: 16–22. PMID 16500041. doi:10.1016/j.gene.2005.12.016.
- ↑ Bodian D, Horstmann DH (1969). Polioviruse. Philadelphia, Penn: Lippincott. pp. 430–73.
- ↑ Sabin AB (June 1956). "Pathogenesis of poliomyelitis; reappraisal in the light of new data". Science 123 (3209): 1151–7. Bibcode:1956Sci...123.1151S. PMID 13337331. doi:10.1126/science.123.3209.1151.
- ↑ Carter JB, Saunders VA (2007). Virology: Principles and Applications (1st ed.). Chichester, England: John Wiley & Sons. ISBN 978-0-470-02386-0. OCLC 124160564.
- ↑ EMedicine 306440 Acute Poliomyelitis
EMedicine 967950 Pediatric Poliomyelitis - ↑ Yang WX, Terasaki T, Shiroki K, Ohka S, Aoki J, Tanabe S, et al. (March 1997). "Efficient delivery of circulating poliovirus to the central nervous system independently of poliovirus receptor". Virology 229 (2): 421–8. PMID 9126254. doi:10.1006/viro.1997.8450.
- ↑ Ohka S, Yang WX, Terada E, Iwasaki K, Nomoto A (October 1998). "Retrograde transport of intact poliovirus through the axon via the fast transport system". Virology 250 (1): 67–75. PMID 9770421. doi:10.1006/viro.1998.9360.
- ↑ Ren R, Racaniello VR (October 1992). "Poliovirus spreads from muscle to the central nervous system by neural pathways". The Journal of Infectious Diseases 166 (4): 747–52. PMID 1326581. doi:10.1093/infdis/166.4.747.
- ↑ Lancaster KZ, Pfeiffer JK (March 2010). Gale Jr M, ed. "Limited trafficking of a neurotropic virus through inefficient retrograde axonal transport and the type I interferon response". PLOS Pathogens 6 (3): e1000791. PMC 2832671. PMID 20221252. doi:10.1371/journal.ppat.1000791.
- ↑ Ida-Hosonuma M, Iwasaki T, Yoshikawa T, Nagata N, Sato Y, Sata T, et al. (April 2005). "The alpha/beta interferon response controls tissue tropism and pathogenicity of poliovirus". Journal of Virology 79 (7): 4460–9. PMC 1061561. PMID 15767446. doi:10.1128/JVI.79.7.4460-4469.2005.
- ↑ Ohka S, Igarashi H, Nagata N, Sakai M, Koike S, Nochi T, et al. (August 2007). "Establishment of a poliovirus oral infection system in human poliovirus receptor-expressing transgenic mice that are deficient in alpha/beta interferon receptor". Journal of Virology 81 (15): 7902–12. PMC 1951287. PMID 17507470. doi:10.1128/JVI.02675-06.
- ↑ Racaniello VR (January 2006). "One hundred years of poliovirus pathogenesis". Virology 344 (1): 9–16. PMID 16364730. doi:10.1016/j.virol.2005.09.015.
- ↑ Carter JB, Saunders VA (2007). Virology: Principles and Applications. Liverpool John Moores University, UK: John Wiley & Sons. ISBN 978-0-470-02386-0.
- ↑ Organización Mundial da Saúde vacina da polio. Cita:"A vacina da polio inactivada foi presentada por primeira vez en 1955 e prodúcese a partir de cepas de poliovirus de tipo salvaxe de cada serrotipo que foron inactivadas (matadas) con formol" // "As vacinas da polio orais foron presentadas por primeira vez en 1961 e constan dunha mestura de tres serotipos de poliovirus vivos atenuados (Sabin tipos 1, 2 e 3), seleccionados pola súa baixa neurovirulencia e reducida transmisibilidade."
- ↑ Ren RB, Costantini F, Gorgacz EJ, Lee JJ, Racaniello VR (October 1990). "Transgenic mice expressing a human poliovirus receptor: a new model for poliomyelitis". Cell 63 (2): 353–62. PMID 2170026. doi:10.1016/0092-8674(90)90168-E.
- ↑ Koike S, Taya C, Kurata T, Abe S, Ise I, Yonekawa H, Nomoto A (February 1991). "Transgenic mice susceptible to poliovirus". Proceedings of the National Academy of Sciences of the United States of America 88 (3): 951–5. Bibcode:1991PNAS...88..951K. PMC 50932. PMID 1846972. doi:10.1073/pnas.88.3.951.
- ↑ Horie H, Koike S, Kurata T, Sato-Yoshida Y, Ise I, Ota Y, et al. (February 1994). "Transgenic mice carrying the human poliovirus receptor: new animal models for study of poliovirus neurovirulence". Journal of Virology 68 (2): 681–8. PMC 236503. PMID 8289371. doi:10.1128/JVI.68.2.681-688.1994.
- ↑ Ohka S, Nomoto A (October 2001). "Recent insights into poliovirus pathogenesis". Trends in Microbiology 9 (10): 501–6. PMID 11597452. doi:10.1016/S0966-842X(01)02200-4.
- ↑ Koike S, Taya C, Aoki J, Matsuda Y, Ise I, Takeda H, et al. (1994). "Characterization of three different transgenic mouse lines that carry human poliovirus receptor gene--influence of the transgene expression on pathogenesis". Archives of Virology 139 (3–4): 351–63. PMID 7832641. doi:10.1007/BF01310797.
- ↑ 65,0 65,1 Nagata N, Iwasaki T, Ami Y, Sato Y, Hatano I, Harashima A, et al. (March 2004). "A poliomyelitis model through mucosal infection in transgenic mice bearing human poliovirus receptor, TgPVR21". Virology 321 (1): 87–100. PMID 15033568. doi:10.1016/j.virol.2003.12.008.
- ↑ Dragunsky E, Nomura T, Karpinski K, Furesz J, Wood DJ, Pervikov Y, et al. (2003). "Transgenic mice as an alternative to monkeys for neurovirulence testing of live oral poliovirus vaccine: validation by a WHO collaborative study". Bulletin of the World Health Organization 81 (4): 251–60. PMC 2572431. PMID 12764491. doi:10.1590/S0042-96862003000400006 (inactivo 2020-05-15).
- ↑ Racaniello VR, Baltimore D (November 1981). "Cloned poliovirus complementary DNA is infectious in mammalian cells". Science 214 (4523): 916–9. Bibcode:1981Sci...214..916R. PMID 6272391. doi:10.1126/science.6272391.
- ↑ Cello J, Paul AV, Wimmer E (August 2002). "Chemical synthesis of poliovirus cDNA: generation of infectious virus in the absence of natural template". Science 297 (5583): 1016–8. Bibcode:2002Sci...297.1016C. PMID 12114528. doi:10.1126/science.1072266.
- ↑ Couzin J (July 2002). "Virology. Active poliovirus baked from scratch". Science 297 (5579): 174–5. PMID 12114601. doi:10.1126/science.297.5579.174b.
- ↑ Brown MC, Dobrikova EY, Dobrikov MI, Walton RW, Gemberling SL, Nair SK, et al. (November 2014). "Oncolytic polio virotherapy of cancer". Cancer 120 (21): 3277–86. PMC 4205207. PMID 24939611. doi:10.1002/cncr.28862.
Véxase tamén editar
Ligazóns externas editar
- ICTVdb virus classification 2006
- Home of Picornaviruses (latest updates of species, serotypes, & proposed changes)Arquivado 04 de novembro de 2010 en Wayback Machine.
- Goodsell D. "Poliovirus and Rhinovirus". August 2001 Molecule of the Month. Arquivado dende o orixinal o 03 de marzo de 2011. Consultado o 20 de xaneiro de 2021.
- 3D macromolecular structures of the Poliovirus archived in the EM Data Bank(EMDB)
- "Human poliovirus 1". NCBI Taxonomy Browser. 12080.
- "Human poliovirus 3". NCBI Taxonomy Browser. 12086.
