Mutación puntual
Unha mutación puntual é unha mutación xenética na que se cambia unha soa base nucleotídica, que é substituída, inserida ou eliminada dunha secuencia de ADN ou ARN.[1] As mutacións puntuais teñen unha variedade de efectos sobre o produto proteico orixinado ao final do proceso, consecuencias que son en parte predicibles baseándose nas especificidades da mutación. Estas consecuencias poden variar desde ser benignas (por exemplo nunha mutación sinónima) a catastróficas (por exemplo nunha mutación de cambio da pauta de lectura), con respecto á produción, composición e función das proteínas.

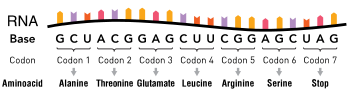
Causas
editarAs mutacións puntuais normalmente prodúcense durante a replicación do ADN. A replicación do ADN ocorre cando unha molécula de ADN bicatenaria se separa creando dúas febras de ADN monocatenarias, cada unha das cales é un molde para a formación da febra complementaria. Unha soa mutación puntual pode cambiar unha secuencia completa de ADN por un cambio de pauta de lectura. Cambiando unha purina ou pirimidina pode cambiar só o aminoácido que o triplete de nucleótidos codificaba.
As mutacións puntuais poden orixinarse por mutacións espontáneas que ocorren durante a replicación do ADN. A taxa de mutación pode incrementarse pola presenza de mutáxenos. Os mutáxenos poden ser físicos, como a radiación por raios UV, raios X ou calor extremo, ou químicos (moléculas que provocan que se coloquen en mal lugar as bases ou distorsionan a forma helicoidal do ADN). Os mutáxenos asociados con cancros son a miúdo estudados para aprender sobre o cancro e a súa prevención.
Hai múltiples xeitos de que ocorran mutacións puntuais. Primeiro, a luz ultravioleta e luz de alta frecuencia poden causar ionizacións, que á súa vez poden afectar o ADN. As especies reactivas do oxíxeno con radicais libres, que son o subproduto do metabolismo celular, poden tamén ser moi nocivas para o ADN. Estes reactivos poden orixinar roturas nunha soa febra do ADN e nas dúas febras. Terceiro, os enlaces do ADN acaban por degradarse, o que crea outro problema para manter a integridade do ADN en perfectas condicións. Tamén pode haber erros de replicación que orixinen mutacións por substitución, inserción ou deleción.
Clasificación
editarTransición/transversión
editarEn 1959 Ernst Freese acuñou os termos "transicións" ou "transversións" para clasificar os diferentes tipos de mutacións puntuais por substitución.[2][3] As transicións son substitucións dunha base púrica por outra tamén púrica ou a substitución dunha base pirimidínica por outra tamén pirimidínica. As transversións son substitucións dunha base púrica por unha pirimidínica ou viceversa. Hai unha diferenza sistemática nas taxas de mutación para as transicións e as transversións, xa que as mutacións por transición son unhas dez veces máis comúns que as transversións.
Categorización funcional
editarAs mutacións sen sentido inclúen as de ganancia de stop e perda de stop. As de ganancia de stop son mutacións resultado da formación dun codón de terminación ou stop prematuro novo (gañouse un stop), que indican o final da tradución. Esta interrupción causa que a proteína quede acurtada anormalmente. Do número de aminoácidos perdidos depende o impacto sobre a funcionalidade da proteína, que pode non ser funcional.[4] As de perda de stop son mutacións no codón de terminación orixinal, que desaparece (perdeuse un stop), o que ten como resultado a extensión anormal da porción do carboxilo terminal da proteína. As chamadas ganancias de inicio crean un codón de iniciación AUG augas arriba do codón de iniciación orixinal. Se o novo AUG está preto sitio do orixinal, en pauta no transcrito procesado e augas abaixo do sitio de unión ao ribosoma, pode usarse para iniciar a tradución. O efecto probable é que se engadan aminoácidos adicionais na porción amino terminal da proteína orixinal. As mutacións de cambio de pauta de lectura son tamén posibles en mutacións de ganancia de inicio, pero normalmente non afectan a tradución da proteína orixinal. A perda de inicio é unha mutación puntual nun codón de iniciación AUG do transcrito, tendo como resultado a redución ou eliminación da produción de proteínas.
As mutacións de cambio de sentido codifican un aminoácido diferente do orixinal. Unha mutación con cambio de sentido cambia un codón de maneira que se crea unha proteína diferente, que contén un cambio non sinónimo.[4] As mutacións conservadoras provocan o cambio dun aminoácido, pero as propiedades deste aminoácido son similares ás do orixinal (os dous eran hidrófilos, hidrófobos etc.). Por veces, un cambio dun aminoácido na proteína non é prexudicial para o organismo. A maioría das proteínas poden soportar unha ou dúas mutacións puntuais deste tipo antes de que a súa función cambie. As mutacións non conservadoras causan o cambio a un aminoácido con propiedades diferentes que as do tipo silvestre. A proteína pode perder máis facilmente a súa función, o cal causa unha enfermidade no organismo. Por exemplo, a anemia falciforme está causada por unha soa mutación puntual (unha mutación de cambio de sentido) no xene da beta-hemoglobina, que converte o codón GAG en GUG, o cal codifica o aminoácido valina (hidrófobo e neutro) en vez do orixinal ácido glutámico (cargado e polar). A proteína pode tamén mostrar unha "ganancia de función" ou quedar inactivada, como é o caso da mutation no xene BRAF de cambio de valina a ácido glutamico (ao revés que na anemia falciciforme); isto orixina a activación indebida da proteína RAF, que causa unha sinalización proliferativa ilimitada en células cancerosas.[5] Estes dous son exemplos de mutacións de cambio de sentido non conservadoras.
As mutacións silenciosas codifican o mesmo aminoácido que o orixinal (trátase dunha "substitución sinónima"). Unha mutación silenciosa non afecta o fundionamento da proteína. Pode cambiar un só nucleótido, pero de maneira que o novo codón especifique o mesmo aminoácido (o que é posible debido á dexeneración do código xenético), tendo como resultado unha proteína inmutada. Este tipo de cambio denomínase sinónimo porque o vello e o novo codóns codifican o mesmo aminoácido. Isto é posible porque 64 codóns especifican só 20 aminoácidos e hai redundancia no código. Non obstante, diferentes codóns sinónimos poden orixinar uns niveis diferenciais de expresión proteica.[4]
Delecións e insercións dun só par de bases
editarÁs veces o termo mutación puntual utilízase para describir insercións ou delecións dun só par de bases. Isto ten efectos moito máis adversos sobre a proteína sintetizada debido que ao ler os nucleótidos de tres en tres e faltar (ou sobrar) un está movida a pauta de lectura, polo que se len en diferentes marcos e cambian multitude de codóns (mutación de cambio de pauta de lectura).[4]
Consecuencias xerais
editarAs mutacións puntuais que ocorren en secuencias non codificantes adoitan non ter consecuencias, aínda que hai excepcións. Se o par de bases mutado está na secuencia promotora dun xene, entón a expresión do xene pode cambiar. Además, se a mutación ocorre no sitio de empalme dun intrón, entón isto pode interferir co empalme correcto do pre-ARNm transcrito.
Ao alterar só un aminoácido, a proteína completa pode cambiar. A nova proteína denomínase variante proteica. Se a proteína orixinal funciona na reprodución celular entón esta mutación puntual única pode cambiar todo o proceso da reprodución celular neste organismo.
As mutacións na liña xerminal puntuais poden orixinar caracteres beneficiosos ou daniños ou enfermidades. Isto leva a adaptacións baseada no ambiente onde vive o organismo. Unha mutación vantaxosa pode crear unha vantaxe para ese organismo e facer que o carácter se transmita dunha xeración a outra, mellorando e beneficiándose toda a poboación. Na teoría científica da evolución son moi importantes as mutacións puntuais. A teoría explica que a diversidade e a historia dos seres vivos da Terra. En relación a mutacións puntuais, as mutacións beneficiosas permiten que o organismo prospere e se reproduza, transmitíndose así os xenes mutados que afectan postivamente á fitness á seguinte xeración. Ao contrario ocorre coas mutacións nocivas, debido á selección natural.
Hai diferentes efectos a curto e longo prazo que poden orixinarse por mutacións. Os máis leves serían unha detención do ciclo celular en numerosos puntos. Isto significa que un codón que codifica o aminoácido glicina pode cambiar a un codón de stop, causando que as proteínas sexan anormais e incapaces de realizar as súas funcións na célula. As mutacións poden impedir que se produza a mitose. Poden crear problemas tamén nos procesos de transcrición e replicación do ADN. Isto pode levar á morte da célula. Os efectos a longo prazo poden ser un cambio permanente nun cromosoma. Unha consecuencia pode ser o cancro.[6]
Outros efectos das mutacións puntuais ou polimorfismos dun só nucleótido ou SNP, dependen da localizacón da mutación dentro do xene. Por exemplo, se a mutación ocorre na rexión do xene responsable da codificación, a secuencia de aminoácidos da proteína codificada pode ser alterada, causando un cambio na función, localización da proteína, estabilidade da proteína ou o complexo proteico. Propuxéronse moitos métodos de predicir os efectos de mutacións con cambio de sentido en proteínas. Poden probarse modelos utilizando algoritmos para distinguir mutacións asociadas a enfermidades coñecidas das mutacións neutras. Asúmese que os cambios en posicións conservadas tenden a ser máis deletéreos. Aínda que a maioría dos métodos proporcionan unha clasificación binaria dos efectos das mutacións en daniñas e benignas, cómpre utilizar un novo nivel de anotación para ofrecer unha explicación de por que e como estas mutacións danan as proteínas.[7]
As mutacións puntuais poden ter varios efectos sobre o comportamento e a reprodución dunha proteína dependendo de onde ocorre a mutación na secuencia de aminoácidos da proteína. Se a mutación ocorre na rexión do xene que codifica a proteínan, a secuencia de aminoácidos pode verse alterada. Este pequeno cambio pode causar un cambio na función e activación da proteína, o que significa como se une a encimas, onde está localizada na célula ou a cantidade de enerxía libre almacenada na proteína.
As mutacións poden fectar tamén á unión de factores de transcrición, os cales se unen por medio do recoñecemento de secuencias curtas. Unha mutación pode alterar estas secuencias e, así, cambia o modo en que se unen os factores de transcrición. Estas mutacións poden afectar á eficiencia da transcrición xénica, que controla tanto os niveis de ARNm coma de proteína.[8]
Enfermidades específicas causadas por mutacións puntuais
editarCancro
editarAs mutacións puntuais en múltiples proteínas supresoras de tumores causan cancro. Por exemplo, as mutacións puntuais na polipose adenotamosa de colon promoven a tumoroxénese.[9] Un novo ensaio, a proteólise paralela rápida (FASTpp), podería axudar a un cribado rápido de defectos na estabilidade específicos en pacientes de cancro.[10]
Neurofibromatose
editarA neurofibromatose é causada por mutacións puntuais nos xenes da neurofibromina 1[11][12] ou da neurofibromina 2.[13]
Anemia falciforme
editarA anemia falciforme é causada por unha mutación puntual na cadea de β-globina da hemoglobina, causando que o aminoácido hidrófilo ácido glutámico sexa substituído polo aminoácido hidrofóbico valina na posición sexta. A hemoglobina é unha proteína que se encontra nos glóbulos vermellos e transporta o oxíxeno polo corpo.[14] A hemoglobina está formada por dúas subunidades: beta-globinas e alfa-globinas.[15][16][17]
O xene da β-globina encóntrase no brazo curto do cromosoma 11. A asociación de dous tipos salvaxes de α-globina con dúas subniunidades mutantes de β-globia forma a hemoglobina S (HbS). En condicións de niveis de oxíxeno baixos (como a grande altitude, por exemplo), a ausencia dun aminoácido polar na posición seis da cadea de β-globina promove a polimerización non covalente de hemoglobina, o cal distorsiona a forma dos glóbulos vermellos que pasan a ter forma de lúa crecente ou falciforme e diminúe a súa elasticidade. Circulan peor polos capilares e o transporte de oxíxeno é menos eficiente.[18]
Secuencia da hemoglobina normal
| AUG | GUG | CAC | CUG | ACU | CCU | GAG | GAG | AAG | UCU | GCC | GUU | ACU |
| START | Val | His | Leu | Thr | Pro | Glu | Glu | Lys | Ser | Ala | Val | Thr |
Secuencia da hemoglobina falciforme ou S
| AUG | GUG | CAC | CUG | ACU | CCU | GUG | GAG | AAG | UCU | GCC | GUU | ACU |
| START | Val | His | Leu | Thr | Pro | Val | Glu | Lys | Ser | Ala | Val | Thr |
Enfermidade de Tay–Sachs
editarA causa da enfermidade de Tay–Sachs é un defecto xenético que se transmite de pais a fillos. Este defecto xenético está localizado no xene HEXA, que se encotra no cromosoma 15.
O xene HEXA forma parte dun encima chamado beta-hexosaminidase A, que xoga un papel crítico no sistema nervioso. Este encima axuda a degradar un lípido chamado gangliósido GM2 nas neuronas. As mutacións no xene HEXA distorsionan a actividade da beta-hexosaminidase A, impedindo a degradación deses lípidos. Como resultado, os gangliósidos acumúlanse ata niveis letais no cerebro e medula espiñal. A acumulación de gangliósido GM2 causa un dano progresivo ás células nerviosoas. Isto é a causa dos signos e síntomas da enfermidade de Tay-Sachs.[19]
Cegueira ás cores
editarAs persoas que teñen cegueira ás cores teñen mutacións nos seus xenes que causan unha perda dos conos para o vermello ou verde, e non poden distinguir esas cores. Hai tres tipos de conos no ollo humano: vermellos, verdes e azuis. Descubriuse que algunhas persoas coa mutación xénica que causa a cegueira ás cores perderon todo o conxunto completo de conos para unha cor pero sen que cambie a súa agudeza ou visión xeral.[20]
Mutación puntual inducida por repeticións
editarEn bioloxía molecular, unha mutación puntual inducida por repeticións (na literatura inglesa abreviada como RIP) é un proceso polo cal o ADN acumula mutacións por transicións de G:C a A:T. As evidencias xenómicas indican que estas mutacións ocorren ou ocorreron en diversos fungos[21] mentres que as probas experimentais indican que a RIP é activa en Neurospora crassa,[22] Podospora anserina,[23] Magnaporthe grisea,[24] Leptosphaeria maculans,[25] Gibberella zeae[26] e Nectria haematococca.[27] En Neurospora crassa, as secuencias mutadas por mutacións puntuais inducidas por repeticións son a miúdo metilatdas de novo.[22]
As mutacións puntuais inducidas por repeticións ocorren durante a etapa sexual en núcleos haploides despois da fertilización, pero antes da replicción do ADN meiótica.[22] En Neurospora crassa, as secuencias repetidas de polo menos 400 pares de bases de lonxitude son vulnerables a este tipo de mutcións. As repeticións con tan só un 80% de identidade de nucleótidos poden estar suxeitas a muracións puntuais inducidas por repeticións. Aínda que non se coñece ben o mecanismo exacto de recoñecemento de repeticións e de mutaxénese, estas mutacións teñen como resultado que as secuencis repetidas sofren múltiples mutacións por transición.
As mutacións RIP non parecen estar limitadas ás secuencias repetidas. De feito, por exemplo, no fungo fitopatóxeno L. maculans, estas mutacións encóntranse en rexións con secuencias cunha soa copia, adxacentes a elementos repetidos. Estas rexións poden ser rexións non codificantes ou xenes que codifican pequenas proteínas segregadas, incluíndo xenes de avirulencia. O grao de RIP dentro destas rexións dunha soa copia era proporcional á súa proximidade a elementos repetitivos.[28]
Rep e Kistler especularon que a presenza de rexións altamente repetitivas que conteñen transposóns, pode promover a mutación de xenes efectores residentes.[29] Así, suxeriuse que a presenza de xenes efectores dentro desas rexións promove a súa adaptación e diversificación cando se expón a unha forte presión selectiva.[30]
Estudáronse as relacións entre os xenes de avirulencia en Leptosphaeria maculans e as RIP,[31] e a asociación entre a recombinación e as RIP, que en N. crassa se informou que non están asociadas ; neste organismo as mutacións RIP espalláronse a secuencias dunha soa copia situadas a polo menos 930 pares de bases do límite das zonas con secuencias duplicadas veciñas.[32] Dilucidar o mecanismo de detección de secuencias repetidas que levan á formación de RIP pode servir para comprender como poden verse afectadas tamén as secuencias flanqueantes.
Mecanismo
editarAs mutacións puntuais inducidas por repeticións causan mutacións por transición de G:C a A:T dentro de repeticións; porén, o mecanismo que detecta as secuencias repetidas non se coñece. A única proteína coñecida esencial para a RIP é RID, a cal é unha proteína similar a unha ADN metiltransferease, que cando está mutada ou sometida a un knock out de xenes causa a perda das mutacións RIP.[33] A deleción do homólogo de rid en Aspergillus nidulans, dmtA, ten como resultado a perda de fertilidade,[34] mentres que a deleción do homólogo de rid en Ascobolus immersens, masc1, ten como resultado defectos de infertilidade e perda da metilación inducida premeioticamente (MIP).[35]
Consecuencias
editarA mutación puntual inducida por repeticións crese que evolucionou como un mecanismo de defensa contra os elementos transpoñibles, que lembran a parasitos ao invadiren e multiplicárense dentro dun xenoma. A RIP crea múltiples mutacións de cambio de sentido e sen sentido na secuencia codificante. Esta hipermutación de G-C a A-T en secuencias repetitivas elimina produtos xénicos funcionais da secuencia (se é que hai algún). Ademais, moitos dos nucleótidos que levan C son metilatdos, o que diminúe a transcrición.
Uso en bioloxía molecular
editarComo a RIP é tan eficiente detectando e mutando as repeticións, os biólogos que estudan os fungos úsana a miúdo como unha ferramenta para a mutaxénese. Primeiro, unha segunda copia dun xene dunha soa copia transfórmase no xenoma. O fungo debe despois reproducirse e pasar ao seu ciclo sexual para activar a maquinaria RIP. Obtéñense moitas mutacións no xene duplicado a partir dun só evento de fertilización para que se obteñan alelos inactivados, xeralmente debido a mutacións sen sentido, e tamén poden obterse alelos que conteñen mutacións de cambio de sentido.[36]
Notas
editar- ↑ "Point Mutation". Biology Dictionary. Consultado o 17 May 2019.
- ↑ Freese, Ernst (April 1959). "The difference between spontaneous and base-analogue induced mutations of phage T4". Proc. Natl. Acad. Sci. U.S.A. 45 (4): 622–33. Bibcode:1959PNAS...45..622F. PMC 222607. PMID 16590424. doi:10.1073/pnas.45.4.622.
- ↑ Freese, Ernst (1959). "The Specific Mutagenic Effect of Base Analogues on Phage T4". J. Mol. Biol. 1 (2): 87–105. doi:10.1016/S0022-2836(59)80038-3.
- ↑ 4,0 4,1 4,2 4,3 "Genetics Primer". Arquivado dende o orixinal o 26 de marzo de 2011. Consultado o 18 de abril de 2020.
- ↑ Davies H, Bignell GR, Cox C, et al. (June 2002). "Mutations of the BRAF gene in human cancer" (PDF). Nature 417 (6892): 949–54. Bibcode:2002Natur.417..949D. PMID 12068308. doi:10.1038/nature00766.
- ↑ Hoeijmakers JH (May 2001). "Genome maintenance mechanisms for preventing cancer". Nature 411 (6835): 366–74. Bibcode:2001Natur.411..366H. PMID 11357144. doi:10.1038/35077232.
- ↑ Li, Minghui; Goncearenco, Alexander; Panchenko, Anna R. (2017). Annotating Mutational Effects on Proteins and Protein Interactions: Designing Novel and Revisiting Existing Protocols. Methods in Molecular Biology 1550. pp. 235–260. ISBN 978-1-4939-6745-2. ISSN 1940-6029. PMC 5388446. PMID 28188534. doi:10.1007/978-1-4939-6747-6_17.
- ↑ "A Shortcut to Personalized Medicine". Genetic Engineering & Biotechnology News. 18 June 2008.
- ↑ Minde DP, Anvarian Z, Rüdiger SG, Maurice MM (2011). "Messing up disorder: how do missense mutations in the tumor suppressor protein APC lead to cancer?". Mol. Cancer 10: 101. PMC 3170638. PMID 21859464. doi:10.1186/1476-4598-10-101.
- ↑ Minde DP, Maurice MM, Rüdiger SG (2012). "Determining biophysical protein stability in lysates by a fast proteolysis assay, FASTpp". PLOS One 7 (10): e46147. Bibcode:2012PLoSO...746147M. PMC 3463568. PMID 23056252. doi:10.1371/journal.pone.0046147.
- ↑ Serra, E; Ars, E; Ravella, A; Sánchez, A; Puig, S; Rosenbaum, T; Estivill, X; Lázaro, C (2001). "Somatic NF1 mutational spectrum in benign neurofibromas: MRNA splice defects are common among point mutations". Human Genetics 108 (5): 416–29. PMID 11409870. doi:10.1007/s004390100514.
- ↑ Wiest, V; Eisenbarth, I; Schmegner, C; Krone, W; Assum, G (2003). "Somatic NF1 mutation spectra in a family with neurofibromatosis type 1: Toward a theory of genetic modifiers". Human Mutation 22 (6): 423–7. PMID 14635100. doi:10.1002/humu.10272.
- ↑ Mohyuddin, A; Neary, W. J.; Wallace, A; Wu, C. L.; Purcell, S; Reid, H; Ramsden, R. T.; Read, A; Black, G; Evans, D. G. (2002). "Molecular genetic analysis of the NF2 gene in young patients with unilateral vestibular schwannomas". Journal of Medical Genetics 39 (5): 315–22. PMC 1735110. PMID 12011146. doi:10.1136/jmg.39.5.315.
- ↑ Hsia CC (January 1998). "Respiratory function of hemoglobin". N. Engl. J. Med. 338 (4): 239–47. PMID 9435331. doi:10.1056/NEJM199801223380407.
- ↑ "HBB — Hemoglobin, Beta". Genetics Home Reference. National Library of Medicine.
- ↑ "Anemia, Sickle Cell". Genes and Disease. Bethesda MD: National Center for Biotechnology Information. 1998. NBK22183.
- ↑ Clancy S (2008). "Genetic Mutation". Nature Education 1 (1): 187.
- ↑ Genes and Disease. National Center for Biotechnology Information (US). 29 September 1998 – vía PubMed.
- ↑ eMedTV. "Causes of Tay-Sachs". Arquivado dende o orixinal o 06 de agosto de 2020. Consultado o 18 de abril de 2020.
- ↑ Ruder K (28 May 2004). "How Gene Mutations Cause Colorblindness". Genome News Network.
- ↑ Clutterbuck AJ (2011). "Genomic evidence of repeat-induced point mutation (RIP) in filamentous ascomycetes.". Fungal Genet Biol 48 (3): 306–26. PMID 20854921. doi:10.1016/j.fgb.2010.09.002.
- ↑ 22,0 22,1 22,2 Selker EU, Cambareri EB, Jensen BC, Haack KR (December 1987). "Rearrangement of duplicated DNA in specialized cells of Neurospora". Cell 51 (5): 741–752. PMID 2960455. doi:10.1016/0092-8674(87)90097-3.
- ↑ Graïa F, Lespinet O, Rimbault B, Dequard-Chablat M, Coppin E, Picard M (May 2001). "Genome quality control: RIP (repeat-induced point mutation) comes to Podospora". Mol Microbiol 40 (3): 586–595. PMID 11359565. doi:10.1046/j.1365-2958.2001.02367.x.
- ↑ Ikeda K, Nakayashiki H, Kataoka T, Tamba H, Hashimoto Y, Tosa Y, Mayama S (September 2002). "Repeat-induced point mutation (RIP) in Magnaporthe grisea: implications for its sexual cycle in the natural field context". Mol Microbiol 45 (5): 1355–1364. PMID 12207702. doi:10.1046/j.1365-2958.2002.03101.x.
- ↑ Idnurm A, Howlett BJ (June 2003). "Analysis of loss of pathogenicity mutants reveals that repeat-induced point mutations can occur in the Dothideomycete Leptosphaeria maculans". Fungal Genet Biol 39 (1): 31–37. PMID 12742061. doi:10.1016/S1087-1845(02)00588-1.
- ↑ Cuomo CA, Güldener U, Xu JR, Trail F, Turgeon BG, Di Pietro A, Walton JD, Ma LJ, et al. (September 2007). "The Fusarium graminearum genome reveals a link between localized polymorphism and pathogen specialization". Science 317 (5843): 1400–2. Bibcode:2007Sci...317.1400C. PMID 17823352. doi:10.1126/science.1143708.
- ↑ Coleman JJ, Rounsley SD, Rodriguez-Carres M, Kuo A, Wasmann CC, Grimwood J, Schmutz J, et al. (August 2009). "The genome of Nectria haematococca: contribution of supernumerary chromosomes to gene expansion". PLOS Genet 5 (8): e1000618. PMC 2725324. PMID 19714214. doi:10.1371/journal.pgen.1000618.
- ↑ Van de Wouw AP, Cozijnsen AJ, Hane JK, et al. (2010). "Evolution of linked avirulence effectors in Leptosphaeria maculans is affected by genomic environment and exposure to resistance genes in host plants". PLOS Pathog. 6 (11): e1001180. PMC 2973834. PMID 21079787. doi:10.1371/journal.ppat.1001180.
- ↑ Rep M, Kistler HC (August 2010). "The genomic organization of plant pathogenicity in Fusarium species". Curr. Opin. Plant Biol. 13 (4): 420–6. PMID 20471307. doi:10.1016/j.pbi.2010.04.004. Arquivado dende o orixinal o 02 de xuño de 2020. Consultado o 18 de abril de 2020.
- ↑ Farman ML (August 2007). "Telomeres in the rice blast fungus Magnaporthe oryzae: the world of the end as we know it". FEMS Microbiol. Lett. 273 (2): 125–32. PMID 17610516. doi:10.1111/j.1574-6968.2007.00812.x.
- ↑ Fudal I, Ross S, Brun H, et al. (August 2009). "Repeat-induced point mutation (RIP) as an alternative mechanism of evolution toward virulence in Leptosphaeria maculans". Mol. Plant Microbe Interact. 22 (8): 932–41. PMID 19589069. doi:10.1094/MPMI-22-8-0932.
- ↑ Irelan JT, Hagemann AT, Selker EU (December 1994). "High frequency repeat-induced point mutation (RIP) is not associated with efficient recombination in Neurospora". Genetics 138 (4): 1093–103. PMC 1206250. PMID 7896093.
- ↑ Freitag M, Williams RL, Kothe GO, Selker EU (2002). "A cytosine methyltransferase homologue is essential for repeat-induced point mutation in Neurospora crassa". Proc Natl Acad Sci U S A 99 (13): 8802–7. Bibcode:2002PNAS...99.8802F. PMC 124379. PMID 12072568. doi:10.1073/pnas.132212899.
- ↑ Lee DW, Freitag M, Selker EU, Aramayo R (2008). "A cytosine methyltransferase homologue is essential for sexual development in Aspergillus nidulans.". PLOS One 3 (6): e2531. Bibcode:2008PLoSO...3.2531L. PMC 2432034. PMID 18575630. doi:10.1371/journal.pone.0002531.
- ↑ Malagnac F, Wendel B, Goyon C, Faugeron G, Zickler D, Rossignol JL, et al. (1997). "A gene essential for de novo methylation and development in Ascobolus reveals a novel type of eukaryotic DNA methyltransferase structure.". Cell 91 (2): 281–90. PMID 9346245. doi:10.1016/S0092-8674(00)80410-9.
- ↑ Selker EU (1990). "Premeiotic instability of repeated sequences in Neurospora crassa.". Annu Rev Genet 24: 579–613. PMID 2150906. doi:10.1146/annurev.ge.24.120190.003051.
Véxase tamén
editarOutros artigos
editarLigazóns externas
editar| Wikimedia Commons ten máis contidos multimedia na categoría: Mutación puntual |
- Point Mutation Medical Subject Headings (MeSH) na Biblioteca Nacional de Medicina dos EUA.