Glicosa-6-fosfato deshidroxenase
A glicosa-6-fosfato deshidroxenase (G6PD ou G6PDH) (EC 1.1.1.49) é un encima citosólico que cataliza a seguinte reacción química na que se produce NADP+:
| Glicosa-6-fosfato deshidroxenase | |||||||||
|---|---|---|---|---|---|---|---|---|---|
 glicosa 6-fosfato deshidroxenase de Leuconostoc mesenteroides | |||||||||
| Identificadores | |||||||||
| Símbolo | G6PD_N | ||||||||
| Pfam | PF00479 | ||||||||
| Pfam clan | CL0063 | ||||||||
| InterPro | IPR022674 | ||||||||
| PROSITE | PDOC00067 | ||||||||
| SCOPe | 1dpg / SUPFAM | ||||||||
| |||||||||
| Glicosa-6-fosfato deshidroxenase | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| Identificadores | |||||||||
| Número EC | 1.1.1.49 | ||||||||
| Número CAS | 9001-40-5 | ||||||||
| Bases de datos | |||||||||
| IntEnz | vista de IntEnz | ||||||||
| BRENDA | entrada de BRENDA | ||||||||
| ExPASy | vista de NiceZyme | ||||||||
| KEGG | entrada de KEGG | ||||||||
| MetaCyc | vía metabólica | ||||||||
| PRIAM | perfil | ||||||||
| Estruturas PDB | RCSB PDB PDBe PDBj PDBsum | ||||||||
| Gene Ontology | AmiGO / EGO | ||||||||
| |||||||||
- D-glicosa 6-fosfato + NADP+ 6-fosfo-D-glicono-1,5-lactona + NADPH + H+
Este encima participa na ruta da pentosa fosfato, unha ruta metabólica que fornece enerxía reducida ás células (como os eritrocitos) ao manter o nivel do coencima fosfato do dinucleótido de nicotinamida e adenina (NADPH). O NADPH, á súa vez, mantén o nivel de glutatión nesas células, o que, por exemplo, axuda a protexer os eritrocitos dos danos oxidativos causados por moléculas como o peróxido de hidróxeno.[1] De grande importancia cuantitativa é a produción de NADPH polos tecidos implicados na biosíntese de ácidos graxos ou isoprenoides, como os do fígado, glándulas mamarias, tecido adiposo e glándulas adrenais. A G6PD reduce o NADP+ a NADPH á vez que oxida a glicosa 6-fosfato.[2]
Clinicamente, unha an deficiencia xenética de G6PD ligada ao cromosoma X predispón as persoas á anemia hemolítica non inmune.[3]
Distribución nas especies
editarA G6PD está amplamente distribuída en moitas especies, desde bacterias a humanos. Os aliñamentos múltiples de secuencias dunhas 100 G6PD coñecidas de diferentes organismos revelan unha identidade de secuencia que vai do 30% ao 94%.[4] A G6PD humana ten un 30% de identidade na secuencia de aminoácidos con secuencias de G6PD doutras especies.[5] Os humanos tamén teñen dúas isoformas dun só xene que codifica a G6PD.[6] Ademais, documentáronse 150 G6PD mutantes humanas.[4] Estas mutacións son principalmente mutacións de cambio de sentido que teñen como resultado substitucións de aminoácidos,[7] e mentres que algunhas delas causan deficiencia de G6PD, outras non parecen orixinar ningunha diferenza funcional salientable.[7] Algúns científicos propuxeron que algunhas das variacións xenéticas na G6PD humana son o resultado de xeracións de adaptación á infección da malaria.[8]
Outras especies tamén experimentan unha variación na G6PD. En plantas superiores, informouse do descubrimento de varias isoformas de G6PD, que están localizadas no citosol, no estroma plastidial, e nos peroxisomas.[9] Unha G6PD modificada dependente de F420 (oposta á dependente de NADP+) encóntrase en Mycobacterium tuberculosis e é interesante para o tratamento da tuberculose.[10] Unha G6PD bacteriana atopada en Leuconostoc mesenteroides é reactiva ao 4-hidroxinonenal, ademais de á G6P.[11]
Estrutura encimática
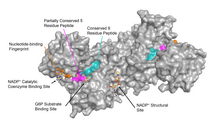
editarA G6PD encóntrase xeralmente en forma de dímero de dous monómeros idénticos.[7] Dependendo das condicións, como o pH, estes dímeros poden dimerizarse e formar tetrámeros.[5] Cada monómero do complexo ten un sitio de unión para o substrato, no que se une a G6P, e un sitio de unión para o coencima catalítico, que se une ao NADP+/NADPH usando o pregamento de Rossmann.[4] Para algúns organismos superiores, como os humanos, a G6PD contén un sitio de unión adicional para o NADP+, chamado o sitio estrutural para o NADP+, que non parece participar directamente na reacción catalizada pola G6PD. O propósito evolutivo do sitio estrutural para o NADP+ non se coñece.[4] En canto ao seu tamaño, cada monómero é de aproximadamente 500 aminoácidos de longo (514 aminoácidos en humanos[5]).
A conservación funcional e estrutural entre a G6PD humana e a de Leuconostoc mesenteroides indica que hai 3 rexións amplamente conservadas no encima, que son: un péptido de 9 residuos no sitio de unión ao substrato, coa secuencia RIDHYLGKE (residuos 198-206 na G6PD humana), unha pegada dactilar de unión a nucleótidos, GxxGDLA (residuos 38-44 na G6PD humana), e unha secuencia parcialmente conservada EKPxG preto do sitio de unión ao substrato (residos 170-174 na G6PD humana), onde utilizamos as abreviaturas dunha letra dos aminoácidos e "x" significa un aminoácido variable.[4] A estrutura cristalina da G6PD revela unha ampla rede de interaccións electrostáticas e enlaces de hidróxeno que implican a G6P, 3 moléculas de auga, 3 lisinas, 1 arxinina, 2 histidinas, 2 ácidos glutámicos e outros aminoácidos polares.
A prolina na posición 172 pénsase que desempeña un papel crucial en posicionar correctamente a Lys171 con respecto ao substrato G6P. Nas dúas estruturas cristalinas coñecidas da G6PD humana normal, a Pro172 vese exclusivamente na conformación cis, mentres que na estrutura cristalina dun mutante causante dunha enfermidade (variante Canton R459L), a Pro172 vese case exclusivamente na conformación trans.[4]
Unha vez que se dispuxo das estruturas cristalinas, algúns científicos trataron de elaborar modelos das estruturas doutros mutantes. Por exemplo, en estudos de antepasados alemáns, nos que a encimopatía debido á deficiencia de G6PD é rara, os sitios de mutación na G6PD estaban preto do sitio de unión ao NADP+, no sitio de unión á G6P, e preto da interface entre os dous monómeros. Así, as mutacións nestas áreas críticas son posibles sen alterar completamente a función da G6PD.[7] De feito, a maioría das mutacións causantes de doenzas da G6PD aparecen preto do sitio estrutural para o NADP+.[12]
Sitio estrutural para o NADP+
editarO sitio estrutural para o NADP+ está localizado a máis de 20 Å do sitio de unión ao substrato e o sitio de unión para o coencima catalítico NADP+. O seu propósito na reacción catalizada polo encima non estivo claro durante moitos anos. Durante algún tempo pensouse que o NADP+ unido ao sitio estrutural era necesario para a dimerización dos monómeros do encima. Porén, despois viuse que isto era incorrecto.[12] Por outra parte, demostrouse que a presenza do NADP+ no sitio estrutural promove a dimerización de dímeros para formar os tetrámeros do encima.[12] Tamén se pensaba que o estado tetramérico era necesario para a actividade catalítica, pero isto tamén era falso.[12] Un dato interesante é que o sitio estrutural para o NADP+ é bastante diferente do sitio de unión para o coencima catalítico NADP+, e non contén a pegada dactilar de unión a nucleótidos.
O sitio estrutural unido ao NADP+ posúe interaccións favorables que o manteñen firmemente unido. En concreto, hai unha forte rede de enlaces de hidróxeno con cargas electrostáticas que difunden a través de moitos átomos por medio de enlaces de hidróxeno con 4 moléculas de auga (ver figura). Ademais, hai un conxunto extremadamente forte de interaccións hidrofóbicas de stacking que orixinan sistemas π solapados.
O sitio estrutural demostrouse que é importante para manter a estabilidade a longo prazo do encima.[12] Máis de 40 mutacións de clase I graves implican mutacións preto do sitio estrutural, afectando así á estabilidade a longo prazo destes encimas no corpo, o que finalmente resulta na deficiencia de G6PD.[12] Por exemplo, dúas mutacións de clase I graves, a G488S e a G488V, causan un drástico incremento da constante de disociación entre o NADP+ e o sitio estrutural, multiplicándoa por de 7 a 13. Coa proximidade do residuo 488 á Arg487, pénsase que unha mutación na posición 488 podería afectar ao posicionamento de Arg487 en relación ao NADP+,[12] e así altera a unión.
Regulación
editarA G6PD converte a G6P en 6-fosfoglicono-δ-lactona e é o e paso limitante encimático da ruta da pentosa fosfato. Así, a regulación da G6PD ten consecuencias augas abaixo da ruta para a actividade do resto da ruta da pentosa fosfato.
A glicosa-6-fosfato deshidroxenase é estimulada polo seu substrato, a G6P. A proporción habitual de NADPH/NADP+ no citosol de tecidos moi implicados nas biosínteses é de aproximadamente 100/1. O aumento da utilización do NADPH para a biosíntese de ácidos graxos incrementa moitísimo o nivel de NADP+, estimulando así a G6PD para producir máis NADPH.
A G6PD está negativamente regulada pola acetilación na lisina 403 (Lys403 ou K403), un residuo evolutivamente conservado. A G6PD acetilada na Lys403 non pode formar dímeros activos e mostrar unha completa perda de actividade. A acetilación da Lys304 impide estericamente que o NADP+ entre no sitio estrutural do NADP+, o cal reduce a estabilidade do encima. As células perciben estímulos oxidativos extracelulares para facer diminuír a acetilación da G6PD de maneira dependente de SIRT2. En ratos a desacetilación mediada por SIRT2 e a activación da G6PD estimula a vía da pentosa fosfato para subministrar NADPH citosólico para contrarrrestar os danos oxidativos e protexer os eritrocitos desa especie.[13]
A regulación pode tamén ocorrer por vías xenéticas. A isoforma, G6PDH, é regulada por factores de transcrición e postranscrición.[14] Ademais, a G6PD é un dos varios encimas glicolíticos activados polo factor de transcrición factor inducible pola hipoxia 1 (HIF1).[15]
Importancia clínica
editarA G6PD é salientable pola súa diversidade xenética. Describíronse moitas variantes da G6PD, principalmente producidas a partir de mutacións de cambio de sentido cunha ampla gama de niveis de actividade encimática e asociadas con síntomas clínicos. Atopáronse dúas variantes de transcrición que codifican diferentes isoformas deste xene.[16]
A deficiencia de glicosa-6-fosfato deshidoxenase é moi común en todo o mundo, e causa unha anemia hemolítica aguda en presenza de infeccións simples, inxestión de feixóns ou reaccións con certas medicinas, antibióticos, antipiréticos, e antimaláricos.[3]
O crecemento e proliferación celular vense afectados pola G6PD.[17] Están investigándose inhibidores da G6PD para tratar o cancro e outras condicións.[15] In vitro un ensaio de proliferación de células indica que os inhibidores da G6PD DHEA (deshidroepiandrosterona) e ANAD (6-aminonicotinamida), fan decrecer o crecemento de liñas celulares AML.[17][18] A G6PD é hipometilatda na Lys403 na leucemia mieloide aguda, o SIRT2 activa a G6PD para aumentar a produción de NADPH e promover a proliferación das células da leucemia.[18]
Notas
editar- ↑ Thomas D, Cherest H, Surdin-Kerjan Y (Mar 1991). "Identification of the structural gene for glucose-6-phosphate dehydrogenase in yeast. Inactivation leads to a nutritional requirement for organic sulfur". The EMBO Journal 10 (3): 547–53. PMC 452682. PMID 2001672.
- ↑ Aster J, Kumar V, Robbins SL, Abbas AK, Fausto N, Cotran RS (2010). Robbins and Cotran Pathologic Basis of Disease. Saunders/Elsevier. pp. Kindle Locations 33340–33341. ISBN 1-4160-3121-9.
- ↑ 3,0 3,1 Cappellini MD, Fiorelli G (Jan 2008). "Glucose-6-phosphate dehydrogenase deficiency". Lancet 371 (9606): 64–74. PMID 18177777. doi:10.1016/S0140-6736(08)60073-2.
- ↑ 4,0 4,1 4,2 4,3 4,4 4,5 Kotaka M, Gover S, Vandeputte-Rutten L, Au SW, Lam VM, Adams MJ (May 2005). "Structural studies of glucose-6-phosphate and NADP+ binding to human glucose-6-phosphate dehydrogenase". Acta Crystallographica Section D (en inglés) 61 (Pt 5): 495–504. PMID 15858258. doi:10.1107/S0907444905002350.
- ↑ 5,0 5,1 5,2 Au SW, Gover S, Lam VM, Adams MJ (Mar 2000). "Human glucose-6-phosphate dehydrogenase: the crystal structure reveals a structural NADP(+) molecule and provides insights into enzyme deficiency". Structure 8 (3): 293–303. PMID 10745013. doi:10.1016/S0969-2126(00)00104-0.
- ↑ "G6PD glucose-6-phosphate dehydrogenase [ Homo sapiens (human) ]". NCBI. Consultado o 13 December 2015.
- ↑ 7,0 7,1 7,2 7,3 Kiani F, Schwarzl S, Fischer S, Efferth T. "Three-dimensional modeling of glucose-6-phosphate dehydrogenase-deficient variants from German ancestry". PLoS One 2 (7): e625. PMC 1913203. PMID 17637841. doi:10.1371/journal.pone.0000625.
- ↑ Luzzatto L, Bienzle U (Jun 1979). "The malaria/G.-6-P.D. hypothesis". Lancet 1 (8127): 1183–4. PMID 86896. doi:10.1016/S0140-6736(79)91857-9.
- ↑ Corpas FJ, Barroso JB, Sandalio LM, Distefano S, Palma JM, Lupiáñez JA, Del Río LA (Mar 1998). "A dehydrogenase-mediated recycling system of NADPH in plant peroxisomes". The Biochemical Journal 330 (Pt 2): 777–84. PMC 1219205. PMID 9480890. doi:10.1042/bj3300777.
- ↑ Bashiri G, Squire CJ, Moreland NJ, Baker EN (Jun 2008). "Crystal structures of F420-dependent glucose-6-phosphate dehydrogenase FGD1 involved in the activation of the anti-tuberculosis drug candidate PA-824 reveal the basis of coenzyme and substrate binding". The Journal of Biological Chemistry (en inglés) 283 (25): 17531–41. PMID 18434308. doi:10.1074/jbc.M801854200.
- ↑ Szweda LI, Uchida K, Tsai L, Stadtman ER (Feb 1993). "Inactivation of glucose-6-phosphate dehydrogenase by 4-hydroxy-2-nonenal. Selective modification of an active-site lysine". The Journal of Biological Chemistry (en inglés) 268 (5): 3342–7. PMID 8429010.
- ↑ 12,0 12,1 12,2 12,3 12,4 12,5 12,6 Wang XT, Chan TF, Lam VM, Engel PC (Aug 2008). "What is the role of the second "structural" NADP+-binding site in human glucose 6-phosphate dehydrogenase?". Protein Science 17 (8): 1403–11. PMC 2492815. PMID 18493020. doi:10.1110/ps.035352.108.
- ↑ Wang YP, Zhou LS, Zhao YZ, Wang SW, Chen LL, Liu LX, Ling ZQ, Hu FJ, Sun YP, Zhang JY, Yang C, Yang Y, Xiong Y, Guan KL, Ye D (Jun 2014). "Regulation of G6PD acetylation by SIRT2 and KAT9 modulates NADPH homeostasis and cell survival during oxidative stress". The EMBO Journal 33 (12): 1304–20. PMC 4194121. PMID 24769394. doi:10.1002/embj.201387224.
- ↑ Kletzien RF, Harris PK, Foellmi LA (Feb 1994). "Glucose-6-phosphate dehydrogenase: a "housekeeping" enzyme subject to tissue-specific regulation by hormones, nutrients, and oxidant stress". FASEB Journal (en inglés) 8 (2): 174–81. PMID 8119488.
- ↑ 15,0 15,1 de Lartigue J (2012-06-12). "Cancer Research Moves Beyond the Original Hallmarks of Cancer". OncLive. Arquivado dende o orixinal o 02 de xaneiro de 2018. Consultado o 01 de xaneiro de 2018.
- ↑ "Entrez Gene: G6PD glucose-6-phosphate dehydrogenase".
- ↑ 17,0 17,1 Tian WN, Braunstein LD, Pang J, Stuhlmeier KM, Xi QC, Tian X, Stanton RC (Apr 1998). "Importance of glucose-6-phosphate dehydrogenase activity for cell growth". The Journal of Biological Chemistry 273 (17): 10609–17. PMID 9553122. doi:10.1074/jbc.273.17.10609.
- ↑ 18,0 18,1 Xu SN, Wang TS, Li X, Wang YP (Sep 2016). "SIRT2 activates G6PD to enhance NADPH production and promote leukaemia cell proliferation". Sci Rep 6: 32734. PMC 5009355. PMID 27586085. doi:10.1038/srep32734.
Véxase tamén
editarOutros artigos
editarBibliografía
editar- Vulliamy T, Beutler E, Luzzatto L (1993). "Variants of glucose-6-phosphate dehydrogenase are due to missense mutations spread throughout the coding region of the gene". Human Mutation 2 (3): 159–67. PMID 8364584. doi:10.1002/humu.1380020302.
- Mason PJ (Sep 1996). "New insights into G6PD deficiency". British Journal of Haematology 94 (4): 585–91. PMID 8826878.
- Wajcman H, Galactéros F (Aug 2004). "[Glucose 6-phosphate dehydrogenase deficiency: a protection against malaria and a risk for hemolytic accidents]". Comptes Rendus Biologies (en French) 327 (8): 711–20. PMID 15506519. doi:10.1016/j.crvi.2004.07.010.
