Mutaxénese (técnica de bioloxía molecular)
En bioloxía molecular, a mutaxénese é unha importante técnica de laboratorio na que se producen por enxeñaría xenética deliberadamente mutacións para producir bibliotecas de xenes mutantes, proteínas, cepas de bacterias ou outros organismos modificados xeneticamente. Os diversos constituíntes dun xene, así como os seus elementos regulatorios e os seus produtos xénicos, poden ser mutados para que o funcionamento dun locus xénico, proceso ou produto poida ser examinado en detalle. A mutación pode producir proteínas mutantes con interesantes propiedades ou con mellores ou novas funcións que poden ter uso comercial. As cepas mutantes tamén se poden producir para que teñan aplicacións prácticas ou permitan as bases moleculares dunha determinada función celular que se quere investigar.
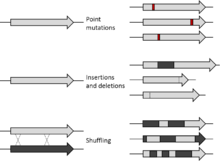
Hoxe en día existen moitos métodos de mutaxénese. Inicialmente, o tipo de mutacións inducidas artificialmente en laboratorio eran completamente aleatorias usando para iso mecanismos como a irradiación UV. Esta mutaxénese ao chou non pode dirixirse a rexións ou secuencias específicas do xenoma; porén, co desenvolvemento da mutaxénese dirixida a sitio, poden facerse cambios máis específicos. Desde 2013, o desenvolvemento da tecnoloxía CRISPR/Cas9, baseada nun sistema de defensa contra virus dos procariotas, permitiu a "edición" (modificación) ou mutaxénese dun xenoma in vivo.[1] A mutaxénese dirixida a sitio probou a súa utilidade en situacións nas que a mutaxénese ao chou non o é. Outras técnicas de mutaxénese son a mutaxénese combinatoria e a insercional. A mutaxénese que non é aleatoria pode utilizarse para clonar ADN,[2] investigar os efectos de mutáxenos,[3] e proteínas modificadas por enxeñaría.[4] Tamén ten aplicacións médicas como axudar a pacientes inmunocomprometidos, investigar o tratamento de doenzas incluíndo a infección por VIH e os cancros, e a curación de doenzas como a beta-talasemia.[5]
Mutaxénese aleatoria
editarAs primeiras aproximacións á mutaxénese confiaban en métodos que producían mutacións completamente aleatorias, ao chou. En tales métodos as células ou organismos expóñense a mutáxenos como a radiación UV ou compostos químicos mutaxénicos e os mutantes coas características desexadas selecciónanse despois. Hermann Muller descubriu en 1927 que os raios X poden causar mutacións xenéticas en moscas do vinagre,[6] e continuou usando os mutantes que creara nos seus estudos de xenética.[7] No caso de Escherichia coli, os mutantes poden seleccionarse primeiro expoñéndoos a radiación UV, despois cultivándoos en placas de ágar. As colonias formadas son despois replicadas en placas de réplica, unha nun medio rico, outra nun medio mínimo e os mutantes que teñen necesidades nutricionais especiais poden entón identificarse pola súa incapacidade de crecer no medio mínimo. Procedementos similares poden repetirse con outros tipos de células e con diferentes medios para a selección.
Máis tarde desenvolvéronse varios métodos para xerar mutacións ao chou en proteínas específicas para cribar buscando os mutantes con propiedades interesantes ou melloradas. Estes métodos poden implicar o uso de nucleótidos dopados na síntese de oligonucleótidos, ou realizar unha PCR en condicións que favorecen a incorporación incorrecta de nucleótidos (PCR tendente ao erro), por exemplo ao reducir a fidelidade de replicación ou usar análogos de nucleótidos.[8] Unha variación deste método para integrar mutacións non nesgadas nun xene é a mutaxénese de saturación de secuencias.[9] Os produtos de PCR que conteñen mutación(s) son despois clonados nun vector de expresión e as proteínas mutantes producidas poden entón caracterizarse.
En estudos en animais de laboratorio, utilizáronse axentes alquilantes como a N-etil-N-nitrosourea (ENU) para xerar ratos mutantes.[10][11] Tamén se usa a miúdo o metanosulfonato de etilo (EMS) para xerar mutantes de animais, plantas e virus.[12][13][14]
Nunha lei da Unión Europea (directriz 2001/18), este tipo de mutaxénese pode utilizarse para producir organismos modificados xeneticamente, pero os produtos están exentos de regulación: non levan etiqueta nin avaliación.[15]
Mutaxénese dirixida a sitio
editar- Artigo principal: Mutaxénese dirixida a sitio.
Antes do desenvolvemento das técnicas da mutaxénese dirixida a sitio, todas as mutacións eran aleatorias e os científicos tiñan que usar a selección para o fenotipo desexado e atopar a mutación buscada. As técnicas de mutaxénese aleatoria teñen a vantaxe do modo en que se poden producir moitas mutacións; porén, aínda que a mutaxénese aleatoria pode producir un cambio nun só nucleótido, non ofrece moito control sobre cal é o nucleótido que é cambiado.[5] Moitos investigadores, por tanto, buscaban introducir cambios seleccionados no ADN de forma precisa e específica de sitio. Os intentos iniciais usaron análogos de nucleótidos e outros compostos químicos para xerar mutacións puntuais localizadas.[16] Entre tales compostos químicos estaba a aminopurina, que induce unha transición de AT a GC,[17] mentres que a nitrosoguanidina,[18] o bisulfito,[19] e a N4-hidroxicitidina pode inducir unha transición de GC a AT.[20][21] Estas técnicas permiten que se preparen por enxeñaría mutacións específicas nunha proteína; porén, non son flexibles con respecto aos tipos de mutantes xerados, nin son tan específicas coma os últimos métodos de mutaxénese dirixida a sitio e, por tanto, teñen certo grao de aleatoriedade. Outras tecnoloxías como o corte do ADN en sitios específicos do cromosoma, a adición de novos nucleótidos e o intercambio de pares de bases fixeron posible decidir onde poden estar as mutacións.[11][8]
As técnicas actuais da mutación dirixida a sitio orixínáronse a partir da técnica de extensión de cebador (primer) desenvolvida en 1978. Tales técnicas normalmente implican o uso de oligonucleótidos mutaxénicos prefabricados nunha reacción de extensión de cebador con ADN polimerase. Estes métodos permiten mutacións puntuais ou delecións ou insercións de pequenos tramos de ADN en sitios específicos. Os avances en metodoloxía converteron agora esta mutaxénese nun proceso relativamente simple e eficiente.[3]
Métodos novos e máis eficientes de mutaxénese dirixida a sitio están en constante desenvolvemento. Por exemplo, unha técnica chamada SLiCE (do inglés "Seamless ligation cloning extract") permite a clonación de certas secuencias de ADN dentro do xenoma e pode inserirse máis dun fragmento de ADN no xenoma á vez.[2]
A mutaxénese dirixida a sitio permite investigar o efecto de mutacións específicas. Ten numerosos usos; por exemplo, foi utilizado para determinar o susceptibles que eran certas especies a compostos químicos que se utilizan decote nos laboratorios. O experimento utilizado usou a mutaxénese dirixida a sitio para imitar as mutacións esperadas do composto químico específico. A mutación tiña como resultado un cambio en aminoácidos específicos e analizáronse os efectos desta mutación.[3]
A estratexia dirixida a sitio pode facerse sistematicamente en técnicas como a mutaxénese de varrido de alanina, onde os residuos son mutados sistematicamente a alanina para identificar residuos importantes da estrutura ou función dunha proteína.[22] Outra estratexia completa é a mutaxénese de saturación de sitios, onde un codón ou un conxnto de codóns pode ser substituído por todos os posibles aminoácidos nas posicións específicas.[23][24]
Mutaxénese combinatoria
editarA mutaxénese combinatoria é unha técica de enxeñaría de proteínas dirixida a sitio na que múltiples mutantes dunha proteína poden ser sometidas a enxeñaría simultaneamente baseándose na análise dos efectos de mutacións individuais aditivas.[25] Proporciona un método útil para avaliar os efectos combinatorios dun gran número de mutacións sobre a función das proteínas.[26] Grandes cantidades de mutantes poden ser cribados buscando unha característica particular por análise combinatoria.[25] Nesta técnica poden modificarse exhaustivamente múltiples posicións ou curtas secuencias ao longo dunha febra de ADN para obter unha biblioteca completa de proteínas mutantes.[25] A taxa de incidencia de variantes beneficiosas pode ser mellorada por diferentes métodos para construír bibliotecas de mutaxénese. Unha estratexia desta técnica é extraer e substituír unha porción da secuencia de ADN por biblioteca de secuencias que conteñen todas as posibles combinacións no sitio de mutación desexado. O contido do segmento inserido pode incluír secuencias de importancia estrutural, propiedades inmunoxénicas ou funcións enerxéticas. Un segmento tamén pode ser inserido aleatoriamente nun xene para avaliar a importancia estrutural ou funcional dunha determinada parte dunha proteína.[25]
Mutaxénese insercional
editar- Artigos principais: Mutaxénese insercional, Mutaxénese etiquetada con sinatura e Mutaxénese de transposón.
A inserción dun ou máis pares de bases, que orixia mutacións no ADN, tamén se coñece como mutaxénese insercional.[27] As mutacións introducidas por enxeñaría como estas poden proporcionar información importante na investigación do cancro, esecialmente sobre a mecánica do desenvolvemento desta doenza. Os retrovirus e transposóns son as ferramentas instrumentais básicas na mutaxénese insercional. Os retrovirus, como o virus do tumor mamario do rato e o virus da leucemia murina, poden utilizarse para identificar xenes implicados na carcinoxénese e comprender as vías biolóxicas de cancros específicos.[28] Os transposóns, segmentos de ADN que poden sufrir transposición, poden deseñarse e aplicarse á mutaxénese insercional como instrumento para o descubrimento de xenes do cancro.[28] Estes segmentos permiten que a mutaxénese insercional se poida aplicar a virtualmente calquera tecido e permite unha secuenciación do ADN mellor, máis completa e sen nesgos.[28]
Os investigadores atoparon catro mecanismos de mutaxénese insercional que poden utilizarse en humanos. O primeiro mecanismo chámase inserción de amplificador (enhancer insertion). Os amplificadores ou enhancers promoven a transcrición dun determinado xene ao interaccionaren cun promotor dese xene. Este mecanismo particular foi utilizado primeiramente para axudar a pacientes gravemente inmunocomprometidos que necesitaban medula ósea. Os gammarretrovirus portan amplificadores que foron despois inseridos en pacientes. O segundo mecanismo denomínase inserción de promotor. Os promotores proporcionan ás células as secuencias específicas necesarias para enmpezar a tradución. A inserción de promotor axudou os investigadores a aprender máis sobre o virus VIH. O terceiro mecanismo é a inactivación de xenes. Un exemplo de inactivación de xenes é usar a mutaxénese insercional para inserir un retrovirus que altere o xenoma das células T en pacientes de leucemia e dálles un antíxeno específico chamado CAR que permite que as células T tomen como diana as células cancerosas. Os mecanismos finais denomínanse substitución de extremo 3' do ARNm. Os nosos xenes ocasionalmente sofren mutacións puntuais causando beta-talasemia, que interrompe a función dos glóbulos vermellos. Para arranxar este problema é introducida a secuencia xénica correcta para os glóbulos vermellos e faise unha substitución.[5]
Recombinación homóloga
editarA recombinación homóloga pode utilizarse para producir mutacións específicas nun organismo. Introdúcese na célula un vector que contén unha secuencia de ADN similar á do xene que vai ser modificado e por un proceso de recombinación substitúe o xene diana no cromosoma. Este método pode utilizarse para introducir unha mutación ou facer o knockout dun xene, por exemplo como o usado na produción dun rato knockout.[29]
CRISPR
editarDesde 2013 o desenvolvemento da tecnoloxía CRISPR-Cas9 permitiu a introdución eficiente de diferentes tipos de mutacións no xenoma dunha ampla variedade de organismos. O método non require un sitio de inserción de transposón, non deixa marcador e a súa eficiencia e simplicidade fíxoo o método preferido de "edición" do xenoma.[30][31]
Síntese de xenes
editarA medida que o custo da síntese de oligonucleótidos de ADN foi baixando, a síntese artificial dun xene completo converteuse finalmente nun método viable para introducir mutacións nun xene. Este método permite introducir mutacións en moitos sitios, incluíndo o redeseño completo do uso de codóns por un xene para optimizalo para un determinado organismo.[32]
Notas
editar- ↑ Hsu PD, Lander ES, Zhang F (xuño de 2014). "Development and applications of CRISPR-Cas9 for genome engineering". Cell 157 (6): 1262–78. PMC 4343198. PMID 24906146. doi:10.1016/j.cell.2014.05.010.
- ↑ 2,0 2,1 Motohashi K (xuño de 2015). "A simple and efficient seamless DNA cloning method using SLiCE from Escherichia coli laboratory strains and its application to SLiP site-directed mutagenesis". BMC Biotechnology 15: 47. PMC 4453199. PMID 26037246. doi:10.1186/s12896-015-0162-8.
- ↑ 3,0 3,1 3,2 Doering JA, Lee S, Kristiansen K, Evenseth L, Barron MG, Sylte I, LaLone CA (novembro de 2018). "In Silico Site-Directed Mutagenesis Informs Species-Specific Predictions of Chemical Susceptibility Derived From the Sequence Alignment to Predict Across Species Susceptibility (SeqAPASS) Tool". Toxicological Sciences 166 (1): 131–145. PMC 6390969. PMID 30060110. doi:10.1093/toxsci/kfy186.
- ↑ Choi GC, Zhou P, Yuen CT, Chan BK, Xu F, Bao S, et al. (agosto de 2019). "Combinatorial mutagenesis en masse optimizes the genome editing activities of SpCas9". Nature Methods 16 (8): 722–730. PMID 31308554. doi:10.1038/s41592-019-0473-0.
- ↑ 5,0 5,1 5,2 Bushman FD (febreiro de 2020). "Retroviral Insertional Mutagenesis in Humans: Evidence for Four Genetic Mechanisms Promoting Expansion of Cell Clones". Molecular Therapy (en inglés) 28 (2): 352–356. PMC 7001082. PMID 31951833. doi:10.1016/j.ymthe.2019.12.009.
- ↑ Muller HJ (xullo de 1927). "Artificial Transmutation of the Gene" (PDF). Science 66 (1699): 84–7. Bibcode:1927Sci....66...84M. PMID 17802387. doi:10.1126/science.66.1699.84.
- ↑ Crow JF, Abrahamson S (decembro de 1997). "Seventy years ago: mutation becomes experimental". Genetics 147 (4): 1491–6. PMC 1208325. PMID 9409815. doi:10.1093/genetics/147.4.1491.
- ↑ 8,0 8,1 Blackburn GM, ed. (2006). Nucleic Acids in Chemistry and Biology (3rd ed.). Royal Society of Chemistry. pp. 191–192. ISBN 978-0854046546.
- ↑ Wong TS, Tee KL, Hauer B, Schwaneberg U (febreiro de 2004). "Sequence saturation mutagenesis (SeSaM): a novel method for directed evolution". Nucleic Acids Research 32 (3): 26e–26. PMC 373423. PMID 14872057. doi:10.1093/nar/gnh028.
- ↑ Justice MJ, Noveroske JK, Weber JS, Zheng B, Bradley A (1999). "Mouse ENU mutagenesis". Human Molecular Genetics 8 (10): 1955–63. PMID 10469849. doi:10.1093/hmg/8.10.1955.
- ↑ 11,0 11,1 Hrabé de Angelis M, Balling R (May 1998). "Large scale ENU screens in the mouse: genetics meets genomics". Mutation Research 400 (1–2): 25–32. PMID 9685575. doi:10.1016/s0027-5107(98)00061-x.
- ↑ Flibotte S, Edgley ML, Chaudhry I, Taylor J, Neil SE, Rogula A, et al. (xuño de 2010). "Whole-genome profiling of mutagenesis in Caenorhabditis elegans". Genetics 185 (2): 431–41. PMC 2881127. PMID 20439774. doi:10.1534/genetics.110.116616.
- ↑ Bökel C (2008). EMS screens : from mutagenesis to screening and mapping. Methods in Molecular Biology 420. pp. 119–38. PMID 18641944. doi:10.1007/978-1-59745-583-1_7.
- ↑ Favor AH, Llanos CD, Youngblut MD, Bardales JA (2020). "Optimizing bacteriophage engineering through an accelerated evolution platform". Scientific Reports 10 (1): 13981. PMC 7438504. PMID 32814789. doi:10.1038/s41598-020-70841-1.
- ↑ Krinke C (March 2018). "GMO directive : the origins of the mutagenesis exemption". Inf'OGM.[Ligazón morta]
- ↑ Shortle D, DiMaio D, Nathans D (1981). "Directed mutagenesis". Annual Review of Genetics 15: 265–94. PMID 6279018. doi:10.1146/annurev.ge.15.120181.001405.
- ↑ Caras IW, MacInnes MA, Persing DH, Coffino P, Martin DW (setembro de 1982). "Mechanism of 2-aminopurine mutagenesis in mouse T-lymphosarcoma cells". Molecular and Cellular Biology 2 (9): 1096–103. PMC 369902. PMID 6983647. doi:10.1128/mcb.2.9.1096.
- ↑ McHugh GL, Miller CG (outubro de 1974). "Isolation and characterization of proline peptidase mutants of Salmonella typhimurium". Journal of Bacteriology 120 (1): 364–71. PMC 245771. PMID 4607625. doi:10.1128/JB.120.1.364-371.1974.
- ↑ Shortle D, Nathans D (maio de 1978). "Local mutagenesis: a method for generating viral mutants with base substitutions in preselected regions of the viral genome". Proceedings of the National Academy of Sciences of the United States of America 75 (5): 2170–4. Bibcode:1978PNAS...75.2170S. PMC 392513. PMID 209457. doi:10.1073/pnas.75.5.2170.
- ↑ Flavell RA, Sabo DL, Bandle EF, Weissmann C (xaneiro de 1975). "Site-directed mutagenesis: effect of an extracistronic mutation on the in vitro propagation of bacteriophage Qbeta RNA". Proceedings of the National Academy of Sciences of the United States of America 72 (1): 367–71. Bibcode:1975PNAS...72..367F. PMC 432306. PMID 47176. doi:10.1073/pnas.72.1.367.
- ↑ Müller W, Weber H, Meyer F, Weissmann C (setembro de 1978). "Site-directed mutagenesis in DNA: generation of point mutations in cloned beta globin complementary dna at the positions corresponding to amino acids 121 to 123". Journal of Molecular Biology 124 (2): 343–58. PMID 712841. doi:10.1016/0022-2836(78)90303-0.
- ↑ Vanessa E. Gray; Ronald J. Hause; Douglas M. Fowler (1 de setembro de 2017). "Analysis of Large-Scale Mutagenesis Data To Assess the Impact of Single Amino Acid Substitutions". Genetics 207 (1): 53–61. PMC 5586385. PMID 28751422. doi:10.1534/genetics.117.300064.
- ↑ Reetz, M. T.; Carballeira J. D. (2007). "Iterative saturation mutagenesis (ISM) for rapid directed evolution of functional enzymes". Nature Protocols 2 (4): 891–903. PMID 17446890. doi:10.1038/nprot.2007.72.
- ↑ Cerchione, Derek; Loveluck, Katherine; Tillotson, Eric L.; Harbinski, Fred; DaSilva, Jen; Kelley, Chase P.; Keston-Smith, Elise; Fernandez, Cecilia A.; Myer, Vic E.; Jayaram, Hariharan; Steinberg, Barrett E.; Xu, Shuang-yong (16 de aril de 2020). "SMOOT libraries and phage-induced directed evolution of Cas9 to engineer reduced off-target activity". PLOS ONE 15 (4): e0231716. Bibcode:2020PLoSO..1531716C. PMC 7161989. PMID 32298334. doi:10.1371/journal.pone.0231716.
- ↑ 25,0 25,1 25,2 25,3 Parker AS, Griswold KE, Bailey-Kellogg C (novembro de 2011). "Optimization of combinatorial mutagenesis". Journal of Computational Biology 18 (11): 1743–56. Bibcode:2011LNCS.6577..321P. PMC 5220575. PMID 21923411. doi:10.1089/cmb.2011.0152.
- ↑ Choi GC, Zhou P, Yuen CT, Chan BK, Xu F, Bao S, Chu HY, Thean D, Tan K, Wong KH, Zheng Z, Wong AS (agosto de 2019). "Combinatorial mutagenesis en masse optimizes the genome editing activities of SpCas9". Nature Methods 16 (8): 722–730. PMID 31308554. doi:10.1038/s41592-019-0473-0.
- ↑ Uren AG, Kool J, Berns A, van Lohuizen M (novembro de 2005). "Retroviral insertional mutagenesis: past, present and future". Oncogene 24 (52): 7656–72. PMID 16299527. doi:10.1038/sj.onc.1209043.
- ↑ 28,0 28,1 28,2 Vassiliou G, Rad R, Bradley A (2010-01-01). Wassarman PM, Soriano PM, eds. "The use of DNA transposons for cancer gene discovery in mice". Methods in Enzymology. Guide to Techniques in Mouse Development, Part B: Mouse Molecular Genetics (2nd ed.) (Academic Press) 477: 91–106. ISBN 9780123848802. PMID 20699138. doi:10.1016/s0076-6879(10)77006-3.
- ↑ "Homologous Recombination Method (and Knockout Mouse)". Davidson College.
- ↑ Damien Biot-Pelletier; Vincent J. J. Martin (2016). "Seamless site-directed mutagenesis of the Saccharomyces cerevisiae genome using CRISPR-Cas9". Journal of Biological Engineering 10: 6. PMC 4850645. PMID 27134651. doi:10.1186/s13036-016-0028-1.
- ↑ Xu S (20 de agosto de 2015). "The application of CRISPR-Cas9 genome editing in Caenorhabditis elegans". J Genet Genomics 42 (8): 413–21. PMC 4560834. PMID 26336798. doi:10.1016/j.jgg.2015.06.005.
- ↑ Khudyakov YE, Fields HA, eds. (25 de setembro de 2002). Artificial DNA: Methods and Applications. CRC Press. p. 13. ISBN 9781420040166.