DD-transpeptidase
A DD-transpeptidase (EC 3.4.16.4, chamada DD-peptidase, DD-transpeptidase, DD-carboxipeptidase, D-alanil-D-alanina carboxipeptidase, peptidase que corta a D-alanil-D-alanina, D-alanina carboxipeptidase, D-alanil carboxipeptidase e serina-tipo D-Ala-D-Ala carboxipeptidase.[1]) é un encima bacteriano que intervén na biosíntese da parede celular bacteriana, concretamente, a transpeptidación que establece enlaces cruzados entre as cadeas laterais peptídicas das febras de peptidoglicano.[2] Cataliza a transferencia do resto R-L-αα-D-alanil procedente de doantes de R-L-αα-D-alanil-D-alanina carbonilo ao γ-OH da serina do seu sitio activo e desde esta a un aceptor final.[3]
| Serina-tipo D-Ala-D-Ala carboxipeptidase | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| |||||||||
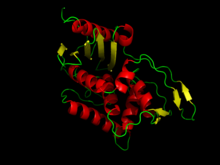
| Estrutura da DD-transpeptidase de Streptomyces K15 | |||||||||
| Identificadores | |||||||||
| Número EC | 3.4.16.4 | ||||||||
| Número CAS | 9077-67-2 | ||||||||
| Bases de datos | |||||||||
| IntEnz | vista de IntEnz | ||||||||
| BRENDA | entrada de BRENDA | ||||||||
| ExPASy | vista de NiceZyme | ||||||||
| KEGG | entrada de KEGG | ||||||||
| MetaCyc | vía metabólica | ||||||||
| PRIAM | perfil | ||||||||
| Estruturas PDB | RCSB PDB PDBe PDBj PDBsum | ||||||||
| |||||||||
O antibiótico penicilina únese irreversiblemente inhibe a actividade deste encima transpeptidase formando un intermediario moi estable peniciloil-encima, polo que a bacteria non pode formar correctamente a parede.[4] Debido á interacción entre a penicilina e a transpeptidase, este encima tamén é unha proteína que se une á penicilina (PBP).
Mecanismo
editarA DD-transpeptidase ten un mecanismo similar ao das reaccións proteolíticas das proteínααas da familia da tripsina.[5]
Os enlaces cruzados dos restos peptidil da febras de glicano adxacentes créanse nunha reacción de dous pasos. O primeiro paso implica o corte do enlace D-alanil-D-alanina dunha unidade péptidica precursora que actúa como doante de carbonilos, a liberación da D-alanina carboxilo terminal e a formación do acil-encima. O segundo paso implica a rotura do intermediario acil-encima e a fomación dun novo enlace peptídico entre o carbonilo do resto D-alanil e o grupo amino doutra unidade peptídica.[6]
A maioría dos puntos discutidos do mecanismo da DD-peptidase scéntranse na catálise de transferencia de protóns. Durante a formación do intermediario acil-encima, debe extraerse un protón do grupo hidroxilo da serina do sitio activo e debe engadirse outro ao grupo saínte amino. Un movemento de protóns similar debe ser facilitado na desacilación. Aínda non se dilucidou a identidade da catálise xeral ácido-básica implicada nestas transferencias de protóns.[7] Porén, propuxéronse tamén a tríades catalíticas tirosina, lisina e serina, así como serina, lisina, serina.[7]
Estrutura
editarAs transpeptidases son membros da superfamilia da peniciloil-serina transferase, que ten unha sinatura co motivo SxxK conservado,[8] onde "x" denota un residuo de aminoácido variable. As transpeptidases desta superfamilia mostran unha tendencia a presentar á vez tres motivos: SxxK, SxN (ou análogo) e KTG (ou análogo). Estes motivos aparecen en sitios equivalentes, e están aproximadamente espazados á mesma distancia, ao longo da cadea polipeptídica. Na proteína pregada estes motivos quedan situados uns preto dos outros no centro catalítico entre un dominio todo α e un dominio α/β.[9][10]
A estrutura da transpeptidase de Streptomyces K15 foi moi estudada e consta dunha soa cadea polipetídica organizada en dous dominios. Un dominio contén principalmente hélices α e o segundo é de tipo α/β.[6] O centro do suco catalítico está ocupado pola tétrade Ser35-Thr36-Thr37-Lys38, que inclúe o residuo Ser35 nucleófilo no extremo amino-terminal da hélice α2. Un lado da cavidade defínese polo bucle Ser96-Gly97-Cys98 conectando as hélices α4 e α5. A tríade Lys213-Thr214-Gly215 sitúase na febra β3 do lado oposto da cavidade. O grupo NH do esqueleto da molécula do residuo Ser35 esencial e a Ser216 de augas abaixo do motivo Lys213-Thr214-Gly215 occupan posicións que son compatibles coa función oco oxianión necesaria para a catálise.[6]
O encima clasifícase como DD-transpeptidase porque o enlace peptídico susceptible do doante de carbonilos establécese entre dous átomos de carbono coa configuración D.[6]
Función biolóxica
editarTodas as bacterais posúen polo menos unha e normalmente varias serina DD-peptidases monofuncionais.[3] Interveñen na formación da parede celular creando pontes peptídicas entre as febras de peptidoglicano.
Importancia en enfermidades
editarEse encima é unha excelente diana para fármacos porque é esencial, é accesible desde o periplasma e non ten equivalente nas células de mamíferos. A DD-transpeptidase é a proteína diana dos antibióticos β-lactámicos (por exemplo, a penicilina). Isto débese a que a estrutura da β-lactama lembra moito ao residuo D-ala-D-ala.
As β-lactamas exercen os seus efectos incativando competitivamente o sitio catalítico da serina DD-transpeptidase. A penicilina é un análogo catalítico dos doantes de carbonilo terminados en D-Ala-D-Ala; por tanto, en presenza deste antibiótico, a reacción detense cando se orixina o peniciloil ligado por éster á serina do encima.[11] Así, os antibióticos β-lactámicos forzan que estes encimas se comporten como proteínas que se unen á penicilina.[12]
Cineticamente, a interacción entre a DD-peptidase e as beta-lactamas é unha reacción en tres pasos:
As beta-lactamas poden formar un aduto E-I* de alta estabilidade coa DD-transpeptidase. A vida media deste aduto é dunhas horas, mentres que a vida media da reacción normal é da orde de milisegundos.[8]
A interferencia cos procesos do encima responsable da formación da parede celular ten como resultado a lise celular e a morte da bacteria debida ao desencadeamento do sistema autolítico da bacteria.[13]
Notas
editar- ↑ "E.C.3.4.16.4 Serine-type D-Ala-D-Ala carboxypeptidase". Enzyme Structures Database. Arquivado dende o orixinal o 17 de maio de 2006. Consultado o 26 de febreiro de 2006.
- ↑ Yocum RR, Waxman DJ, Rasmussen JR, Strominger JL (1979). "Mechanism of penicillin action: penicillin and substrate bind covalently to the same active site serine in two bacterial D-alanine carboxypeptidases". Proc Natl Acad Sci U S A 76 (6): 2730–4. Bibcode:1979PNAS...76.2730Y. PMC 383682. PMID 111240. doi:10.1073/pnas.76.6.2730.
- ↑ 3,0 3,1 Grandchamps J, Nguyen-Distèche M, Damblon C, Frère JM, Ghuysen JM (1995). "Streptomyces K15 active-site serine DD-transpeptidase: specificity profile for peptide, thiol ester and ester carbonyl donors and pathways of the transfer reactions". Biochem J. 307 ( Pt 2) (2): 335–9. PMC 1136653. PMID 7733866. doi:10.1042/bj3070335.
- ↑ Gordon E, Mouz N, Duée E, Dideberg O (xuño de 2000). "The crystal structure of the penicillin-binding protein 2x from Streptococcus pneumoniae and its acyl-enzyme form: implication in drug resistance". Journal of Molecular Biology 299 (2): 477–85. PMID 10860753. doi:10.1006/jmbi.2000.3740.
- ↑ Goffin C, Ghuysen JM (decembro de 2002). "Biochemistry and comparative genomics of SxxK superfamily acyltransferases offer a clue to the mycobacterial paradox: presence of penicillin-susceptible target proteins versus lack of efficiency of penicillin as therapeutic agent". Microbiology and Molecular Biology Reviews 66 (4): 702–38, table of contents. PMC 134655. PMID 12456788. doi:10.1128/MMBR.66.4.702-738.2002.
- ↑ 6,0 6,1 6,2 6,3 Fonzé E, Vermeire M, Nguyen-Distèche M, Brasseur R, Charlier P (xullo de 1999). "The crystal structure of a penicilloyl-serine transferase of intermediate penicillin sensitivity. The DD-transpeptidase of streptomyces K15". The Journal of Biological Chemistry 274 (31): 21853–60. PMID 10419503. doi:10.1074/jbc.274.31.21853.
- ↑ 7,0 7,1 Pratt RF (xullo de 2008). "Substrate specificity of bacterial DD-peptidases (penicillin-binding proteins)". Cellular and Molecular Life Sciences 65 (14): 2138–55. PMID 18408890. doi:10.1007/s00018-008-7591-7.
- ↑ 8,0 8,1 Walsh, Christopher; Wencewicz, Timothy (2016). Antibiotics: Challenges, Mechanisms, Opportunities (2ª ed.). American Society for Microbiology (Verlag). ISBN 978-1-55581-930-9.
- ↑ Ghuysen JM (outubro de 1994). "Molecular structures of penicillin-binding proteins and beta-lactamases". Trends in Microbiology 2 (10): 372–80. PMID 7850204. doi:10.1016/0966-842X(94)90614-9. hdl:2268/96404.
- ↑ Kelly JA, Kuzin AP, Charlier P, Fonzé E (abril de 1998). "X-ray studies of enzymes that interact with penicillins". Cellular and Molecular Life Sciences (Submitted manuscript) 54 (4): 353–8. PMID 9614972. doi:10.1007/s000180050163. hdl:2268/77968.
- ↑ Nguyen-Distèche M, Leyh-Bouille M, Ghuysen JM (outubro de 1982). "Isolation of the membrane-bound 26 000-Mr penicillin-binding protein of Streptomyces strain K15 in the form of a penicillin-sensitive D-alanyl-D-alanine-cleaving transpeptidase". The Biochemical Journal 207 (1): 109–15. PMC 1153830. PMID 7181854. doi:10.1042/bj2070109.
- ↑ 12,0 12,1 Ghuysen JM, Frère JM, Leyh-Bouille M, Nguyen-Distèche M, Coyette J, Dusart J, Joris B, Duez C, Dideberg O, Charlier P (1984). "Bacterial wall peptidoglycan, DD-peptidases and beta-lactam antibiotics". Scandinavian Journal of Infectious Diseases. Supplementum 42: 17–37. PMID 6597561.
- ↑ Spratt BG (maio de 1983). "Penicillin-binding proteins and the future of beta-lactam antibiotics. The Seventh Fleming Lecture". Journal of General Microbiology 129 (5): 1247–60. PMID 6352855. doi:10.1099/00221287-129-5-1247.
Véxase tamén
editarOutros artigos
editar- Vancomicina, un antibiótico que se une a residuos D-ala-D-ala, inhibindo a elongación pola glicosiltransferase
Ligazóns externas
editar- A base de datos on line MEROPS de peptidases e os seus inhibidores: S11.001[Ligazón morta]
- EC 3.4.16.4
- Serine-Type D-Ala-D-Ala Carboxypeptidase Medical Subject Headings (MeSH) na Biblioteca Nacional de Medicina dos EUA.