Electroforese en xel de agarosa
A electroforese en xel de agarosa é un método de electroforese en xel usado en bioquímica, bioloxía molecular, xenética e química clínica para separar unha poboación mixta de macromoléculas como ADN e proteínas nunha matriz de agarosa, un dos dous compoñentes principais do ágar. As biomoléculas sepáranse aplicando un campo eléctrico para mover as moléculas cargadas a través da matriz de agarosa, e as biomoléculas son separadas por tamaños na matriz do xel de agarosa.[1] As proteínas poden separarse pola carga e o tamaño (a electroforese en agarosa de isoelectroenfoque é esencialmente dependente do tamaño), e a lonxitude dos fragmentos de ADN e ARN.[2]
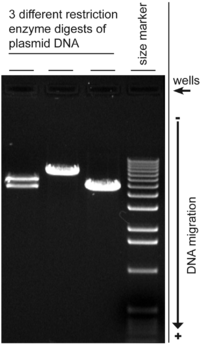
O xel de agarosa é doado de preparar, e ten relativamente poucos grupos cargados e é especialmente axeitado para a separación de ADN dos tamaños que se adoitan encontrar normalmente nos laboratorios, o que explica o estendido que está o seu uso. O ADN separado pode visulizarse con tinguiduras, normalmente baixo a luz ultravioleta, e os fragmentos poden ser extraídos do xel con relativa facilidade. A maioría das veces a agarosa usada está nunha concentración entre o 0,7 e o 2% disolta nun tampón electroforético adecuado.
Propiedades do xel de agarosa editar
O xel de agarosa é unha matriz tridimensional formada por moléculas helicoidais de agarosa en feixes supeenrolados que se agregan en estruturas tridimensionais con canles e poros polos cales poden pasar as biomoléculas.[3] A estrutura tridimensional mantense grazas a enlaces de hidróxeno e pode, por tanto, ser alterada quentándoa ata que volva ao estado líquido. A temperatura de fusión é diferente da temperatura de xelificación, dependendo das fontes, o xel de agarosa ten unha temperatura de xelificación de entre 35 e 42 °C e unha temperatura de fusión de 85 a 95 °C. Tamén se dispón de agarosas de fusión baixa e xelificación baixa que se obteñen por modificacións químicas.
O xel de agarosa ten un gran tamaño de poro e unha boa forza de xel, polo que é axeitado como medio anticonvección para a electroforese do ADN e de moléculas proteicas grandes. O tamaño do poro do xel ao 1% estimouse que está entre 100 nm e 200–500 nm,[4][5] e a forza do xel permite que os xeles se poidan diluír ata o 0,15% para formar un bloque para electroforese en xel.[6] Porén, os xeles de baixa concentración (0,1–0,2%) son fráxiles e, por tanto, difíciles de manexar. O xel de agrosa ten un poder de resolución inferior que o do xel de poliacrilamida para o ADN pero un maior rango de separación e, por tanto, úsase para fragmentos de ADN xeralmente de 50–20.000 pares de bases de tamaño. O límite de resolución dun xel de agarosa para a electroforese en xel de agarosa estándar é dunhas 750 kb, pero é posible tamén unha resolución duns 6 Mb con electroforese en xel de campo pulsado (PFGE).[7] Pode tamén utilizarse para separar grandes proteínas e é a matriz preferida para a electroforese en xel de partículas con raios efectivos maiores de 5 a 10 nm. Un xel de agarosa ao 0,9% ten poros dabondo grandes como para permitir a entrada do virus bacteriófago T4.[6]
O polímero de agarosa contén grupos cargados de piruvato e sulfato.[8] Estes grupos cargados negativamente crean un fluxo de auga en dirección oposta ao movemento do ADN nun proceso chamado electroendosmose (EEO), e pode, por tanto, retardar o movemento do ADN e causar unha difuminción das bandas. Os xeles de maior concentración teñen un maior fluxo electroosmótico. A agarosa de baixa EEO é, por tanto, a normalmente preferida para a electroforese en xel de ácidos nucleicos, pero a agarosa de alta EEO pode utilizarse para outros propósitos. O baixo contido en sulfato da agarosa de baixa EEO, especialmente a agarosa de baixo punto de fusión, é tamén beneficioso en casos nos que o ADN extraído do xel vai ser usado para unha posterior manipulación, xa que a presenza de sulfatos contaminantes pode afectar algúns dos procedementos posteriores, como a ligazón e a reacción en cadea da polimerase (PCR). Porén, as agarosas con EEO cero non son desexables para algunhas aplicacións, xa que poden prepararse engadindo grupos cargados positivamente e ditos grupos poden afectar reaccións encimáticas posteriores.[9] A electroendosmose (EEO) é a razón pola cal se usa a agarosa en vez de ágar, xa que o compoñente de agaropectina do ágar contén unha canidade significativa de sulfato cargado negativamente e grupos carboxilo. A retirada da agaropectina da agarosa reduce substancilmente a EEO, así como reduce a adsorción non específica de biomoléculas á matriz do xel. Porén, para algunhas aplicacións como a electroforese de proteínas do soro sanguíneo, pode ser conveniente unha alta EEO, e pode engadirse agaropectina ao xel utilizado.[10]
Factores que afectan a migración de ácidos nucleicos no xel editar
Hai varios factores que poden afectar a migración dos ácidos nucleicos no xel: a dimensión dos poros do xel (concentración do xel), tamaño do ADN que vai ser sometido a electroforese, a voltaxe utilizada, a forza iónica do tampón, e a concentración da tinguidura intercalante como o bromuro de etidio se for usado durante a electroforese.[11]
As moléculas pequenas viaxan máis rápido polo xel que as grandes, e o ADN bicatenario móvese a unha velocidade que é inversamente proporcional ao logaritmo do número de pares de bases. Porén, estas relacións non se cumpren con fragmentos de ADN moi grandes, e a separación de fragmentos de ADN moi grandes require o uso de electroforese en xel de campo pulsado (PFGE), a cal aplica correntes que se alternan en dúas direccións e os fragmentos de ADN grandes son separdos a medida que se reorientan coa corrente cambiante.[12]
Para unha electroforese en xel de agarosa estándar, as moléculas grandes resólvense mellor usando un xel a baixa concentración mentres que as moléculas menores sepáranse mellor con xeles de alta concentración. Porén, o xel de alta concentración require períodos de electroforese máis prolongados (ás veces de días).
O movemento do ADN pode vese afectado pola conformación da molécula de ADN, por exemplo, o ADN superenrolado xeralmente móvese máis rápido que o ADN relaxado porque está enrolado apertadamente e é máis compacto. En preparacións de ADN de plásmidos normais poden estar presentes múltiples formas de ADN.[13] A electroforese en xel dos plásmidos normalmente mostra na banda principal a forma con superenrolamento negativo, mentres que o ADN con amosega (forma circular aberta) e a forma circular pechada relaxada aparecen nas bandas menores. Non obstante, a velocidade á que se moven estas diversas formas pode cambiar usando condicións electroforéticas diferentes[14] e a mobilidade do ADN circular máis grande pode verse máis fortemente afectada que a do ADN liñal polo tamaño de poro do xel.[15]
O bromuro de etidio que se intercala no ADN circular pode cambiar a carga, lonxitude e superhelicidade da molécula de ADN, polo que a súa presenza no xel durante a electroforese pode afectar o seu movemento. Por exemplo, a carga positiva do bromuro de etidio pode reducir o movemento do ADN nun 15%.[12] A electroforese en xel de agarosa pode utilizarse para resolver o ADN circular con diferentes topoloxías de superenrolamento.[16]
Os danos no ADN debidos ao aumento do establecemento de enlaces cruzados tamén reduce a migración electroforética do ADN de maneira dependente da dose.[17][18]
A velocidade de migración do ADN é proporcional á voltaxe aplicada, é dicir, canta maior voltaxe, máis rápido se move o ADN. Porén, a resolución de grandes fragmentos de ADN é peor a voltaxes altas. A mobilidade do ADN pode tamén cambiar nun campo inestable, xa que nun campo que é invertido periodicamente a mobilidade do ADN dun determinado tamaño pode diminuír significativamente a unha determinada frecuencia de ciclos.[4] Este fenómeno pode ter como resultado a inversión de bandas na electroforese en xel de inversión de campo (FIGE), na que os fragmentos grandes de ADN se moven máis rápido que os pequenos.
Anomalías na migración editar
- Xeles "Smiley".- Este efecto de bordo é causado cando a voltaxe aplicada é demasiado alta para a concentración do xel usada.[19]
- Sobrecarga de ADN.- Esta sobrecarga fai máis lenta a migración dos fragmentos de ADN.
- Contaminación.- A presenza de impurezas, como sales ou proteínas pode afectar o movemento do ADN.
Mecanismo de migración e separación editar
A carga negativa do seu esqueleto de fosfato fai que o ADN se mova cara ao ánodo cargado positivamente durante a electroforese. Porén, a migración de moléculas de ADN na solución, e ausencia dunha matriz de xel, é independente do peso molecular durante a electroforese.[4][20] A matriz do xel é, por tanto, responsable da separación do ADN por tamaño durante a electroforese, e existen varios modelos para explicar o mecnismo de separación de biomoléculas na matriz do xel. Un modelo amplamente aceptado é o modelo Ogston, que trata a matriz do polímero como un baruto. Unha proteína globular ou un ADN con enrolamento ao azar móvese a través de porors interconectados, e o movemento das moléculas máis grandes é máis probable que sexa empecido e ralentizado polas colisións coa matriz do xel e as moléculas de diferentes tamaños poden separarse neste proceso de peneirado.[4]
Porén, o modelo Ogston falla para as moléculas grandes cando os poros son significativamente menores que a molécula. Para as moléculas de ADN de tamaño maior de 1 kb, utilízase xeralmente un modelo de reptación (ou as súas variantes). Este modelo asume que o ADN pode reptar "ao xeito das cobras" a través dos poros como unha molécula alongada. A forzas de campo eléctrico maiores aplícase un modelo de reptación nesgado, no que o extremo en posición dianteira da molécula está fortemente nesgado a ir na dirección cara a adiante e tira do resto da molécula.[21] Porén, a microscopia de fluorescencia en tempo real de moléculas tinguidas mostrou unha dinámica máis sutil durante a electroforese, na que o ADN mostraba unha considerable elasticidade a medida que se estiraba na dirección do campo aplicado e despois se contraía formando unha bóla, ou quedaba enganchado en forma de U cando era agarrado polas fibras do polímero.[22][23]
Procedemento xeral editar
Os detalles dun experimento de electroforese en xel de agarosa poden variar dependendo do método, pero a maioría segue un procedemento xeral.
Preparación do xel editar
O xel prepárase disolvendo pos de agarosa nun tampón apropiado, como o TAE ou o TBE, para usalo na electroforese.[24] A agarosa é dispersada no tampón antes de quentalo ata case o punto de ebulición, pero evitando chegar a el. A agarosa fundida déixase arrefriar o suficiente antes de verter a solución nun molde, xa que este podería torcerse ou rachar se a solución de agarosa está demasiado quente. Sitúase un peite no molde para crear os pozos para a carga da mostra e o xel debería estar completamente formado antes do seu uso.
A concentración do xel afecta a resolución da separación do ADN. Para unha electroforese en xel de agarosa estándar, un xel ao 0,8% dá lugar a unha boa separación ou resolución de grandes fragmentos de 5 a 10 kb, mentres que un xel ao 2% dá lugar a unha boa resolución para fragmentos pequenos de 0,2 a 1 kb. Os xeles ao 1% son utilizados a miúdo para unha electroforese estándar.[25] A concentración mídese en peso de agarosa por volume de tampón utilizado (g/ml). Xeles de altas porcentaxes adoitan ser crebadizos e poden non endurecerse uniformente, mentres que os xeles de porcentaxes baixas (do 0,1 ao 0,2%) son fráxiles e nada doados de manexar. Os xeles de agarosa de baixo punto de fusión son tamén máis fráxiles que os xeles de agarosa normais. A agarosa de baixo punto de fusión poden utilizarse por si soa ou simultaneamente con agarosa estándar para a separación e illamento de ADN.[26] A PFGE e a FIGE fanse a miúdo con xeles de agarosa de alta porcentaxe.
Carga das mostras editar
Unha vez que o xel calla, retírase o peite, que deixa os pozos onde se cargarán as mostras de ADN. O tampón de carga mestúrase coa mostra de ADN antes de que a mestura se cargue nos pozos. O tampón de carga contén un composto denso, que pode ser glicerol, sacarosa ou Ficoll, que aumenta a densidade da mostra de ADN para que esta se afunda ben ata o fondo do pozo.[27] Se a mostra de ADN contén etanol residual despois da preparación, este pode flotar fóra do pozo. O tampón de carga tamén inclúe tinguiduras coloreadas como o xileno cianol e o azul bromofenol usados para monitorizar o avance da electroforese. As mostras de ADN son cargadas usando unha pipeta.
Electroforese editar
A electroforese en xel de agarosa faise normalmente en horizontal en modo submarino no que o bloque do xel está completamente mergullado no tampón durante a electroforese. É tamén posible, pero menos común, realizar a electroforese verticalmente, ou horizontalmente co xel de elevado sobre patas de agarosa usando un aparello apropiado.[28] O tampón utilizado no xel é o mesmo que o tampón para correr o xel no tanque de electroforese, o cal explica por que é posible realizar a electroforese en modo submarino con xel de agarosa.
Para a resolución óptima do ADN maior de 2 kb de tamaño na electroforese en xel estándar, recoméndase de 5 a 8 V/cm (a distancia en cm refírese á distancia entre eléctrodos, polo que esta voltaxe recomendada sería de 5 a 8 multiplicada pola distancia entre os eléctrodos en cm).[14] A voltaxe pode tamén limitarse porque quenta o xel e pode causar que o xel funda se este é corrido a alta voltaxe durante un período prolongado, especialmente se o xel usado é un xel de agrosa de baixo punto de fusión. Unha voltaxe demasiado alta pode tamén reducir a resolución, e tamén orixina bandas moi finas para grandes moléculas de ADN. Voltaxes demasiado baixas poden causar o ensanchamento da banda para pequenos fragmentos de ADN debido á dispersión e difusión.[29]
Como o ADN non é visible á luz natural, o progreso da electroforese monitorízase usando tinguiduras. O xileno cianol (de cor azul clara) comigra cos grandes fragmentos de ADN, mentres que o azul bromofenol (cor azul escura) comigra cos fragmentos máis pequenos. Tinguiduras usadas menos comunmente son o vermello de cresol e o Orange G, que migran por diante do azul bromofenol. Tamén se fai correr á vez un marcador de ADN para a estimación do peso molecular de fragmentos de ADN. Porén, nótese que o tamaño dun ADN circular como o dos plásmidos non pode ser estimado con exactitude cos marcadores estándar a non ser que fose linearizado por dixestión con encimas de restrición, e alternativamente pode usarse un marcador de ADN superenrolado.
Tinguidura e visualización editar
O ADN e o ARN son visualizados normalmente tinguíndoos con bromuro de etidio, o cal se intercala no suco maior da hélice do ADN e fluoresce baixo a luz UV. A intercalación depende da concentración de ADN e así, unha banda cunha alta intensidade indica unha maior cantidade de ADN comparada cunha banda con menos intensidade.[12] O bromuro de etidio pode engadirse á solución de agarosa antes de que xelifique, ou o xel co ADN pode tinguirse máis tarde despois de rematada a electroforese. Destinguir o xel non é necesario pero pode producir imaxes mellores. Existen outros métodos de tinguido; exeplos son o SYBR Green, GelRed, azul de metileno, azul de cresilo brillante, azul Nilo sulfato e cristal violeta.[30] O SYBR Green, GelRed e outros produtos comerciais similares véndense como alternativas máis seguras que o bromuro de etidio, xa que este demostrou ser mutaxénico no test de Ames, aínda que a carcinoxenicidade do bromuro de etidio non foi realmente establecida polo momento. O SYBR Green require o uso dun transiluminador de luz azul. O ADN tinguido con cristal violeta pode verse con luz natural sen usar un transiluminador UV, o cal é unha vantaxe, aínda que pode non producir bandas fortes.
Cando se tingue con bromuro de etidio, o xel visualízase cun transiluminador de luz ultravioleta (UV). A luz UV excita os electróns do anel aromático do bromuro de etidio, e unha vez que volve ao estado fundamental, emítese luz que fai que o complexo do ADN e o bromuro de etidio fluoresza.[12] Os transiluminadores estándar usan lonxitudes de onda de 302/312-nm (UV-B), aínda que a exposición do ADN á radiación UV durante menos de 45 segundos pode producir danos ao ADN e afectar procedementos subseguintes, por exemplo reducir a eficiencia da transformación, da transcrición in vitro e da PCR.[31] A exposición do ADN á radiación UV debería, pois, ser limitada. Usar unha maior lonxitude de onda de 365 nm (no rango do UV-A) causa menos danos ao ADN pero tamén produce unha fluorescencia moito máis feble con bromuro de etidio. Sempre que se poidan seleccionar varias lonxitudes de onda no transiluminador, deberían utilizarse as lonxitudes de onda máis curtas para capturar as imaxes, mentres que as lonxitudes de onda máis longas son as que cómpre usar se hai que traballar co xel durante longos períodos de tempo.
O aparello transiluminador pode tamén conter dispositivos para a captura de imaxes, como unha cámara dixital ou polaroid, que permitan tomar ou imprimir unha imaxe do xel.
Para a electroforese en xel de proteínas, as bandas poden visualizarse con azul Coomassie ou tinguidura de prata.
Procedementos finais editar
As bandas de ADN separadas utilízanse a miúdo para procedementos posteriores, e poden cortarse do xel láminas que conteñan a banda de ADN, que despois é disolto e purificado. Porén, os contaminantes poden afectar algúns procedementos posteriores como a PCR, e a agarosa de baixo punto de fusión é preferible nalgúns casos, xa que contén poucos sulfatos que poidan afectar algunhas reaccións encimáticas. Os xeles poden tamén utilizarse en técnicas de blotting.
Tampóns editar
En xeral, o tampón ideal debería ter unha boa condutividade, producir menos calor e ter unha vida longa.[32] Na electroforese con agarosa pódense utilizar varios tampóns; algúns comúns para ácidos nucleicos son o Tris/Acetato/EDTA (TAE) e o Tris/Borato/EDTA (TBE). Os tampóns usados conteñen EDTA para inactivar moitas nucleases que requiren un catión divalente para funcionar. O borato no tampón TBE pode ser problemático xa que o borato pode polimerizarse ou interaccionar con cis dioles como os que se encontran no ARN. O TAE ten a menor capacidade de tamponamento, pero proporciona a mellor resolución para ADN longos. Isto significa o uso dunha menor voltaxe e máis tempo, pero obtense un mellor produto.
Propúxose o uso de moitos outros tampóns, por exemplo o borato de litio (LB), a histidina isoeléctrica, tampóns con pK coincidente etc.; na maioría dos casos a razón aducida para o seu uso é utilizar unha menor corrente eléctrica (menos calor) e mobilidades iónicas coincidentes, o que fai que o tampón teña unha maior duración. O tampón Tris-fosfato ten unha alta capacidade de tamponamento peron on pode utilizarse se o ADN extraído ten que usarse nunha reacción sensible ao fosfato. O LB é relativamente novo e non é efectivo á hora de resolver fragmentos maiores de 5 kbp. Porén, pola súa baixa condutividade, pode utilizarse unha voltaxe moito maior (ata 35 V/cm), o que significa un tempo de análise máis curto nunha electroforese de rutina. Poden resolverse diferenzas en tamaño de tan só dun par de bases usando un xel de agarosa ao 3% cun medio de condutividade extremadamente baixa (1 mM de borato de litio).[33]
Para aplicacións específicas poden utilizarse outros sistemas de tamponamento, por exemplo, ácido barbitúrico-barbiturato de sodio ou tamén tampóns Tris-barbiturato na electroforese en xel de agarosa de proteínas, por exemplo na detección da distribución anormal de proteínas.[34]
Aplicacións editar
- Estimación do tamaño de moléculas de ADN despois da dixestión con encimas de restrición, por exemplo para facer mapas de restrición de ADN clonado.
- Análise de produtos dunha reacción en cadea da polimerase (PCR), por exemplo, en diagnose xenética molecular ou pegada xenética.
- Separación de fragmentos de ADN para a extracción e a purificación.
- Separación de ADN xenómico restrinxido antes da transferencia Southern, ou de ARN antes da transferencia northern.
- Separación de proteínas, por exemplo, cribado de anormalidades de proteínas en química clínica.[35]
Os xeles de agarosa son doados de colocar no molde e de manexarse en comparación con outras matrices e os ácidos nucleicos non son alterados quimicamente durante a electroforese. As mostras son tamén recuperadas facilmente. Unha vez que o experimento remata, o xel resultante pode ser almacenado nunha bolsa de plástico nun refrixerador.
A electroforese realízase en solucións tampón para reducir os cambios de pH debidos ao campo eléctrico, o cal é importante porque a crga do ADN e ARN depende do pH, pero poñela a funcionar durante longo tempo pode esgotar a capacidade de tamponamento da solución. Ademais, diferentes prepracións de material xenético poden non migrar de maneira igual por razóns morfolóxicas ou outras.
Notas editar
- ↑ Sambrook J, Russel DW (2001). Molecular Cloning: A Laboratory Manual 3rd Ed. Cold Spring Harbor Laboratory Press. Cold Spring Harbor, NY.
- ↑ Kryndushkin DS, Alexandrov IM, Ter-Avanesyan MD, Kushnirov VV (December 2003). "Yeast [PSI+] prion aggregates are formed by small Sup35 polymers fragmented by Hsp104". The Journal of Biological Chemistry 278 (49): 49636–43. PMID 14507919. doi:10.1074/jbc.M307996200.
- ↑ Joseph Sambrook; David Russell. "Chapter 5, protocol 1". Molecular Cloning - A Laboratory Manual 1 (3rd ed.). p. 5.4. ISBN 978-0-87969-577-4.
- ↑ 4,0 4,1 4,2 4,3 Zimm BH, Levene SD (May 1992). "Problems and prospects in the theory of gel electrophoresis of DNA" (PDF). Quarterly Reviews of Biophysics 25 (2): 171–204. PMID 1518924. doi:10.1017/s0033583500004662.
- ↑ Jean-Louis Viovy (2000). "Electrophoresis of DNA and other polyelectrolytes: Physical mechanisms". Reviews of Modern Physics 72: 813–872. Bibcode:2000RvMP...72..813V. doi:10.1103/RevModPhys.72.813.
- ↑ 6,0 6,1 Philip Serwer (1983). "Agarose gels: Properties and use for electrophoresis". Electrophoresis 4 (6): 375–382. doi:10.1002/elps.1150040602.
- ↑ Joseph Sambrook; David Russell. "Chapter 5, protocol 1". Molecular Cloning - A Laboratory Manual 1 (3rd ed.). p. 5.2–5.3. ISBN 978-0-87969-577-4.
- ↑ "Appendix B: Agarose Physical Chemistry" (PDF). Lonza Group.
- ↑ Joseph Sambrook; David Russell. "Chapter 5, protocol 1". Molecular Cloning - A Laboratory Manual 1 (3rd ed.). p. 5.7. ISBN 978-0-87969-577-4.
- ↑ Keren, David (26 September 2003). Protein Electrophoresis in Clinical Diagnosis. CRC Press. pp. 7–8. ISBN 978-0340812136.
- ↑ G. Lucotte; F. Baneyx (1993). Introduction to Molecular Cloning Techniques. Wiley-Blackwell. p. 32. ISBN 978-0471188490.
- ↑ 12,0 12,1 12,2 12,3 Lee PY, Costumbrado J, Hsu CY, Kim YH (April 2012). "Agarose gel electrophoresis for the separation of DNA fragments". Journal of Visualized Experiments (62). PMC 4846332. PMID 22546956. doi:10.3791/3923.
- ↑ Richard R. Sinden. DNA Structure and Function. Academic Press Inc. p. 97. ISBN 978-0126457506.
- ↑ 14,0 14,1 Joseph Sambrook; David Russell. "Chapter 5, protocol 1". Molecular Cloning - A Laboratory Manual 1 (3rd ed.). p. 5.5-5.6. ISBN 978-0-87969-577-4.
- ↑ Aaij C, Borst P (May 1972). "The gel electrophoresis of DNA". Biochimica et Biophysica Acta 269 (2): 192–200. PMID 5063906. doi:10.1016/0005-2787(72)90426-1.
- ↑ Donald Voet; Judith G. Voet (1995). Biochemistry (2nd ed.). John Wiley & Sons. pp. 877–878. ISBN 978-0471586517.
- ↑ Blasiak J, Trzeciak A, Malecka-Panas E, Drzewoski J, Wojewódzka M (August 2000). "In vitro genotoxicity of ethanol and acetaldehyde in human lymphocytes and the gastrointestinal tract mucosa cells". Toxicology in Vitro 14 (4): 287–95. PMID 10906435. doi:10.1016/S0887-2333(00)00022-9.
- ↑ Lu Y, Morimoto K (July 2009). "Is habitual alcohol drinking associated with reduced electrophoretic DNA migration in peripheral blood leukocytes from ALDH2-deficient male Japanese?". Mutagenesis 24 (4): 303–8. PMID 19286920. doi:10.1093/mutage/gep008.
- ↑ G. Lucotte; F. Baneyx (1993). Introduction to Molecular Cloning Techniques. Wiley-Blackwell. p. 41. ISBN 978-0471188490.
- ↑ Robert W. Old; Sandy B. Primrose. Principle of Gene Manipulation - An Introduction to Genetic Engineering (5th ed.). Blackwell Scientific. p. 9. ISBN 9780632037124.
- ↑ Li Zhu; Hong Wang. "Chapter 4 - Genetic Analysis in Miniaturized Electrophoresis Systems". En Tian, Wei-Cheng; Finehout, Erin. Microfluidics for Biological Applications. Springer. p. 125. ISBN 978-0-387-09480-9.
- ↑ Smith SB, Aldridge PK, Callis JB (January 1989). "Observation of individual DNA molecules undergoing gel electrophoresis". Science 243 (4888): 203–6. Bibcode:1989Sci...243..203S. PMID 2911733. doi:10.1126/science.2911733.
- ↑ Schwartz DC, Koval M (April 1989). "Conformational dynamics of individual DNA molecules during gel electrophoresis". Nature 338 (6215): 520–2. Bibcode:1989Natur.338..520S. PMID 2927511. doi:10.1038/338520a0.
- ↑ Pei Yun Lee; John Costumbrado; Chih-Yuan Hsu; Yong Hoon Kim. "Agarose Gel Electrophoresis for the Separation of DNA Fragments". The Journal of Visualized Experiments. PMID 22546956.
- ↑ "Agarose gel electrophoresis (basic method)". Biological Protocols. Consultado o 23 August 2011.
- ↑ Fotadar U, Shapiro LE, Surks MI (February 1991). "Simultaneous use of standard and low-melting agarose for the separation and isolation of DNA by electrophoresis". BioTechniques 10 (2): 171–2. PMID 2059440.
- ↑ Lee PY, Costumbrado J, Hsu CY, Kim YH (April 2012). "Agarose gel electrophoresis for the separation of DNA fragments". Journal of Visualized Experiments 62 (62). PMC 4846332. PMID 22546956. doi:10.3791/3923.
- ↑ David Freifelder (1982). Physical Biochemistry: Applications to Biochemistry and Molecular Biology (2nd ed.). WH Freeman. pp. 292–293. ISBN 978-0716714446.
- ↑ "Section III: Loading and Running DNA in Agarose Gels" (PDF). Lonza Group.
- ↑ "DNA revealed" (PDF). National Centre for Biotechnology education. University of Reading. Arquivado dende o orixinal (PDF) o 04 de marzo de 2012. Consultado o 01 de outubro de 2018.
- ↑ Gründemann D, Schömig E (November 1996). "Protection of DNA during preparative agarose gel electrophoresis against damage induced by ultraviolet light" (PDF). BioTechniques 21 (5): 898–903. PMID 8922632. Arquivado dende o orixinal (PDF) o 04 de marzo de 2016. Consultado o 01 de outubro de 2018.
- ↑ Sameh Magdeldin, ed. (2012). Gel electrophoresis – Principles and Basics. InTech. ISBN 978-953-51-0458-2.
- ↑ Brody JR, Kern SE (October 2004). "History and principles of conductive media for standard DNA electrophoresis" (PDF). Analytical Biochemistry 333 (1): 1–13. PMID 15351274. doi:10.1016/j.ab.2004.05.054. Arquivado dende o orixinal (PDF) o 24 de decembro de 2012. Consultado o 01 de outubro de 2018.
- ↑ Jeppsson JO, Laurell CB, Franzén B (April 1979). "Agarose gel electrophoresis". Clinical Chemistry 25 (4): 629–38. PMID 313856.
- ↑ "Agarose gel electrophoresis: Application in Clinial Chemistry" (PDF). Journal of Medical Biochemistry 29: 9–14. 2010. doi:10.2478/v10011-009-0033-8.
Véxase tamén editar
Outros artigos editar
- Electroforese en xel
- Inmunodifusión, Inmunoelectroforese
- SDD-AGE
- Northern blot
- SDS-PAGE
- Southern blot
Ligazóns externas editar
- Como facer a electroforese dun xel de ADN ou ARN
- Animación da análise en xel de fragmentos de restrición de ADN
- Vídeo e artigo sobre a electroforese en xel de agarosa
- Fotos paso a paso da electroforese dun xel e extracción de ADN
- Drinking straw electrophoresis!
- A typical method from wikiversity
- Construción dunha cámara de electroforese en xel