Northern blot
O northern blot (transferencia northern) é unha técnica utilizada en bioloxía molecular para estudar a expresión xénica por medio da detección de ARN (ou ARNm illado) nunha mostra.[1][2]
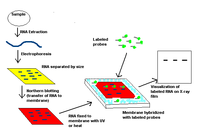
Co northern blot é posible observar o control celular sobre as estruturas e funcións determinando os niveis de expresión xénica particulares durante a diferenciación, morfoxénese, e en condicións patolóxicas ou anormais.[3] No northern blot utilízase primeiro electroforese para separar mostras de ARN polo seu tamaño e despois faise unha detección con sondas de hibridación complementarias dunha parte da secuencia completa diana. Sendo moi específico, o termo "northern blot" refírese, en realidade, só á transferencia por capilaridade do ARN desde o xel de electroforese á membrana de transferencia, mentres que o proceso completo xeralmente se denominaría "northern blotting".[4] A técnica do northern blot foi desenvolvida en 1977 por James Alwine, David Kemp, e George Stark na Universidade Stanford.[5] O northern blot recibiu ese nome facendo un xogo de palabras coa técnica previa do Southern blot, denominada así polo biólogo que a inventou, Edwin Southern (neste caso como é un apelido escríbese con maiúscula; western blot non é necesario escribilo con maiúscula, aínda que moitas veces tamén se fai por analoxía).[1] A principal diferenza entre estas dúas técnicas é que no northern blot analízase ARN e no Southern blot ADN.[6]
Procedemento
editarUn procedemento xeral de blotting[4] comeza coa extracción do ARN total dunha mostra de tecido homoxeneizado ou de células. O ARNm eucariótico pode despois illarse utilizando cromatografía en celulosa con oligo (dT) para illar só aqueles ARNs que teñan cola poli(A).[7][8] As mostras de ARN son despois separadas por electroforese en xel. Como os xeles son fráxiles e as sondas son incapaces de entrar na matriz, as mostras de ARN, agora separadas por tamaño, son transferidas a unha membrana de nailon por medio dun sistema de capilaridade ou de baleiro.
O máis efectivo no northern blot é usar unha membrana de nailon con carga positiva, xa que as cargas negativas dos ácidos nucleicos (dos seus fosfatos) teñen unha alta afinidade por elas. O tampón de transferencia utilizado para o blotting xeralmente contén formamida porque isto rebaixa a temperatura de aliñamento (annealing) da interacción sonda-ARN, o que evita ter que utilizar temperaturas elevadas, o cal podería causar a degradación do ARN.[9] Unha vez que o ARN foi transferido á membrana, inmobilízase uníndoo á membrana por medio de enlaces covalentes, con luz UV ou con calor. Unha vez que se etiquetou a sonda, esta hibrídase co ARN na membrana. As condicións experimentais que poden afectar á eficiencia e especificidade da hibridación inclúen a forza iónica, viscosidade, lonxitude do dúplex, pares de bases mal apareados, e composición de bases.[10] A membrana lávase para asegurar que a sonda se uniu especificamente e impedir que se orixinen sinais de fondo ("ruído"). Os sinais híbridos son despois detectados cunha película de raios X e poden cuantificarse por densitometría. Despois dunha determinación por micromatrices ou RT-PCR, poden crearse controis para a comparación da northern blot utilizando unha mostra que non presenta o produto xénico de interese.[10]
Xeles
editarAs mostras de ARN son normalmente separadas en xeles de agarosa que conteñen formaldehido como axente [[Desnaturalización de ácidos nucleicos|desnaturalizante para o ARN para limitar a súa estrutura secundaria.[10][11] Os xeles poden tinguirse con bromuro de etidio (EtBr) e despois ser ollados baixo luz UV para observar a calidade e cantidade de ARN antes da transferencia (blotting).[10] Pode tamén utilizarse a electroforese en xel de poliacrilamida con urea para a separación do ARN, pero o seu uso máis común é para ARN fragmentado ou microARNs.[12] Xeralmente na electroforese en xel faise correr unha escada de ARN xunto coas mostras para observar así o tamaño dos fragmentos obtidos, pero nas mostras de ARN total as propias subunidades ribosómicas poden actuar como marcadores de tamaño.[10] Como a subunidade ribosómica maior é de 28S (aproximadamente 5kb) e a menor é de 18S (aproximadamente 2kb) aparecerán no xel dúas prominentes bandas, a maior de case o dobre de intensidade que a menor.[10][13]
Sondas
editarAs sondas para northern blot están compostas de ácidos nucleicos cunha secuencia complementaria a unha parte ou a todo o ARN de interese, poden ser de ADN, ARN, ou oligonucleótidos cun mínimo de 25 bases complementarias coa secuencia diana.[4] As sondas de ARN (ribosondas) que se transcriben in vitro poden soportar uns pasos de lavado máis rigorosos, o que impide que apareza nos resultados parte do "ruído" de fondo.[10] Normalmente, para funcionar como sonda de northern blot créase ADNc con cebadores etiquetados para a secuencia do ARN de interese.[14] As sondas deben etiquetarse con isótopos radioactivos (32P) ou ben con quimioluminescencia, na cal os encimas fosfatase alcalina ou peroxidase do ravo picante (Armoracia rusticana) degradan os substratos quimioluminescentes producindo unha emisión de luz detectable.[15] A etiquetaxe quimiolunimescente pode facerse de dous xeitos: pode unirse o encima á sonda, ou pode etiquetarse a sonda cun ligando (por exemplo, biotina), para o cal se une ao encima unha proteína (por exemplo, avidina ou estreptavidina).[10] Cunha película de raios X poden detectarse tanto os sinais radioactivos coma os quimioluminescentes, e moitos investigadores prefiren os sinais quimioluminescentes porque son máis rápidos, máis sensibles, e reducen os perigos para a saúde aociados coas marcaxes radioactivas.[15] Unha mesma membrana pode sondarse ata cinco veces sen que se produza unha perda significativa de ARN diana.[9]
Aplicacións
editarO northern blot permite a observación do patrón de expresión dun xene particular en distintos tecidos, órganos, estadios do desenvolvemento, niveis de estrés ambiental, infeccións de patóxenos, e ao longo do curso dun tratamento.[8][14][16] A técnica foi utilizada para mostrar a sobreexpresión de oncoxenes e a regulación á baixa de xenes supresores de tumores en células cancerosas cando se comparan co tecido "normal",[10] e tamén a expresión xénica no rexeitamento de órganos transplantados.[17] Se se observa que un xene é regulado porque se detecta unha abundancia do seu ARNm no northern blot feito á mostra, este xene pode despois ser secuenciado para determinar se é un xene coñecido polos investigadores ou se é un descubrimento novo.[17] Os patróns de expresión obtidos baixo unhas condicións determinadas pode servir para comprender mellor a función dese xene. Como o ARN se separa primeiro por tamaños, en caso de que se use só un tipo de sonda as variacións no nivel de cada banda na membrana poden dar unha idea do tamaño do produto, o que pode suxerir a existencia de produtos de splicing alternativo do mesmo xene ou motivos de secuencia repetidos.[7][13] A variación en tamaño dun produto dun xene pode tamén indicar delecións ou erros no procesamento do transcrito. Alterando a diana da sonda utilizada na secuencia coñecida, é posible determinar que rexión do ARN se perdeu.[1]
BlotBase é unha base de datos online que publica northern blots. BlotBase leva publicados uns 700 northern blots de mostras humanas e de ratos, duns 650 xenes de máis de 25 tipos de tecido.[3] Os northern blots poden buscarse na base de datos por un ID de blot, referencia de publicación, identificador de xene, ou por tecido.[3] Os resultados dunha busca proporcionan a ID de blot, especie, tecido, xene, nivel de expesión, imaxe do blot (se está dispoñible), e ligazóns á publicación do traballo no que se orixinaron os datos.[3]
Vantaxes e desvantaxes
editarAs análises de expresión xénica poden facerse por varios métodos como RT-PCR, ensaios de protección de RNase, micromatrices, análises en serie da expresión xénica (SAGE), e tamén por northern blotting.[3][4] As micromatrices son bastante usadas e xeralmente son concordantes cos datos obtidos por northern blots; porén, ás veces o northern blot pode detectar pequenos cambios na expresión xénica que pasan desapercibidos nas micromatrices.[18] A vantaxe que teñen as micromatrices sobre os northern blots é que poden visualizarse miles de xenes á vez, mentres que no northern blot o normal é observar un ou uns poucos xenes.[16][18]
Un problema frecuente do northern blot é a degradación da mostra por RNases (tanto endóxenas da mostra coma procedentes de contaminación ambiental), o cal pode evitarse facendo unha axeitada esterilización do material de vidro e utilizando inhibidores da RNase como DEPC (dietilpirocarbonato).[4] Os compostos químicos utilizados na maioría dos northern blots poden supoñer un risco para o investigador, xa que o formaldehido, o material radioactivo, o bromuro de etidio, o DEPC, e a luz UV son todos nocivos con certas exposicións.[10] Comparada coa RT-PCR, o northern blot ten unha baixa sensibilidade, mais tamén ten unha alta especificidade, o cal é importante para reducir os resultados de falsos positivos.[10]
Entre as vantaxes de usar o northern blot están a detección dos tamaños do ARN, a observación de produtos de splicing alternativos, o uso de sondas con homoloxía parcial, a calidade e cantidade de ARN pode medirse no xel antes de facer o blotting, e as membranas poden almacenarse e resondarse durante anos despois do blotting.[10]
Northern blot inverso
editarOs investigadores en ocasións usan unha variante desta técnica denominada northern blot inverso ou reverso. Neste procedemento, o substrato ácido nucleico (que está fixado a unha membrana) é un conxunto de fragmentos de ADN, e a sonda é un ARN extraído dun tecido e marcado radioactivamente.
O uso de micromatrices de ADN que se xeneralizou a finais da década de 1990 e inicios da de 2000 é moi similar ao procedemento inverso, porque implica o uso de fragmentos de ADN illados fixados a un substrato, e a hibridación cunha sonda feita de ARN celular. Así, o procedemento inverso, aínda que inicialmente era pouco común, permitiu que as análises mediante northern evolucionasen para permitir o estudo de perfís de expresión xénica, nos cales se pode monitorizar a expresión de moitos dos xenes (ou posiblemente de todos) dun organismo.
Notas
editar- ↑ 1,0 1,1 1,2 Alberts, B., Johnson, A., Lewis, J. Raff, M., Roberts, K., Walter, P. 2008. Molecular Biology of the Cell, 5th ed. Garland Science, Taylor & Francis Group, NY, pp 538–539.
- ↑ Kevil, C. G., Walsh, L., Laroux, F. S., Kalogeris, T., Grisham, M. B., Alexander, J. S. (1997) An Improved, Rapid Northern Protocol. Biochem. and Biophys. Research Comm. 238:277–279.
- ↑ 3,0 3,1 3,2 3,3 3,4 Schlamp, K.; Weinmann, A.; Krupp, M.; Maass, T.; Galle, P. R.; Teufel, A. (2008). "BlotBase: A northern blot database". Gene 427 (1–2): 47–50. PMID 18838116. doi:10.1016/j.gene.2008.08.026.
- ↑ 4,0 4,1 4,2 4,3 4,4 Trayhurn, P. (1996) Northern Blotting. Pro. Nutrition Soc. 55:583–589.
- ↑ Alwine JC, Kemp DJ, Stark GR (1977). "Method for detection of specific RNAs in agarose gels by transfer to diazobenzyloxymethyl-paper and hybridization with DNA probes". Proc. Natl. Acad. Sci. U.S.A. 74 (12): 5350–4. PMC 431715. PMID 414220. doi:10.1073/pnas.74.12.5350.
- ↑ Bor, Y.C.; Swartz, J.; Li, Y.; Coyle, J.; Rekosh, D.; Hammarskjold, Marie-Louise (2006). "Northern Blot analysis of mRNA from mammalian polyribosomes". Nature Protocols. doi:10.1038/nprot.2006.216.
- ↑ 7,0 7,1 Durand, G. M.; Zukin, R. S. (1993). "Developmental Regulation of mRNAs Encoding Rat Brain Kainate/AMPA Receptors: A Northern Analysis Study". J. Neurochem 61 (6): 2239–2246. PMID 8245974. doi:10.1111/j.1471-4159.1993.tb07465.x.
- ↑ 8,0 8,1 Mori, H.; Takeda-Yoshikawa, Y.; Hara-Nishimura, I.; Nishimura, M. (1991). "Pumpkin malate synthase Cloning and sequencing of the cDNA and Northern blot analysis". Eur. J. Biochem 197 (2): 331–336. PMID 1709098. doi:10.1111/j.1432-1033.1991.tb15915.x.
- ↑ 9,0 9,1 Yang, H.; McLeese, J.; Weisbart, M.; Dionne, J.-L.; Lemaire, I.; Aubin, R. A. (1993). "Simplified high throughput protocol for Northern hybridization". Nucleic Acids Research 21 (14): 3337–3338. PMC 309787. PMID 8341618. doi:10.1093/nar/21.14.3337.
- ↑ 10,00 10,01 10,02 10,03 10,04 10,05 10,06 10,07 10,08 10,09 10,10 10,11 Streit, S.; Michalski, C. W.; Erkan, M.; Kleef, J.; Friess, H. (2009). "Northern blot analysis for detection of RNA in pancreatic cancer cells and tissues". Nature Protocols 4 (1): 37–43. PMID 19131955. doi:10.1038/nprot.2008.216.
- ↑ Yamanaka, S.; Poksay, K. S.; Arnold, K. S.; Innerarity, T. L. (1997). "A novel translational repressor mRNA is edited extensively in livers containing tumors caused by the transgene expression of the apoB mRNA-editing enzyme". Genes Dev. 11 (3): 321–333. PMID 9030685. doi:10.1101/gad.11.3.321.
- ↑ Valoczi, A., Hornyik, C., Varga, N., Burgyan, J., Kauppinen, S., Havelda, Z. (2004) Sensitive and specific detection of microRNAs by northern blot analysis using LNA-modified oligonucleotide probes. Nuc. Acids Research. 32: e175.
- ↑ 13,0 13,1 Gortner, G.; Pfenninger, M.; Kahl, G.; Weising, K. (1996). "Northern blot analysis of simple repetitive sequence transcription in plants". Electrophoresis 17 (7): 1183–1189. PMID 8855401. doi:10.1002/elps.1150170702.
- ↑ 14,0 14,1 Liang, P. Pardee, A. B. (1995) Recent advances in differential display. Current Opinion Immunol. 7: 274–280.
- ↑ 15,0 15,1 Engler-Blum, G.; Meier, M.; Frank, J.; Muller, G. A. (1993). "Reduction of Background Problems in Nonradioactive Northern and Southern Blot Analysis Enables Higher Sensitivity Than 32P-Based Hybridizations". Anal. Biochem 210 (2): 235–244. PMID 7685563. doi:10.1006/abio.1993.1189.
- ↑ 16,0 16,1 Baldwin, D., Crane, V., Rice, D. (1999) A comparison of gel-based, nylon filter and microarray techniques to detect differential RNA expression in plants. Current Opinion in Plant Biol. 2: 96–103.
- ↑ 17,0 17,1 Utans, U.; Liang, P.; Wyner, L. R.; Karnovsky, M. J.; Russel, M. E. (1994). "Chronic cardiac rejection: Identification of five upregulated genes in transplanted hearts by differential mRNA display". Proc. Natl. Acad. Sci. USA 91 (14): 6463–6467. PMC 44222. PMID 8022806. doi:10.1073/pnas.91.14.6463.
- ↑ 18,0 18,1 Taniguchi, M.; Miura, K.; Iwao, H.; Yamanaka, S. (2001). "Quantitative Assessment of DNA Microarrays – Comparison with Northern Blot Analysis". Genomics 71 (1): 34–39. PMID 11161795. doi:10.1006/geno.2000.6427.
Véxase tamén
editarOutros artigos
editarExternal links
editar| Wikimedia Commons ten máis contidos multimedia na categoría: Northern blot |