Operón trp
O operón trp é un operón, é dicir, un grupo de xenes que se utilizan ou transcriben xuntos de forma regulada, que codifica os compoñentes necesarios para a produción do aminoácido triptófano. O operón trp está presente en moitas bacterias, e foi primeiramente caracterizado na especie Escherichia coli. Este operón está regulado para que cando o triptófano estea presente no ambiente, os xenes para a súa síntese non funcionen, e cando non estea presente si. Foi un importante sistema experimental para comprender a regulación xénica e utilízase frecuentemente para explicala.
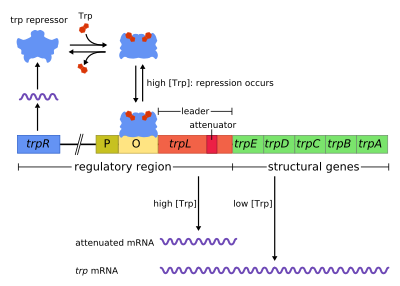
O operón trp contén cinco xenes estruturais: trpE, trpD, trpC, trpB e trpA, que codifican compoñentes enzimáticos da vía metabólica. Tamén contén un xene regulador represivo chamado trpR. O trpR ten un promotor ao que se une a ARN polimerase e sintetiza o ARNm dunha proteína regulatoria. A proteína que se sintetiza a partir do trpR únese despois ao operador, o que causa o bloqueo da transcrición. No operón trp, o triptófano únese á proteína represora bloqueando a transcrición do xene. Nesta situación, a represión significa que a ARN polimerase vai transcribir os xenes do operón. Ademais, a diferenza do que ocorre no operón lac, o operón trp contén un péptido líder e unha secuencia atenuadora, que permite unha regulación graduada.[1]
É un exemplo de regulación negativa represible da expresión xénica. Dentro da secuencia regulatoria do operón, o operador é bloqueado pola proteína represora en presenza de triptófano (o que impide a transcrición e a formación de máis triptófano, xa que xa hai dabondo) e é liberado do bloqueo en ausencia de triptófano (permitindo así a transcrición).
Xenes
editarO operón trp consta de cinco xenes estruturais, cuxas funcións son:
- TrpE (P00895): xene da antranilato sintase, que produce antranilato.
- TrpD (P00904): coopera con TrpE.
- TrpC (P00909): xene dun enzima bifuncional. O seu dominio de fosforribosilantranilato isomerase, primeiro converte o N-(5-fosfo-β-D-ribosil)antranilato en 1-(2-carboxifenilamino)-1-desoxi-D-ribulosa 5-fosfato. A porción de indol-3-glicerol-fosfato sintase da mesma proteína converte ese produto en (1S,2R)-1-C-(indol-3-il)glicerol 3-fosfato.
- TrpA (P0A877), TrpB (P0A879): xenes de dúas subunidades da triptófano sintetase, cadea alfa e beta, respectivamente. Combina o produto do TrpC con serina para orixinar triptófano.
Represión
editarO operón funciona por medio dun mecanismo de retroalimentación represible negativa. O represor do operón trp prodúcese augas arriba no xene trpR, que se expresa constitutivamente a baixo nivel. Os monómeros de trpR sintetizados asócianse formando dímeros. Cando está presente o triptófano, estes dímeros represores do triptófano únense ao triptófano, causando un cambio na conformación do represor, que permiten que o represor se una ao operador. Isto impide que a ARN polimerase se una e transcriba o operón, polo que o triptófano non se produce a partir do seu precursor. Cando non está presente o triptófano, o represor está na súa conformación activa e non pode unirse á rexión operadora, polo que a transcrición non é inhibida polo represor.
Atenuación
editarA atenuación é un segundo mecanismo de retroalimentación negativa no operón trp. O sistema de represión depende da concentración intracelular de triptófano, mentres que a atenuación responde á concentración de moléculas cargadas con triptófano de ARNttrp.[2] Así, o represor trpR diminúe a expresión xénica alterando a iniciación da transcrición, mentres que a atenuación faino alterando o proceso de transcrición que xa está en progreso.[2] Mentres que o represor TrpR diminúe a transcrición nun factor de 70, a atenuación pode diminuílo adicionalmente nun factor de 10, o que permite chegar a unha represión total 700 veces maior.[3] A atenuación faise posible porque en procariotas (que non teñen núcleo celular), os ribosomas empezan a traducir o ARNm cando a ARN polimerase está aínda transcribindo a secuencia de ADN. Deste modo, o proceso de tradución afecta directamente a transcrición do operón.
Na parte inicial dos xenes transcritos do operón trp hai unha secuencia de polo menos 130 nucleótidos denominada transcrito líder (trpL; P0AD92 ).[4] Lee e Yanofsky (1977) descubriron que a atenuación está eficazmente correlacionada coa estbilidade dunha estrutura secundaria incrustada no trpL,[5] e as forquitas constituíntes da estrutura terminadora foron despois determinadas por Oxender et al. (1979).[6] Este transcrito inclúe catro curtas secuencias designadas do 1 ao 4, cada unha das cales é parcialmente complementaria coa seguinte. Así, poden formarse tres estruturas secundarias distintas (forquitas): 1–2, 2–3 ou 3–4. A hibridación das secuencias 1 e 2 para formar a estrutura 1–2 é rara porque a ARN polimerase espera a que o ribosoma se una antes de continuar a transcrición despois da secuencia 1, porén se se forma a forquita 1–2, isto impediría a formación da estrutura 2–3 (pero non a da 3–4). A formación dun bucle forquita entre as secuencias 2–3 impide a formación de bucles forquita entre 1–2 e 3–4. A estrutura 3–4 é unha secuencia de terminación da transcrición (rica en G/C e inmediatamente seguida de varios residuos de uracilo), unha vez que se forma a ARN polimerase disóciase do ADN e a transcrición do ADN dos xenes estruturais do operón non se pode producir (véxase máis adiante unha explicación máis detallada). A importancia funcional da segunda forquita para a terminación da transcrición queda ilustrada pola redución na frecuencia de terminación da transcrición observada en experimentos que desestabilizan o apareamento G+C central desta forquita.[5][7][8][9]
Parte do transcrito líder codifica un curto polipéptido de 14 aminoácidos denominado péptido líder. Este péptido contén dous residuos de triptófano adxacentes, o cal é pouco común, xa que o triptófano é un aminoácido bastante pouco frecuente (só un de cada cen residuos son de triptófano nunha proteína típica de Escherichia coli). A febra 1 do trpL comprende a rexión que codifica os residuos de cola do péptido líder: Trp, Trp, Arg, Thr, Ser;[2] observouse unha conservación nestes 5 codóns, mentres que se comprobou que mutar os codóns de augas arriba non altera a expresión do operón.[2][10][11][12] Se o ribosoma intenta traducir este péptido cando os niveis de triptófano na célula son baixos, quedará bloqueado nun dos dous codóns trp. Mentres está parado alí, o ribosoma cobre fisicamente a secuencia 1 do transcrito, impedindo a formación da estrutura secundaria 1–2. A secuencia 2 queda entón libre para hibridarse coa secuencia 3 e formar a estrutura 2–3, o cal imposibilita a formación da forquita de terminación 3–4, polo cal a estrutura 2–3 se chama forquita antiterminación. En presenza da estrutura 2–3, a ARN polimerase pode continuar trancribindo o operón. A análise mutacional e os estudos que usan oligonucleótidos complementarios demostran que a estabilidade da estrutura 2–3 se corresponde co nivel de expresión do operón.[10][13][14][15] Se os niveis de triptófano na célula son altos, o ribosoma traducirá o péptido líder completo sen interrupción e só se detén durante a terminación da tradución no codón de terminación. Nese punto o ribosoma cobre fisicamente as secuencias 1 e 2. As secuencias 3 e 4 quedan así libres para formar a estrutura 3–4 que termina a transcrición. Esta estrutura terminadora fórmase cando ningún ribosoma está detido na veciñanza do tándem de triptófanos (é dicir, un codón de Trp ou de Arg): ou o péptido líder non se traduce ou a tradución avanza de vagariño pola febra 1 con abundantes moléculas cargadas de triptófano de ARNttrp.[2][10] Ademais, propúxose que o ribosoma só bloquea uns 10 nucleótidos augas abaixo, así a detención do ribosoma, xa sexa na secuencia Gly ou xa sexa na Thr situada máis augas abaixo, non parece afectar a formación da forquita de terminación.[2][10] O resultado final é que operón se transcribirá soamente cando o ribosoma non dispón de triptófano, mentres que o transcrito trpL se expresa constitutivamente.
Este mecanismo de atenuación está apoiado experimentalmente. Primeiro, a tradución do péptido líder e a parada do ribosoma probaron ser directamente necesarios para inhibir a terminación da transcrición.[13] Ademais, a análise mutacional que desestabiliza ou altera o apareamento de bases da forquita antiterminador ten como resultado un incremento na terminación; isto é consistente co modelo de atenuación, esta mutación non pode aliviar a atenuación mesmo se hai escaseza de triptófano (Trp).[10][13] En contraste con isto, oligonucleótidos complementarios dirixidos á febra 1 aumentan a expresión do operón ao promocionaren a fomación do antiterminador.[10][14] Ademais, no operón da histidina, a mutación complementaria mostra que a capacidade de apareamento das febras 2–3 ten máis importancia que as súas secuencias primarias á hora de inhibir a atenuación.[10][15]
Na atenuación, o punto onde o ribosoma que está traducindo queda parado determina se se formará unha forquita de terminación.[10] Para que a polimerase en transcrición capture ao mesmo tempo a estrutura alternativa, a escala de tempo da modulación estrutural debe ser comparable coa da transcrición.[2] Para asegurar que o ribosoma se une e empeza a tradución do transcrito líder inmediatamente despois de que este se sintetiza, existe un sitio de pausa na secuencia trpLe. Unha vez chegado a este sitio, a ARN polimerase fai unha pausa na transcrición e aparentemente agarda a que empece a tradución. Este mecanismo posibilita unha sincronización da transcrición e da tradución, o cal é un elemento clave na atenuación.
Un mecanismo de atenuación similar regula as sínteses da histidina, fenilalanina e treonina.
Notas
editar- ↑ William Klug, Cummings, and Spencer. "Concepts of Genetics." 8th Ed. Pearson Education Inc, New Jersey: 2006. pg. 394-402
- ↑ 2,0 2,1 2,2 2,3 2,4 2,5 2,6 Yanofsky, C (1981). "Attenuation in the control of expression of bacterial operons". Nature 289 (5800): 751–758. Bibcode:1981Natur.289..751Y. PMID 7007895. doi:10.1038/289751a0.
- ↑ Lehninger, Albert L.; Nelson, David L.; Cox, Michael M. (2008). Principles of Biochemistry (5th ed.). New York, NY: W.H. Freeman and Company. p. 1128. ISBN 978-0-7167-7108-1.
- ↑ Bertrand, K; Squires, C; Yanofsky, C (May 15, 1976). "Transcription termination in vivo in the leader region of the tryptophan operon of Escherichia coli.". Journal of Molecular Biology 103 (2): 319–37. PMID 781269. doi:10.1016/0022-2836(76)90315-6.
- ↑ 5,0 5,1 Lee, F.; Yanofsky, C. (1977). "Transcription termination at the trp operon attenuators of Escherichia coli and Salmonella typhimurium: RNA secondary structure and regulation of termination". Proceedings of the National Academy of Sciences of the USA 74 (10): 4365–9. Bibcode:1977PNAS...74.4365L. PMC 431942. PMID 337297. doi:10.1073/pnas.74.10.4365.
- ↑ Oxender, D.; Zurawski, G.; Yanofsky, C. (1979). "Attenuation in the Escherichia coli tryptophan operon: role of RNA secondary structure involving the tryptophan codon region". Proceedings of the National Academy of Sciences of the USA 76 (11): 5524–5528. Bibcode:1979PNAS...76.5524O. PMC 411681. PMID 118451. doi:10.1073/pnas.76.11.5524.
- ↑ Stroynowski, I.; Yanofsky, C. (1982). "Transcript secondary structures regulate transcription termination at the attenuator of S. marcescens tryptophan operon". Nature 298 (5869): 34–38. Bibcode:1982Natur.298...34S. doi:10.1038/298034a0.
- ↑ Zurawski, G.; Yanofsky, C. (1980). "Escherichia coli tryptophan operon leader mutations, which relieve transcription termination, are cis-dominant to trp leader mutations, which increase transcription termination". Journal of Molecular Biology 142 (1): 123–9. doi:10.1016/0022-2836(80)90210-7.
- ↑ Stauffer, G. V.; Zurawski, G.; Yanofsky, C. (1978). "Single base-pair alterations in the Escherichia coli trp operon leader region that relieve transcription termination at the trp attenuator". Proceedings of the National Academy of Sciences of the USA 75 (10): 4833–7. Bibcode:1978PNAS...75.4833S. PMC 336215. PMID 368800. doi:10.1073/pnas.75.10.4833.
- ↑ 10,0 10,1 10,2 10,3 10,4 10,5 10,6 10,7 Kolter, R (1982). "Attenuation in amino acid biosynthetic operons". Annu Rev Genet 16: 113–34. PMID 6186194. doi:10.1146/annurev.ge.16.120182.000553.
- ↑ Lee, F.; Bertrand, K.; Bennett, G.; Yanofsky, C. (1978). "Comparison of the nucleotide sequences of the initial transcribed regions of the tryptophan operons of Escherichia coli and Salmonella typhimurium". Journal of Molecular Biology 121 (2): 193–217. doi:10.1016/s0022-2836(78)80005-9.
- ↑ Miozzari, G. F.; Yanofsky, C. (1978). "Naturally occurring promoter down mutation: Nucleotide sequence of the trp promoter/operator/leader region of Shigella dysenteriae 16". Proceedings of the National Academy of Sciences of the USA 75 (11): 5580–4. Bibcode:1978PNAS...75.5580M. PMC 393010. PMID 364484. doi:10.1073/pnas.75.11.5580.
- ↑ 13,0 13,1 13,2 Zurawski, G.; Elseviers, D.; Stauffer, G. V.; Yanofsky, C. (1978). "Translational control of transcription termination at the attenuator of the Escherichia coli tryptophan operon". Proceedings of the National Academy of Sciences of the USA 75 (12): 5988–92. Bibcode:1978PNAS...75.5988Z. PMC 393102. PMID 366606. doi:10.1073/pnas.75.12.5988.
- ↑ 14,0 14,1 Winkler, M. E.; Mullis, K.; Barnett, J.; Stroynowski, I.; Yanofsky, C. (1982). "Transcription termination at the tryptophan attenuator is decreased in vitro by an oligomer complementary to a segment of the leader transcript". Proceedings of the National Academy of Sciences of the USA 79 (7): 2181–5. PMC 346154. doi:10.1073/pnas.79.7.2181.
- ↑ 15,0 15,1 Johnston, H.; Roth, J. (1981). "DNA sequence changes of mutations altering attenuation control of the histidine operon of Salmonella typhimurium". Journal of Molecular Biology 145 (4): 735–56. PMID 6167727. doi:10.1016/0022-2836(81)90312-0.
Véxase tamén
editarBibliografía
editar- Morse, DE; Mosteller RD; Yanofsky C (1969). "Dynamics of synthesis, translation, and degradation of trp operon messenger RNA in E. coli.". Cold Spring Harb Symp Quant Biol. 34: 725–40. PMID 4909527. doi:10.1101/sqb.1969.034.01.082.
- Yanofsky, Charles (1981). "Attenuation in the control of expression of bacterial operons". Nature 289 (5800): 751–58. Bibcode:1981Natur.289..751Y. PMID 7007895. doi:10.1038/289751a0.