Fosfatidilinositol 5-fosfato
O fosfatidilinositol 5-fosfato (PtdIns5P ou PI5P) é un fosfoinosítido, un dos derivados fosforilados do fosfatidilinositol (PtdIns), que son moléculas reguladoras ancoradas na membrana. Os fosfoinosítidos participan en eventos de sinalización celular que controlan a dinámica do citoesqueleto, o tráfico de membranas intracelulares, a proliferación celular e moitas outras funcións celulares. Xeralmente, os fosfoinosítidos transducen sinais ao recrutar nas membranas intracelulares proteínas que se unen a fosfoinosítidos.[3] O PtdIns5P está fosforilado na posición D5 do grupo inositol, o cal está unido por enlace fosfodiéster ao diacilglicerol (que xeralmente leva ácidos graxos coa composición 1-estearoil-2-araquidonoil).
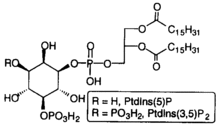
O fosfatidilinositol 5-fosfato é un dos fosfoinosítidos celulares do que se coñece peor as súas funcións. En células quiescentes, como media, o PtdIns5P ten unha abundancia similar á do PtdIns3P, pero ~20-100 veces menor que a do PtdIns4P e PtdIns(4,5)P2.[4] Os niveis de PtdIns5P son máis de 5 veces maiores que os do PtdIns(3,5)P2.[5][6]
A presenza de PtdIns5P demostrouse primeiro por cromatografía líquida de alta presión (HPLC) en fibroblastos de rato como un substrato para a síntese do PtdIns(4,5)P2 realizada pola PIP quinase de tipo II (1-fosfatidilinositol-5-fosfato 4-quinase).[7] Porén, en moitos tipos celulares, o PtdIns5P non se detecta por HPLC debido a limitacións técnicas asociadas coa súa mala separación do máis abondoso PtdIns4P.[8] Neses casos o PtdIns5P mídese por "ensaio de masas", no que o PtdIns5P (como parte dos lípidos celulares extraídos) é convertido in vitro pola PtdIns5P 4-quinase purificada en PtdIns(4,5)P2, o cal é seguidamente cuantificado.[9]
Baseándose en estudos con ensaio de masas[8] e unha técnica mellorada de HPLC,[10] o PtdIns5P detéctase en todas as células animais estudadas. A maioría do PtdIns5P celular encóntrase nas membranas citoplasmáticas, mentres que unha pequena fracción reside no núcleo.[11] Os conxuntos citoplásmico e nuclear teñen distintas funcións e regulación.[12]
Metabolismo
editarO PtdIns5P celular pode producirse por fosforilación en D5 do fosfatidilinositol ou por desfosforilación do PtdIns(3,5)P2 ou do PtdIns(4,5)P2. Todas estas posibilidades teñen apoio experimental. O PtdIns5P sintetízao in vitro o encima PIKfyve, un encima responsable principalmente da produción de PtdIns(3,5)P2,[13][14] e tamén os encimas PIP5Ks.[15] A importancia de PIKfyve na síntese de PtdIns5P celulares indícana os datos de niveis de masa de PtdIns5P reducidos atopados despois da sobreexpresión heteróloga do mutante puntual para PIKfyve inactiva encimaticamente (PIKfyveK1831E)[8][16] e do silenciamento de PIKfyve por ARNs interferentes pequenos.[17] Este papel está reforzado por datos de fibroblastos transxénicos cun alelo PIKfyve alterado xeneticamente, que demostran unha redución igual dos niveis de estado estacionario de PtdIns5P e PtdIns(3,5)P2. [5]
Igualmente, atopouse unha redución similar do PtdIns5P e o PtdIns(3,5)P2 en fibroblastos con knockout do activador PIKfyve[18] ArPIKfyve/VAC14.[6] Estas probas experimentais xunto co feito de que os niveis celulares de PtdIns5P son máis de 5 veces superiores aos de PtdIns(3,5)P2 indican un papel predminante de PIKfyve no mantemento do estado estacionario dos niveis de PtdIns5P por medio da fosforilación D5 do fosfatidilinositol.
Propúxose tamén que a familia de proteínas da miotubularina exerce un papel na produción de PtdIns5P baseándose na desfosforilación de PtdIns(3,5)P2 pola miotubularina 1 sobreexpresada. [19] En concordancia co anterior, a ablación xenética da proteína 2 relacionada coa miotubularina (MTMR2) causa a elevación das PtdIns(3,5)P2 celulares e un descenso do PtdIns5P.[20] Os baixos niveis celulares de PtdIns(3,5)P2 suxiren que a actividade da miotubularina fosfatase xoga un papel menor no mantemento do estado estacionario dos niveis de PtdIns5P. O PtdIns(3,5)P2 é sintetizado a partir de PtdIns3P polo complexo PIKfyve, que inclúe a ArPIKfyve e a Sac3/Fig4.[21] O complexo PIKfyve intervén na síntese e recambio de PtdIns(3,5)P2 a partir de PtdIns3P. [22] Non se coñece a proporción relativa de recambio de PtdIns(3,5)P2 feito polas fosfatases miotubularinas en comparación co realizado por Sac3.
O PtdIns5P pode tamén producirse por desfosforilación de PtdIns(4,5)P2. Esta actividade fosfatase móstrase por medio do efector de Shigella flexneri IpgD[23] e de dúas fosfatases de mamífero: as PtdIns(4,5)P2 4-fosfatases tipo I e tipo II.[24]
Actualmente, non se coñece ningunha fosfatase de mamífero que desfosforile especificamente o PtdIns5P. A vía para a eliminación do PtdIns5P implica a síntese de PtdIns(4,5)P2.[12]
Funcións
editarOs niveis de PtdIns5P cambian significativamente en resposta de estímulos fisiolóxicos e patolóxicos. A insulina,[10][25] a trombina,[9] a activación de células T,[26] e a transformación de células coa tirosina quinase de linfoma anaplástico nucleofosmina (NPM-ALK),[17] causa a elevación dos niveis de PtdIns5P celulares. Ao contrario, o shock hipoosmótico[8] e o tratamento con histamina[27] fan descender os niveis de PtdIns5P. En células T, propuxéronse dúas proteínas “tirosina quinases de augas abaixo” chamadas DOK1 e DOK2 como proteínas que se unen ao PtdIns5P e son efectores.[26]
Igual que os outros fosfoinosítidos, o PtdIns5P está tamén presente no núcleo das células de mamíferos.[28] O PtdIns5P nuclear é controlado pola PtdIns(4,5)P2 4-fosfatase de tipo I nuclear, a cal, en conxunción coa quinase PIPKIIbeta, xoga un papel no estrés por luz ultravioleta, a apoptose e a progresión do ciclo celular.[11][29][30]
Na función do PtdIns5P na sinalización nuclear probablemente está implicada ING2, un membro da familia proteica ING. As proteínas desta familia asócianse con acetilases e desacetilases de histonas e modulan a súa actividade, e tamén inducen a apoptose por medio da acetilación de p53. A proteína ING2 interacciona co PtdIns5P por medio do seu motivo dedo homeodominio de planta (PHD). [31]
En resumo, as probas dispoñibles indican que a actividade PIKfyve é a principal fonte de PtdIns5P celular no estado estaacionario. En certas condicións, o PtdIns5P prodúcese por desfosforilación de bis-fosfoinosítidos. O PtdIns5P está envolvido na regulación de funcións celulares básicas e de respostas a unha multitude de estímulos fisiolóxicos e patolóxicos por medio de mecanismos que aínda non foron especificados.
Notas
editar- ↑ PubChem - Phosphatidylinositol 5-phosphate
- ↑ CHEBI - 1-phosphatidyl-1D-myo-inositol 5-phosphate (CHEBI:16500)
- ↑ Di Paolo G, De Camilli P. Phosphoinositides in cell regulation and membrane dynamics. Nature. 2006 Oct 12;443(7112):651-7. PMID 17035995
- ↑ Shisheva A. Regulating Glut4 vesicle dynamics by phosphoinositide kinases and phosphoinositide phosphatases. Front Biosci. 2003 Sep 1;8:s945-6. Review. PMID 12957825
- ↑ 5,0 5,1 Ikonomov OC, Sbrissa D, Delvecchio K, Xie Y, Jin JP, Rappolee D, Shisheva A. The phosphoinositide kinase PIKfyve is vital in early embryonic development: preimplantation lethality of PIKfyve-/- embryos but normality of PIKfyve+/- mice. J Biol Chem. 2011 Apr 15;286(15):13404-13. Epub 2011 Feb 24. PMID 21349843
- ↑ 6,0 6,1 Zhang Y, Zolov SN, Chow CY, Slutsky SG, Richardson SC, Piper RC, Yang B, Nau JJ, Westrick RJ, Morrison SJ, Meisler MH, WeismanLS. Loss of Vac14, a regulator of the signaling lipid phosphatidylinositol 3,5-bisphosphate, results in neurodegeneration in mice. Proc Natl Acad Sci U S A. 2007 Oct 30;104(44):17518-23. Epub 2007 Oct 23. PMID 17956977
- ↑ Rameh LE, Tolias K, Duckworth BC, Cantley LC. A new pathway for synthesis of phosphatydilinositol-4,5-bisphosphate. Nature. 1997 Nov 13;390(6656):192-6. PMID 9367159
- ↑ 8,0 8,1 8,2 8,3 Sbrissa D, Ikonomov OC, Deeb R, Shisheva A. Phosphatidylinositol 5-phosphate biosynthesis is linked to PIKfyve and is involved in osmotic response pathway in mammalian cells. J Biol Chem. 2002 Dec 6;277(49):47276-84. Epub 2002 Sep 20. PMID 12270933
- ↑ 9,0 9,1 Morris JB, Hinchliffe KA, Ciruela A, Letcher AJ, Irvine RF. Thrombin stimulation of platelets causes an increase in phosphatydilinositol 5-phosphate revealed by mass assay. FEBS Lett. 2000 Jun 9;475(1):57-60. PMID 10854858
- ↑ 10,0 10,1 Sarkes D, Rameh LE. A novel HPLC-based approach makes possible the spatial characterization of cellular PtdIns5P and other phosphoinositides. Biochem J. 2010 May 27;428(3):375-84. PMID 20370717
- ↑ 11,0 11,1 Zou J, Marjanovic J, Kisseleva MV, Wilson M, Majerus PW. Type I phosphatidylinositol-4,5-bisphosphate 4-phosphatase regulates stress-induced apoptosis. Proc Natl Acad Sci U S A. 2007 Oct 23;104(43):16834-9. Epub 2007 Oct 16. PMID 17940011
- ↑ 12,0 12,1 Grainger DL, Tavelis C, Ryan AJ, Hinchliffe KA. The emerging role of PtdIns5P: another signalling phosphoinositide takes its place. Biochem Soc Trans. 2012 Feb 1;40(1):257-61. PMID 22260701
- ↑ Sbrissa D, Ikonomov OC, Shisheva A. PIKfyve, a mammalian ortholog of yeast Fab1p lipid kinase, synthesizes 5-phosphoinositides. Effect of insulin. J Biol Chem. 1999 Jul 30;274(31):21589-97. PMID 10419465
- ↑ Shisheva A. PIKfyve: the road to PtdIns 5-P and PtdIns 3,5-P(2). Cell Biol Int. 2001;25(12):1201-6. PMID 11748912.
- ↑ Tolias KF, Rameh LE, Ishihara H, Shibasaki Y, Chen J, Prestwich GD, Cantley LC, Carpenter CL. Type I phosphatidylinositol-4-phosphate 5-kinases synthesize the novel lipids phosphatidylinositol 3,5-bisphosphate and phosphatidylinositol 5-phosphate. J Biol Chem. 1998 Jul 17;273(29):18040-6. PMID 9660759
- ↑ Shisheva A. PIKfyve: Partners, significance, debates and paradoxes. Cell Biol Int. 2008 Jun;32(6):591-604. Epub 2008 Jan 25. Review.PMID 18304842
- ↑ 17,0 17,1 Coronas S, Lagarrigue F, Ramel D, Chicanne G, Delsol G, Payrastre B, Tronchère H. Elevated levels of PtdIns5P in NPM-ALK transformed cells: implication of PIKfyve. Biochem Biophys Res Commun. 2008 Jul 25;372(2):351-5. Epub 2008 May 22. PMID 18501703
- ↑ Sbrissa D, Ikonomov OC, Strakova J, Dondapati R, Mlak K, Deeb R, Silver R, Shisheva A. A mammalian ortholog of Saccharomyces cerevisiae Vac14 that associates with and up-regulates PIKfyve phosphoinositide 5-kinase activity. Sbrissa D, Ikonomov OC, Strakova J, Dondapati R, Mlak K, Deeb R, Silver R, Shisheva A. Mol Cell Biol. 2004 Dec;24(23):10437-47.PMID 15542851
- ↑ Tronchère H, Laporte J, Pendaries C, Chaussade C, Liaubet L, Pirola L, Mandel JL, Payrastre B. Production of phosphatidylinositol 5-phosphate by the phosphoinositide 3-phosphatase myotubularin in mammalian cells. J Biol Chem. 2004 Feb 20;279(8):7304-12. Epub 2003 Dec 1. PMID 14660569
- ↑ Vaccari I, Dina G, Tronchère H, Kaufman E, Chicanne G, Cerri F, Wrabetz L, Payrastre B, Quattrini A, Weisman LS, Meisler MH, Bolino A. Genetic interaction between MTMR2 and FIG4 phospholipid phosphatases involved in Charcot-Marie-Tooth neuropathies. PLoS Genet. 2011 Oct;7(10):e1002319. Epub 2011 Oct 20. PMID 22028665
- ↑ Sbrissa D, Ikonomov OC, Fu Z, Ijuin T, Gruenberg J, Takenawa T, Shisheva A. Core protein machinery for mammalian phosphatidylinositol 3,5-bisphosphate synthesis and turnover that regulates the progression of endosomal transport. Novel Sac phosphatase joins the ArPIKfyve-PIKfyve complex. J Biol Chem. 2007 Aug 17;282(33):23878-91. Epub 2007 Jun 7. PMID 17556371
- ↑ Ikonomov OC, Sbrissa D, Fenner H, Shisheva A. PIKfyve-ArPIKfyve-Sac3 core complex: contact sites and their consequence for Sac3 phosphatase activity and endocytic membrane homeostasis. J Biol Chem. 2009 Dec 18;284(51):35794-806. PMID 19840946
- ↑ Niebuhr K, Giuriato S, Pedron T, Philpott DJ, Gaits F, Sable J, Sheetz MP, Parsot C, Sansonetti PJ, Payrastre B. Conversion of PtdIns(4,5)P(2) into PtdIns(5)P by the S.flexneri effector IpgD reorganizes host cell morphology. EMBO J. 2002 Oct 1;21(19):5069-78. PMID 12356723
- ↑ Ungewickell A, Hugge C, Kisseleva M, Chang SC, Zou J, Feng Y, Galyov EE, Wilson M, Majerus PW. The identification and characterization of two phosphatidylinositol-4,5-bisphosphate 4-phosphatases. Proc Natl Acad Sci U S A. 2005 Dec 27;102(52):18854-9. Epub 2005 Dec 19. PMID 16365287
- ↑ Sbrissa D, Ikonomov OC, Strakova J, Shisheva A. Role for a novel signaling intermediate, phosphatidylinositol 5-phosphate, in insulin-regulated F-actin stress fiber breakdown and GLUT4 translocation. Endocrinology. 2004 Nov;145(11):4853-65. Epub 2004 Jul 29. PMID 15284192.
- ↑ 26,0 26,1 Guittard G, Gérard A, Dupuis-Coronas S, Tronchère H, Mortier E, Favre C, Olive D, Zimmermann P, Payrastre B, Nunès JA. Cutting edge: Dok-1 and Dok-2 adaptor molecules are regulated by phosphatidylinositol 5-phosphate production in T cells. J Immunol. 2009 Apr 1;182(7):3974-8. PMID 19299694
- ↑ Roberts HF, Clarke JH, Letcher AJ, Irvine RF, Hinchliffe KA. Effects of lipid kinase expression and cellular stimuli on phosphatidylinositol 5-phosphate levels in mammalian cell lines. FEBS Lett. 2005 May 23;579(13):2868-72. PMID 15876433
- ↑ Barlow CA, Laishram RS, Anderson RA. Nuclear phosphoinositides: a signaling enigma wrapped in a compartmental conundrum. Trends Cell Biol. 2010 Jan;20(1):25-35. Epub 2009 Oct 19. PMID 19846310.
- ↑ Clarke JH, Letcher AJ, D'santos CS, Halstead JR, Irvine RF, Divecha N. Inositol lipids are regulated during cell cycle progression in the nuclei of murine erythroleukaemia cells. Biochem J. 2001 Aug 1;357(Pt 3):905-10. PMID 11463365
- ↑ Jones DR, Bultsma Y, Keune WJ, Halstead JR, Elouarrat D, Mohammed S, Heck AJ, D'Santos CS, Divecha N. Nuclear PtdIns5P as a transducer of stress signaling: an in vivo role for PIP4Kbeta. Mol Cell. 2006 Sep 1;23(5):685-95. PMID 16949365
- ↑ Gozani O, Karuman P, Jones DR, Ivanov D, Cha J, Lugovskoy AA, Baird CL, Zhu H, Field SJ, Lessnick SL, Villasenor J, Mehrotra B, Chen J, Rao VR, Brugge JS, Ferguson CG, Payrastre B, Myszka DG, Cantley LC, Wagner G, Divecha N, Prestwich GD, Yuan J. The PHD finger of the chromatin-associated protein ING2 functions as a nuclear phosphoinositide receptor. Cell. 2003 Jul 11;114(1):99-111. PMID 12859901