Reparación por escisión de nucleótidos
A reparación por escisión de nucleótidos (abreviada como NER, do inglés nucleotide excision repair) é un mecanismo de reparación do ADN.[2] Os danos no ADN ocorren constantemente causados pola exposición a certos compostos químicos, como por exemplo, axentes intercalantes), radiación e outros mutáxenos. Existen tres vías de reparación por escisión para reparar os danos no ADN de febra simple, que son: reparación por escisión de nucleótidos (NER), reparación por escisión de bases (BER), e reparación de discordancias no ADN (MMR). Aínda que a vía BER pode recoñecer lesións non voluminosas específicas no ADN, só pode corrixir as bases danadas que son retiradas por glicosidases específicas. De xeito similar, a vía de reparación de discordancias só vai dirixida a pares de bases de Watson e Crick.
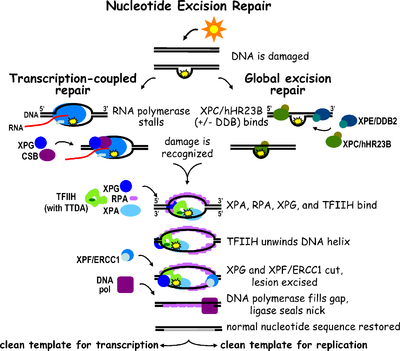
A reparación por escisión de nucleótidos (NER) é un mecanismo de escisión especialmente importante que elimina os danos no ADN inducidos pola luz ultravioleta (UV). Os danos no ADN por luz UV dan lugar a adutos do ADN voluminosos; estes adutos son principalmente dímeros de timina e 6,4-fotoprodutos. O recoñecemento dos danos orixina a eliminación dun curto segmento de ADN de febra simple que contén a lesión. A febra simple do ADN que non sufriu danos permanece no seu lugar e a ADN polimerase utilízaa como molde para sintetizar unha curta secuencia complementaria. A ligazón final coa que se completa a NER e se forma un ADN de dobre febra lévaa a cabo a ADN ligase. A NER pode dividirse en dúas subvías: NER xenómica global (GG-NER ou GGR) e NER acoplada á transcrición (TC-NER ou TCR). As dúas subvías difiren no xeito en que recoñecen danos no ADN, pero teñen en común o mesmo proceso para a incisión, reparación e ligazón da lesión.
A importancia da NER evidénciase polas graves doenzas humanas que se orixinan en mutacións xenéticas conxénitas que afectan ás proteínas da NER. O xeroderma pigmentoso e a síndrome de Cockayne son dous exemplos de doenzas asociadas á NER.
Reparación por escisión de nucleótidos en eucariotas
editarA reparación por escisión de nucleótidos é máis complexo en eucariotas que en procariotas, pero o principio xeral é similar. Hai 9 proteínas princiapis implicadas na NER en células de mamíferos. En deficiencias en certas proteínas que orixinan doenzas, os nomes das proteínas están asociados cos das doenzas. As denominacións das proteínas XPA, XPB, XPC, XPD, XPE, XPF, e XPG derivan de хeroderma pigmentoso, mentres que CSA e CSB son proteínas ligadas á síndrome de Cockayne. Adicionalmente, as proteínas ERCC1, RPA, RAD23A, RAD23B e outras participan tamén na reparación por escisión de nucleótidos. Unha lista máis completa de proteínas implicadas na NER pode consultarase máis abaixo.
A reparación por escisión de nucleótidos pode dividirse en dúas subvías: NER xenómica global (GG-NER) e NER acoplada á transcrición (TC-NER). Interveñen tres conxuntos distintos de proteínas no recoñecemento de danos no ADN en cada subvía. Despois do recoñecemento de danos, que é distinto en cada unha, as subvías converxen nos pasos da incisión dual, reparación e ligazón.
Recoñecemento dos danos
editarNER xenómica global (GG-NER)
editarA NER xenómica global repara os danos tanto na febra do ADN que se transcribe coma na que non se transcribe en xenes activos e inactivos de todo o xenoma. Este proceso non depende da transcrición. Esta vía emprega varias proteínas que "detectan o dano" como o complexo que se une aos danos do ADN (DDB) e o XPC-Rad23B, que constantemente revisan o xenoma e recoñecen distorsións da hélice: o complexo XPC-Rad23B é responsable do recoñecemento da distorsión, mentres que DDB1 e DDB2 (XPE) poden tamén recoñecer algúns tipos de danos causados pola luz UV. Ademais, a XPA realiza unha función de recoñecemento de danos que polo momento non foi ben definida. Despois da identificación dos sitios danados, recrútanse despois no ADN danado as seguintes proteínas de reparación para verificar a presenza de danos, escindir o ADN danado arredor da lesión e despois encher o parche da reparación.
Doenzas asociadas á GG-NER
editarAs mutacións na maquinaria da GG-NER son responsables de moitos trastornos xenéticos como: xeroderma pigmentoso (XP): fotosensibilidade grave, altas taxas de cancro en áreas do corpo expostas ao sol, como a pel.
Reparación acoplada á transcrición (TC-NER)
editarEn calquera momento dado, a maioría do xenoma nun organismo non está realizando a transcrición. Hai unha diferenza na eficiencia da NER entre as rexións do xenoma transcricionalmente silenciosas e as activas. Para moitos tipos de lesións, a NER repara máis rápido as febras transcritas de xenes activos transcricionalmente que as febras non transcritas e o ADN transcricionalmente silencioso.
A TC-NER e a GG-NER difiren só nos pasos iniciais do recoñecemento dos danos no ADN. A diferenza principal entre a TC-NER e a GG-NER é que a TC-NER non require proteínas XPC ou DDB para o recoñecemento da distorsión en células de mamíferos. En lugar diso, a TC-NER iníciasee cando a ARN polimerase queda atascada nunha lesión no ADN: a ARN polimerase bloqueada serve como un sinal de recoñecemento dos danos, que substitúe a necesidade de ter os complexos XPC-RAD23B e DDB con propiedades de recoñecemento de distorsións. As proteínas CS (CSA e CSB) únense a algúns tipos de danos no ADN en lugar de XPC-Rad23B.
Son posibles outros mecanismos de reparación, pero son menos exactos e eficientes.
Doenzas asociadas á TC-NER
editarA TC-NER iníciase cando a ARN polimerase queda atascada ao chegar a unha lesión no ADN e uns complexos proteicos axudan á polimerase a retroceder. As mutacións que afectan á maquinaria da TC-NER son responsables de múltiples trastornos xenéticos como:
- Tricotiodistrofia: algúns individuos son fotosensibles, ictiose, retardo mental/físico.
- Síndrome de Cockayne: fotosensibilidade, atraso mental, características similares á proxeria, microcefalia.
Incisión dual
editarO encima clave que intervén na escisión dual é o factor de transcrición II H (TFIIH). Primeiramente, recrútanse o TFIIH e a XPG no sitio dos danos no ADN (XPG estabiliza a TFIIH). As subunidades TFIIH de XPD e XPB actúan como helicases 5'-3' e 3'-5', respectivamente, e axudan a desenrolar o ADN e xerar unha unión entre o ADN de dobre febra e o de febra simple arredor da burbulla de transcrición. Ademais de estabilizar a TFIIH, a XPG tamén ten unha actividade de endonuclease; corta os danos no ADN no lado 3' mentres que a proteína heterodímera XPF–ERCC1 corta no lado 5'. A incisión dual causa a eliminación dun ADN monocatenario cun oco de febra simple de 25 a ~30 nucleótidos. Os oligonucleótidos de ADN que contén o dano, escindido pequeno (sedDNA, do inglés small excised damage-containing DNA) son inicialmente liberados do dúplex en complexo co TFIIH pero despois disócianse de maneira dependente do ATP e únense á RPA. A inhibición da síntese de ADN de recheo de ocos e a ligazón dá lugar a unha acumulación na célula de sedDNAs unidos a RPA.
A proteína de replicación A (RPA) e XPA son as últimas dúas proteínas asociadas co principal complexo de reparación da NER. Estas dúas proteínas están presentes xa antes da unión de TFIIH, xa que interveñen na verificación dos danos no ADN. Poden tamén protexer o ADN de febra simple. Despois da verificación, faise a incisión no lado 5' e empeza a reparación do ADN antes da incisión do lado 3'. Isto axuda a reducir o ADN de febra simple que queda exposto durante o proceso de reparación.
Reparación e ligazón
editarO factor de replicación C (RFC) carga o antíxeno nuclear de célula proliferante (PCNA) sobre a febra de ADN. Isto permite que a ADN polimerases implicada na reparación (δ, ε e/ou κ) copie as febras non danadas por medio de translocación. A ADN ligase I e a endonuclease flap 1 ou o complexo ligase-III-XRCC1 selan a amosega para completar a NER.
A NER en procariotas: proteínas Uvr
editar- Véxase tamén: Endonuclease UvrABC.
O proceso da reparación por escisión de nucleótidos é controlado en Escherichia coli polo complexo encimático da endonuclease UvrABC, que consta de catro proteínas Uvr: UvrA, UvrB, UvrC, e a ADN helicase II (ás veces chamada UvrD neste complexo). Primeiro, un complexo UvrA-UvrB fai unha revisión do ADN, na cal a subunidade UvrA é a encargada de recoñecer as distorsións na hélice, causadas por exemplo por dímeros de pirimidina. Cando o complexo recoñece ditas distorsións, a subunidade UvrA sae do complexo e despois chega unha proteína UvrC, que se une ao monómero UvrB e forma así un novo dímero UvrBC. A UvrB cliva un enlace fosfodiéster situado a 4 nucleótidos augas abaixo do lugar do dano no ADN, e a UvrC, pola súa parte, cliva un enlace fosfodiéster situado 8 nucleótidos augas arriba, creándose un segmento escindido de 12 nucleótidos. A ADN helicase II (ou UvrD) chega despois e elimina o segmento escindido ao romper activamente as pontes de hidróxeno entre as bases complementarias. O oco resultante énchese despois usando a ADN polimerase I e a ADN ligase. O proceso básico de escisión é moi similar en células eucariotas, pero nestas células xeralmente interveñen máis proteínas. O que ocorre en E. coli é un exemplo simple.[5]
A modalidade da TC-NER tamén existe en bacterias, e realízase coa mediación da proteína TRCF (Mfd). A TRCF é unha ATPase SF2 que usa a hidrólise do ATP para translocarse sobre o ADN de dobre febra augas arriba da burbulla de transcrición e translocar cara a adiante a ARN polimerase, iniciando así a disociación do complexo de elongación ternario da ARN polimerase. A TRCF tamén recruta a maquinaria de reparación de escisión de nucleótidos Uvr(A)BC por interacción física directa coa subunidade UvrA.
A NER e o cancro
editarAínda que en estudos históricos se obtiveron resultados contraditorios, a variación xenética ou a mutación nos xenes da reparación por escisión de nucleótidos poden ten un impacto sobre o risco de contraer cancro ao afectaren á eficacia da reparación. Os polimorfismos un só nucleótido (SNPs) e os SNPs codificantes non sinónimos (SNPns) están presentes a moi baixos niveis (<1%) na poboación humana.[7] Se están localizados en xenes da NER ou secuencias regulatorias, tales mutacións poden afectar negativamente á capacidade de reparación do ADN o que ten como resultado un incremento da probabilidade de que se orixine un cancro. Aínda que non está caracterizado o impacto funcional de todos os polimorfismos, sábese que algúns polimorfismos nos xenes de reparación do ADN ou das súas secuencias regulatorias inducen cambios fenotípicos e están implicados no desenvolvemento do cancro.[8] Un estudo de casos de cancro de pulmón encontrou unha feble asociación entre os polimorfismos SNP específicos de NER e o risco de cancro de pulmón.[9] Os resultados indican que algunhas variacións polimórficas herdadas en xenes da NER causan predisposición ao cancro de pulmón e potencialmente a outros cancros.
A disfunción da NER orixina polimorfismos no ADN
editarDous xenes importantes na vía da NER nos que se atopou que os seus polimorfismos tiñan un impacto funcional e fenotípico son os xenes XPD e XPC.[10] O XPD, tamén chamado ERCC2, serve para abrir o ADN arredor do sitio dos danos durante a NER, ademais doutras actividades transcricionais. Os estudos mostraron que os polimorfismos no exón 10 (G>A)(Asp312Asn) e no exón 23 (A>T)(Lys751Gln) están ligados coa predisposición xenética a varios tipos e cancro.[11][12] O xene XPC é responsable da codificación dunha proteína que recoñece o ADN durante a porción inicial da vía NER. Este xene pode ter polimorfismos no intrón 9 e SNPs no exón 15, que tamén foron correlacionados co risco de cancro. Un polimorfismo de inserción/deleción poli (AT) bialélico no intrón 9 do xene XPC está asociado cun incremento dos cancros de pel, mama e próstata,[12][13][14] especialmente en poboacións do norte da India.
Impacto sobre o prognóstico do cancro
editarO estudo do cancro hereditario xeroderma pigmentoso (XP) axudou a identificar varios xenes que codificaban proteínas na vía da NER, dúas das cales son XPC e XPD. O xeroderma pigmentoso está causado por unha deficiencia homocigota que afecta á reparación de danos no ADN por UV (TC-NER), que multiplica por 1000 o risco do paciente de ter cancro de pel. En pacientes heterocigotos, o risco de cancro é esporádico mais pode predicirse baseándose nunha estimación analítica dos polimorfismos no xeroderma pigmentoso relacionados con xenes de reparación do ADN purificados de linfocitos.[15] Nun estudo as taxas de recaída de cancros colorrectais en estadios II e III de alto risco, o polimorfismo 2251A>C en XPD (ERCC2) estaba correlacionado significativamente coa recaída temperá despois dun tratamento de quimioterapia.[16] Os efectos dos xenes da NER polimórficos son aditivos, e canta maior frecuencia de variantes, maior risco de cancro hai.[15][16][17]
Xenes asociados á NER
editar| Xene humano (proteína) | Ortólogo no rato | Ortólogo no lévedo | Subvía | Función na NER | Entrada en GeneCards |
|---|---|---|---|---|---|
| CCNH (ciclina H) | Ccnh | CCL1 | Ambas | Subunidade quinase activadora de CDK (CAK) | CCNH |
| CDK7 (quinase dependente de ciclina (CDK) 7)) | Cdk7 | KIN28 | Ambas | Subunidade CAK | CDK7 |
| CETN2 (centrina-2) | Cetn2 | Descoñecido | GGR | Recoñecemento do dano; forma un complexo con XPC | CETN2 |
| DDB1 (DDB1) | Ddb1 | Descoñecido | GGR | Recoñecemento do dano; forma un complexo con DDB2 | DDB1 |
| DDB2 (DDB2) | Ddb2/Xpe | Descoñecdio | GGR | Recoñecemento do dano; recruta XPC | DDB2 |
| ERCC1 (ERCC1) | Ercc1 | RAD10 | Ambas | Implicada na escisión no lado 3' do dano; forma un complexo con XPF | ERCC1 |
| ERCC2 (XPD) | Ercc2 | RAD3 | Ambas | Actividade de ATPase e helicase; subunidade do factor de transcrición II H (TFIIH) | ERCC2 |
| ERCC3 (XPB) | Ercc3 | RAD25 | Ambas | Actividade de ATPase e helicase; subunidade do factor de transcrición II H (TFIIH) | ERCC3 |
| ERCC4 (XPF) | Ercc4 | RAD1 | Ambas | Implicada na incisión no lado 3' do dano; endonuclease específica de estrutura | ERCC4 |
| ERCC5 (XPG) | Ercc5 | RAD2 | Ambas | Implicada na incisión no lado 5' do dano; estabiliza a TFIIH; enconuclease específica de estrutura | ERCC5 |
| ERCC6 (CSB) | Ercc6 | RAD26 | TC-NER | Factor de elongación da transcrición; implicado no acoplamento da transcrición e a remodelación da cromatina | ERCC6 |
| ERCC8 (CSA) | Ercc8 | RAD28 | TC-NER | Complexo da ubiquitina ligase; interacciona con CSB e p44 de TFIIH | ERCC8 |
| LIG1 (ADN ligase I) | Lig1 | CDC9 | Ambas | Ligación final | LIG1 |
| MNAT1 (MNAT1) | Mnat1 | TFB3 | Ambas | Estabiliza o complexo CAK | MNAT1 |
| MMS19 (MMS19) | Mms19 | MET18 | Ambas | Interacciona coas subunidades XPD e XPB das helicases TFIIH | MMS19 |
| RAD23A (RAD23A) | Rad23a | RAD23 | GGR | Recoñecemento dos danos; forma un complexo con XPC | RAD23A |
| RAD23B (RAD23B) | Rad23b | RAD23 | GGR | Recoñecementodos danos, forma un complexo con XPC | RAD23B |
| RPA1 (RPA1) | Rpa1 | RFA1 | Ambas | Subunidade do complexo RFA | RPA1 |
| RPA2 (RPA2) | Rpa2 | RFA2 | Ambas | Subunidade do complexo RFA | RPA2 |
| TFIIH (factor de transcrición II H) | Descoñecido | Descoñecido | Ambas | Implicada na incisión, forma un complexo arredor da lesión | Sen GeneCards |
| XAB2 (XAB2) | Xab2 | SYF1 | TC-NER | Recoñecemento de danos; interacciona con XPA, CSA, e CSB | XAB2 |
| XPA (XPA) | Xpa | RAD14 | Ambas | Recoñecemento de danos | XPA |
| XPC (XPC) | Xpc | RAD4 | GGR | Recoñecemento de danos | XPC |
Notas
editar- ↑ Fuss JO, Cooper PK (June 2006). "DNA repair: dynamic defenders against cancer and aging". PLoS Biol. 4 (6): e203. PMC 1475692. PMID 16752948. doi:10.1371/journal.pbio.0040203.
- ↑ Carroll SB; Wessler SR; Griffiths AJFl; Lewontin RC (2008). Introduction to genetic analysis. New York: W.H. Freeman and CO. pp. 534. ISBN 0-7167-6887-9.
- ↑ 3,0 3,1 Le May N, Egly JM, Coin F (2010). "True lies: the double life of the nucleotide excision repair factors in transcription and DNA repair". J Nucleic Acids 2010. PMC 2915888. PMID 20725631. doi:10.4061/2010/616342.
- ↑ Morita R, Nakane S, Shimada A, et al. (2010). "Molecular mechanisms of the whole DNA repair system: a comparison of bacterial and eukaryotic systems". J Nucleic Acids 2010: 179594. PMC 2957137. PMID 20981145. doi:10.4061/2010/179594.
- ↑ Truglio JJ, Croteau DL, Van Houten B, Kisker C (February 2006). "Prokaryotic nucleotide excision repair: the UvrABC system". Chem. Rev. 106 (2): 233–52. PMID 16464004. doi:10.1021/cr040471u.
- ↑ Zhang Y, Rohde LH, Wu H (June 2009). "Involvement of nucleotide excision and mismatch repair mechanisms in double strand break repair". Curr. Genomics 10 (4): 250–8. PMC 2709936. PMID 19949546. doi:10.2174/138920209788488544.
- ↑ Kwok PY, Gu Z (December 1999). "Single nucleotide polymorphism libraries: why and how are we building them?". Mol Med Today 5 (12): 538–43. PMID 10562720. doi:10.1016/S1357-4310(99)01601-9.
- ↑ Karahalil B, Bohr V, Wilson D (October 2012). "Impact of DNA polymorphisms in key DNA base excision repair proteins on cancer risk". Hum Exp Toxicol 31 (10): 981–1005. PMID 23023028. doi:10.1177/0960327112444476.
- ↑ Sakoda LC, Loomis MM, Doherty JA, Julianto L, Barnett MJ, Neuhouser ML, Thornquist MD, Weiss NS, Goodman GE, Chen C (2012). "Germ line variation in nucleotide excision repair genes and lung cancer risk in smokers". Int J Mol Epidemiol Genet 3 (1): 1–17. PMC 3316453. PMID 22493747.
- ↑ Hou SM, Fält S, Angelini S, Yang K, Nyberg F, Lambert B, Hemminki K (April 2002). "The XPD variant alleles are associated with increased aromatic DNA adduct level and lung cancer risk". Carcinogenesis 23 (4): 599–603. PMID 11960912. doi:10.1093/carcin/23.4.599.
- ↑ Wang M, Gu D, Zhang Z, Zhou J, Zhang Z (2009). "XPD polymorphisms, cigarette smoking, and bladder cancer risk: a meta-analysis". J. Toxicol. Environ. Health Part A 72 (11–12): 698–705. PMID 19492231. doi:10.1080/15287390902841029.
- ↑ 12,0 12,1 Mittal RD, Mandal RK (January 2012). "Genetic variation in nucleotide excision repair pathway genes influence prostate and bladder cancer susceptibility in North Indian population". Indian J Hum Genet 18 (1): 47–55. PMC 3385179. PMID 22754221. doi:10.4103/0971-6866.96648.
- ↑ Blankenburg S, König IR, Moessner R, Laspe P, Thoms KM, Krueger U, Khan SG, Westphal G, Berking C, Volkenandt M, Reich K, Neumann C, Ziegler A, Kraemer KH, Emmert S (June 2005). "Assessment of 3 xeroderma pigmentosum group C gene polymorphisms and risk of cutaneous melanoma: a case-control study". Carcinogenesis 26 (6): 1085–90. PMID 15731165. doi:10.1093/carcin/bgi055.
- ↑ Shore RE, Zeleniuch-Jacquotte A, Currie D, Mohrenweiser H, Afanasyeva Y, Koenig KL, Arslan AA, Toniolo P, Wirgin I (May 2008). "Polymorphisms in XPC and ERCC2 genes, smoking and breast cancer risk". Int. J. Cancer 122 (9): 2101–5. PMID 18196582. doi:10.1002/ijc.23361.
- ↑ 15,0 15,1 Qiao Y, Spitz MR, Guo Z, Hadeyati M, Grossman L, Kraemer KH, Wei Q (November 2002). "Rapid assessment of repair of ultraviolet DNA damage with a modified host-cell reactivation assay using a luciferase reporter gene and correlation with polymorphisms of DNA repair genes in normal human lymphocytes". Mutat. Res. 509 (1–2): 165–74. PMID 12427537. doi:10.1016/S0027-5107(02)00219-1.
- ↑ 16,0 16,1 Huang MY, Fang WY, Lee SC, Cheng TL, Wang JY, Lin SR (2008). "ERCC2 2251A>C genetic polymorphism was highly correlated with early relapse in high-risk stage II and stage III colorectal cancer patients: a preliminary study". BMC Cancer 8: 50. PMC 2262891. PMID 18267032. doi:10.1186/1471-2407-8-50.
- ↑ Spitz MR, Wu X, Wang Y, Wang LE, Shete S, Amos CI, Guo Z, Lei L, Mohrenweiser H, Wei Q (February 2001). "Modulation of nucleotide excision repair capacity by XPD polymorphisms in lung cancer patients". Cancer Res. 61 (4): 1354–7. PMID 11245433.
Véxase tamén
editarOutros artigos
editarBibliografía
editar- Ellenberger T, Friedberg EC, Walker GS, Wolfram S, Wood RJ, Schultz R (2006). DNA repair and mutagenesis. Washington, D.C: ASM Press. ISBN 1-55581-319-4.
- Satoh MS, Hanawalt PC (September 1996). "TFIIH-mediated nucleotide excision repair and initiation of mRNA transcription in an optimized cell-free DNA repair and RNA transcription assay". Nucleic Acids Res. 24 (18): 3576–82. PMC 146147. PMID 8836185. doi:10.1093/nar/24.18.3576. Article on the relation between TFIIH and NER
- Frit P, Kwon K, Coin F, Auriol J, Dubaele S, Salles B, Egly JM (December 2002). "Transcriptional activators stimulate DNA repair". Mol. Cell 10 (6): 1391–401. PMID 12504014. doi:10.1016/S1097-2765(02)00732-3.
- Mellon I (September 2005). "Transcription-coupled repair: a complex affair". Mutat. Res. 577 (1–2): 155–61. PMID 15913669. doi:10.1016/j.mrfmmm.2005.03.016.