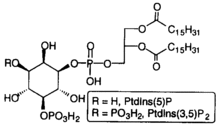
Fosfatidilinositol (3,5)-bisfosfato
O fosfatidilinositol 3,5-bisfosfato (PtdIns(3,5)P2 ou PI(3,5)P2) é un dos sete fosfoinosítidos que se encontran nas membranas das células eucarióticas. [3] En células quiescentes os niveis de PtdIns(3,5)P2, cuantifícanse normalmente por HPLC, e son os máis baixos de entre os fosfoinosítidos presentes constitutivamente na membrana. Son aproximadamente de 3 a 5 veces menores que os do PtdIns3P e o PtdIns5P, e máis de 100 veces menores que o dos abondosos PtdIns4P e PtdIns(4,5)P2. [4] O PtdIns(3,5)P2 descubriuse nos fibroblastos de rato e no lévedo Saccharomyces cerevisiae en 1997. [5] [6] En S. cerevisiae os niveis de PtdIns(3,5)P2 increméntanse drasticamente durante un shock hiperosmótico.[6] Esta resposta ás condicións hiperosmóticas non está conservada na maioría das células de mamíferos examinadas agás nos adipocitos 3T3L1 diferenciados. [6] [7]
Metabolismo
editarA única vía coñecida actualmente para a produción de PtdIns(3,5)P2 é a síntese catalizada pola fosfoinosítido quinase PIKfyve. Experimentos realizados en fibroblastos de ratos revelaron que o PtdIns(3,5)P2 é revertido a PtdIns3P pouco despois da súa síntese.[5] En células de mamíferos, os PtdIns(3,5)P2 son sintetizados a partir do PtdIns3P por un complexo proteico que contén dous encimas con actividades opostas: a fosfoinosítido quinase PIKfyve e a PtdIns(3,5)P2 5-fosfatase Sac3/Fig4, que contén o dominio Sac1. [8] Estes dous encimas non interaccionan directamente, senón que son reunidos por un regulador asociado de PIKfyve, chamado ArPIKfyve/VAC14, que crea o armazón dun complexo regulador ternario, chamado o complexo PAS (siglas das primeiras letras de PIKfyve/ArPIKfyve/Sac3). [9] A PIKfyve únese ao complexo PAS sobre microdominios endosómicos enriquecidos en Rab5GTP/PtdIns3P por medio do seu dominio dedo FYVE, que se une selectivamente a PtdIns3P. [10] [11] [12] O papel esencial do complexo PAS na síntese de PtdIns(3,5)P2 e o seu recambio está apoiado en datos de silenciamento de proteínas mediada por ARN interferente pequeno e a expresión heteróloga dos compoñentes do complexo PAS en varios tipos celulares, e en datos de knockout de xenes de proteínas da complexo PAS. [7] [8] [13] [14] [15] [16] [17]
Unha vía adicional para o recambio do PtdIns(3,5)P2 implica as fosfatases da familia da miotubularina. A miotubularina 1 e a MTMR2 desfosforilan a posición 3 do PtdIns(3,5)P2; por tanto, o produto desta hidrólise é o PtdIns5P, en vez do PtdIns3P. [18] As proteínas do complexo PAS están conservadas evolutivamente con ortólogos que se encontran en S. cerevisae (é dicir, as proteínas Fab1p, Vac14p, e Fig4p) e en todos os eucariotas aos que se lles secuenciou o xenoma. Por tanto, crese que o PtdIns(3,5)P2 está presente en todos os eucariotas, nos que regula funcións celulares similares. As proteínas de lévedo Fab1p, Vac14p, e Fig4p tamén forman un complexo, chamado complexo Fab1. [19] Porén, o complexo Fab1 contén a maiores outras proteínas adicionais, [20] que poderían engadir unha capa regulatoria adicional do PtdIns(3,5)Psub>2 en lévedos. A composición de complexos proteicos que regulan os niveis de PtdIns(3,5)P2 noutras especies aínda non está aclarado.
Funcións e regulación
editarO PtdIns(3,5)P2 regula operacións endosómicas (fisión e fusión) que manteñen a homeostase das endomembranas e o correcto funcionamento das vías de tráfico celular que proceden ou pasan polos endosomas. A diminución dos niveis de PtdIns(3,5)P2 por perturbacións da PIKfyve celular por expresión heteróloga de mutantes puntuais de PIKfyve encimaticamente inactiva, [21] silenciamento mediado por ARN interferente pequeno, [22] inhibición farmacolóxica [23] e knockout de PIKFYVE [15] causan a formación de moitos vacúolos citosólicos, que se fan meirandes co tempo. Un feito importante é que a vacuolización inducida pola disfunción de PIKfyve e a diminución do PtdIns(3,5)P2 é reversible e podería ser rescatada selectivamente por microinxección citosólica de PtdIns(3,5)P2, [24] sobreexpresión de PIKfyve [21] ou eliminación por lavado do inhibidor YM201636 de PIKfyve. [23] A actividade da fosfatase Sac3 no complexo PAS tamén xoga un importante papel na regulación dos niveis de PtdIns(3,5)P2 e no mantemento da homeostase das endomenbranas. Así, a vacuolización citoplasmática inducida polo mutante dominante negativo PIKfyveK1831E é suprimida pola coexpresión dun mutante puntual de fosfatase Sac3 inactiva xunto con ArPIKfyve. [14] Os ensaios de reconstitución in vitro da fusión de endosomas e a formación/separación (fisión) de corpos multivesiculares (MVB) suxire un papel positivo do PtdIns(3,5)P2 na fisión de corpos multivesiculares a partir de endosomas temperáns e un papel negativo na fusión de endosomas. [8] [10] O PtdIns(3,5)P2 está implicado no transporte retrógrado dependente de microtúbulos desde endosomas temperáns e tardíos á rede do trans Golgi. [22] [25]
Un tratamento agudo con insulina incrementa os niveis de PtdIns(3,5)P2 nos adipocitos 3T3L1, tanto en membranas illadas coma en células intactas para promover o efecto da insulina sobre a translocación na superficie celular do transportador GLUT4 e o transporte de glicosa. [13] [14] Estas células tamén presentan un incremento marcado de PtdIns(3,5)P2 durante os shocks hiperosmóticos. [7] Outros estímulos, entre eles os sinais mitoxénicos como a IL-2 e a luz ultravioleta en linfocitos, [26] a activación da proteína quinase C polo PMA en plaquetas [27] e a estimulación por factor de crecemento epidérmico (EGF) de células COS, [28] tamén incrementan os niveis de PtdIns(3,5)P<asub>2.
O PtdIns(3,5)P2 xoga un papel clave no crecemento e desenvolvemento como se evidencia na letalidade preimplantación en modelos de ratos knockout para PIKfyve. [15] O feito de que os ratos heterocigotos para PIKfyve son claramente normais e viven ata unha idade adulta avanzada con só ~60% dos niveis de PtdIns(3,5)P2 normais ou "salvaxes" suxire que o PtdIns(3,5)P2 podería estar normalmente en exceso. [15]
Nos ratos knockout para ArPIKfyve/Vac14 ou Sac3/Fig4 obsérvase un descenso do 30-50% nos niveis de PtdIns(3,5)P2 e prodúcese unha dexeneración central masiva e neuropatía periférica. [16] [17] Estes estudos indican que os niveis reducidos de PtdIns(3,5)P>sub>2, por un mecanismo aínda non identificado, median a morte neuronal. Ao contrario, o knockout para a fosfatase MTMR2, que tamén causa neuropatía periférica, está acompañada pola elevación dos niveis de PtdIns(3,5)P2. [29] Xa que logo, non está claro se os niveis anormais de PtdIns(3,5)P2 afectan selectivamente ás funcións das neuronas periféricas e como o fan.
Efectores
editarOs fosfoinosítidos considéranse xeralmente como sinais ancorados na membrana que recrutan proteínas efectoras citosólicas específicas. Ata agora, propuxéronse varias destas proteínas como posibles efectores do PtdIns(3,5)P2. Desafortunadamente, as expectativas que se tiñan de que eses efectores estarían evolutivamente conservados e que compartirían un moitvo común de unión ao PtdIns(3,5)P2 de alta afinidade non se cumpriron. Por exemplo, a deleción de Atg18p, unha proteína implicada tamén na autofaxia en S. cerevisae, causa o agrandamento dos vacúolos e unha multiplicación por 10 dos niveis de PtdIns(3,5)P2. A Atg18p únese ao PtdIns(3,5)P2 con alta afinidade e especificidade. [30] Porén, excepto para a autofaxia, os ortólogos de mamífero da Atg18p non comparten funcións similares. [31] Outras dúas proteínas dos lévedos (Ent3p e Ent5p) que se atoparon en estruturas endosómicas e prevacuolares son efectores potenciais do PtdIns(3,5)P2 na clasificación dos corpos multivesiculares. Conteñen un dominio ENTH para a unión de fosfoinosítidos e a súa deleción causa defectos na clasificación dos corpos multivesiculares que lembran aos observados na deleción de Fab1p. [32] Porén, nin Ent3p nin Ent5p teñen especificidade de unión de alta afinidade preferencial para o PtdIns(3,5)P2 in vitro. [33] O VPS24 de mamífero (un membro da familia de proteínas de corpos multivesiculares cargados ou CHMPs) é outro posible efector do PtdIns(3,5)P2. [34] As medidas de resonancia plasmónica de superficie non apoian que haxa un recoñecemento específico ou de alta afinidade do PtdIns(3,5)P2 para os VPS24 de mamíferos ou de lévedos. [33] O canal catiónico transmembrana humano TRPML1 (cuxa inactivación xenética causa unha enfermidade de almacenamento lisosómico) foi proposto recentemente como un posible efector do PtdIns(3,5)P2, baseándose nos ensaios de unión in vitro e a súa capacidade para rescatar o fenotipo de vacuolación en fibroblastos en ratos knockout ArPIKfyve/Vac14. [35] Pero a deleción da proteína ortóloga en lévedos non causa o agrandamento dos vacúolos, [36] o que pon en dúbida a conservación evolutiva deste mecanismo efector. Cómpren máis estudos para validar isto ou para descubrir efectores aínda non coñecidos do PtdIns(3,5)P2.
Notas
editar- ↑ PubChem - Phosphatidylinositol 3,5-diphosphate
- ↑ CHEBI - 1-phosphatidyl-1D-myo-inositol 3,5-bisphosphate(5−) (CHEBI:57923) [1]
- ↑ Di Paolo G, De Camilli P. Phosphoinositides in cell regulation and membrane dynamics. Nature. 2006 Oct 12;443(7112):651-7. PMID 17035995
- ↑ Shisheva A. Regulating Glut4 vesicle dynamics by phosphoinositide kinases and phosphoinositide phosphatases. Front Biosci. 2003 Sep 1;8:s945-56. Review. PMID 12957825
- ↑ 5,0 5,1 Whiteford CC, Brearley CA, Ulug ET. Phosphatidylinositol 3,5-bisphosphate defines a novel PI 3-kinase pathway in resting mouse fibroblasts. Biochem J. 1997 May 1;323 ( Pt 3):597-601. PMID 9169590
- ↑ 6,0 6,1 6,2 Dove SK, Cooke FT, Douglas MR, Sayers LG, Parker PJ, Michell RH. Osmotic stress activates phosphatidylinositol-3,5-bisphosphate synthesis. Nature. 1997 Nov 13;390(6656):187-92. PMID 9367158
- ↑ 7,0 7,1 7,2 Sbrissa D, Shisheva A. Acquisition of unprecedented phosphatidylinositol 3,5-bisphosphate rise in hyperosmotically stressed 3T3-L1 adipocytes, mediated by ArPIKfyve-PIKfyve pathway. J Biol Chem. 2005 Mar 4;280(9):7883-9. Epub 2004 Nov 16. PMID 15546865
- ↑ 8,0 8,1 8,2 Sbrissa D, Ikonomov OC, Fu Z, Ijuin T, Gruenberg J, Takenawa T, Shisheva A. Core protein machinery for mammalian phosphatidylinositol 3,5-bisphosphate synthesis and turnover that regulates the progression of endosomal transport. Novel Sac phosphatase joins the ArPIKfyve-PIKfyve complex. J Biol Chem. 2007 Aug 17;282(33):23878-91. Epub 2007 Jun 7. doi 10.1074/jbc.M611678200 PMID 17556371
- ↑ Sbrissa D, Ikonomov OC, Fenner H, Shisheva A. ArPIKfyve homomeric and heteromeric interactions scaffold PIKfyve and Sac3 in a complex to promote PIKfyve activity and functionality. J Mol Biol. 2008 Dec 26;384(4):766-79. Epub 2008 Oct 11. doi 10.1016/j.jmb.2008.10.009 PMID 18950639
- ↑ 10,0 10,1 Ikonomov OC, Sbrissa D, Shisheva A. Localized PtdIns 3,5-P2 synthesis to regulate early endosome dynamics and fusion. Am J Physiol Cell Physiol. 2006 Aug;291(2):C393-404. Epub 2006 Mar 1. doi 10.1152/ajpcell.00019.2006 PMID 16510848
- ↑ Shisheva A, Sbrissa D, Ikonomov O. Cloning, characterization, and expression of a novel Zn2+-binding FYVE finger-containing phosphoinositide kinase in insulin-sensitive cells. Mol Cell Biol. 1999 Jan; 19(1):623-34. PMID 9858586
- ↑ Sbrissa D, Ikonomov OC, Shisheva A. Phosphatidylinositol 3-phosphate-interacting domains in PIKfyve. Binding specificity and role in PIKfyve. Endomenbrane localization. J Biol Chem. 2002 Feb 22;277(8):6073-9. Epub 2001 Nov 12. doi 10.1074/jbc.M110194200 PMID 11706043
- ↑ 13,0 13,1 Ikonomov OC, Sbrissa D, Dondapati R, Shisheva A. ArPIKfyve-PIKfyve interaction and role in insulin-regulated GLUT4 translocation and glucose transport in 3T3-L1 adipocytes. Exp Cell Res. 2007 Jul 1;313(11):2404-16. Epub 2007 Mar 30. doi 10.1016/j.yexcr.2007.03.024 PMID 17475247
- ↑ 14,0 14,1 14,2 Ikonomov OC, Sbrissa D, Fenner H, Shisheva A. PIKfyve-ArPIKfyve-Sac3 core complex: contact sites and their consequence for Sac3 phosphatase activity and endocytic membrane homeostasis. J Biol Chem. 2009 Dec 18;284(51):35794-806. Epub . doi 10.1074/jbc.M109.037515 PMID 19840946
- ↑ 15,0 15,1 15,2 15,3 Ikonomov OC, Sbrissa D, Delvecchio K, Xie Y, Jin JP, Rappolee D, Shisheva A. The phosphoinositide kinase PIKfyve is vital in early embryonic development: preimplantation lethality of PIKfyve-/- embryos but normality of PIKfyve+/- mice. J Biol Chem. 2011 Apr 15;286(15):13404-13. Epub 2011 Feb 24. doi 10.1074/jbc.M111.222364 PMID 21349843
- ↑ 16,0 16,1 Zhang Y, Zolov SN, Chow CY, Slutsky SG, Richardson SC, Piper RC, Yang B, Nau JJ, Westrick RJ, Morrison SJ, Meisler MH, Weisman LS. Loss of Vac14, a regulator of the signaling lipid phosphatidylinositol 3,5-bisphosphate, results in neurodegeneration in mice. Proc Natl Acad Sci U S A. 2007 Oct 30;104(44):17518-23. Epub 2007 Oct 23.PMID 17956977
- ↑ 17,0 17,1 Chow CY, Zhang Y, Dowling JJ, Jin N, Adamska M, Shiga K, Szigeti K, Shy ME, Li J, Zhang X, Lupski JR, Weisman LS, Meisler MH. Mutation of FIG4 causes neurodegeneration in the pale tremor mouse and patients with CMT4J. Nature. 2007 Jul 5;448(7149):68-72. Epub 2007 Jun 17.PMID 17572665
- ↑ Shisheva A. PIKfyve: Partners, significance, debates and paradoxes. Cell Biol Int. 2008 Jun;32(6):591-604. Epub 2008 Jan 25. Review. doi 10.1016/j.cellbi.2008.01.006 PMID 18304842
- ↑ Botelho RJ, Efe JA, Teis D, Emr SD. Assembly of a Fab1 phosphoinositide kinase signaling complex requires the Fig4 phosphoinositide phosphatase. Mol Biol Cell. 2008 Oct;19(10):4273-86. Epub 2008 Jul 23. PMID 1865348
- ↑ Jin N, Chow CY, Liu L, Zolov SN, Bronson R, Davisson M, Petersen JL, Zhang Y, Park S, Duex JE, Goldowitz D, Meisler MH, Weisman LS. VAC14 nucleates a protein complex essential for the acute interconversion of PI3P and PI(3,5)P(2) in yeast and mouse. EMBO J. 2008 Dec 17;27(24):3221-34. Epub 2008 Nov 27. doi 10.1038/emboj.2008.248 PMID 19037259
- ↑ 21,0 21,1 Ikonomov OC, Sbrissa D, Shisheva A. Mammalian cell morphology and endocytic membrane homeostasis require enzymatically active phosphoinositide 5-kinase PIKfyve. J Biol Chem. 2001 Jul 13;276(28):26141-7. Epub 2001 Apr 2. doi 10.1074/jbc.M101722200 PMID 11285266
- ↑ 22,0 22,1 Rutherford AC, Traer C, Wassmer T, Pattni K, Bujny MV, Carlton JG, Stenmark H, Cullen PJ. The mammalian phosphatidylinositol 3-phosphate 5-kinase (PIKfyve) regulates endosome-to-TGN retrograde transport. J Cell Sci. 2006 Oct 1;119(19):3944-57. Epub 2006 Sep 5. doi 10.1242/jcs.03153 PMID 16954148
- ↑ 23,0 23,1 Jefferies HB, Cooke FT, Jat P, Boucheron C, Koizumi T, Hayakawa M, Kaizawa H, Ohishi T, Workman P, Waterfield MD, Parker PJ. A selective PIKfyve inhibitor blocks PtdIns(3,5)P(2) production and disrupts endomembrane transport and retroviral budding. EMBO Rep. 2008 Feb;9(2):164-70. Epub 2008 Jan 11. doi 10.1038/sj.embor.7401155 PMID 18188180
- ↑ Ikonomov OC, Sbrissa D, Mlak K, Kanzaki M, Pessin J, Shisheva A. Functional dissection of lipid and protein kinase signals of PIKfyve reveals the role of PtdIns 3,5-P2 production for endomembrane integrity. J Biol Chem. 2002 Mar 15;277(11):9206-11. Epub 2001 Nov 19. PMID 11714711
- ↑ Ikonomov OC, Fligger J, Sbrissa D, Dondapati R, Mlak K, Deeb R, Shisheva A. Kinesin adapter JLP links PIKfyve to microtubule-based endosome-to-trans-Golgi network traffic of furin. J Biol Chem. 2009 Feb 6;284(6):3750-61. Epub 2008 Dec 4. doi 10.1074/jbc.M806539200 PMID 19056739.
- ↑ Jones DR, González-García A, Díez E, Martinez-A C, Carrera AC, Meŕida I. The identification of phosphatidylinositol 3,5-bisphosphate in T-lymphocytes and its regulation by interleukin-2. J Biol Chem. 1999 Jun 25;274(26):18407-13. PMID 10373447
- ↑ Banfić H, Downes CP, Rittenhouse SE. Biphasic activation of PKBalpha/Akt in platelets. Evidence for stimulation both by phosphatidylinositol 3,4-bisphosphate, produced via a novel pathway, and by phosphatidylinositol 3,4,5-trisphosphate. J Biol Chem. 1998 May 8;273(19):11630-7. PMID 9565582
- ↑ Tsujita K, Itoh T, Ijuin T, Yamamoto A, Shisheva A, Laporte J, Takenawa T. Myotubularin regulates the function of the late endosome through the gram domain-phosphatidylinositol 3,5-bisphosphate interaction. J Biol Chem. 2004 Apr 2;279(14):13817-24. Epub 2004 Jan 12. PMID 14722070
- ↑ Vaccari I, Dina G, Tronchère H, Kaufman E, Chicanne G, Cerri F, Wrabetz L, Payrastre B, Quattrini A, Weisman LS, Meisler MH, Bolino A. Genetic interaction between MTMR2 and FIG4 phospholipid phosphatases involved in Charcot-Marie-Tooth neuropathies. PLoS Genet. 2011 Oct;7(10):e1002319. Epub 2011 Oct 20. PMID 22028665
- ↑ Dove SK, Piper RC, McEwen RK, Yu JW, King MC, Hughes DC, Thuring J, Holmes AB, Cooke FT, Michell RH, Parker PJ, Lemmon MA. Svp1p defines a family of phosphatidylinositol 3,5-bisphosphate effectors. EMBO J. 2004 May 5;23(9):1922-33. Epub 2004 Apr 22. PMID 15103325
- ↑ Dove SK, Dong K, Kobayashi T, Williams FK, Michell RH. Phosphatidylinositol 3,5-bisphosphate and Fab1p/PIKfyve underPPIn endo-lysosome function. Biochem J. 2009 Apr 1;419(1):1-13. Review.PMID 19272020
- ↑ Friant S, Pécheur EI, Eugster A, Michel F, Lefkir Y, Nourrisson D, Letourneur F. Ent3p Is a PtdIns(3,5)P2 effector required for protein sorting to the multivesicular body. Dev Cell. 2003 Sep;5(3):499-511. PMID 12967568
- ↑ 33,0 33,1 Michell RH, Heath VL, Lemmon MA, Dove SK. Phosphatidylinositol 3,5-bisphosphate: metabolism and cellular functions. Trends Biochem Sci. 2006 Jan;31(1):52-63. Epub 2005 Dec 20. Review. doi 10.1016/j.tibs.2005.11.013 PMID 16364647
- ↑ Whitley P, Reaves BJ, Hashimoto M, Riley AM, Potter BV, Holman GD. Identification of mammalian Vps24p as an effector of phosphatidylinositol 3,5-bisphosphate-dependent endosome compartmentalization. J Biol Chem. 2003 Oct 3;278(40):38786-95. Epub 2003 Jul 23. PMID 12878588
- ↑ Dong XP, Shen D, Wang X, Dawson T, Li X, Zhang Q, Cheng X, Zhang Y, Weisman LS, Delling M, Xu H. PI(3,5)P(2) controls membrane trafficking by direct activation of mucolipin Ca(2+) release channels in the endolysosome. Nat Commun. 2010 Jul 13;1:38. doi: 10.1038/ncomms1037. PMID 20802798
- ↑ Chang Y, Schlenstedt G, Flockerzi V, Beck A. Properties of the intracellular transient receptor potential (TRP) channel in yeast, Yvc1. FEBS Lett. 2010 May 17;584(10):2028-32. Epub 2009 Dec 24. PMID 20035756
Véxase tamén
editarOutros artigos
editar- Fosfoinosítidos
- Fosfatidilinositol bisfosfato
- Fosfatidilinositol (3,4)-bisfosfato
- Fosfatidilinositol (4,5)-bisfosfato
Ligazóns externas
editar- phosphatidylinositol 3,5-bisphosphate Medical Subject Headings (MeSH) na Biblioteca Nacional de Medicina dos EUA.