Virus Ebola
- Este artigo trata sobre o virus Ebola da especie Ebolavirus Zaire. Para o xénero ver Ebolavirus. Para a enfermidade ver febre hemorráxica do Ebola
| Species Ebolavirus Zaire | |||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|
| |||||||||||
| Clasificación científica | |||||||||||
| |||||||||||
| Virus membros (abreviación) | |||||||||||
|
Virus Ebola (EBOV) | |||||||||||
O virus Ebola (ás veces ortografado Ébola) (ver na táboa as denominacións do ICTV en inglés) abreviado como EBOV, é o único "virus" pertencente á "especie" Ebolavirus Zaire.[1][2] Antes este "virus" chamábase Ebolavirus Zaire, escrito igual que o nome da especie, pero sen letra cursiva, e despois propúxose o cambio do nome a virus Ebola, pero o da especie permaneceu igual.[3][4] O Ebolavirus Zaire é o máis perigoso das cinco especies de ebolavirus, todas as cales poden causar a enfermidade do virus Ebola, xa sexa en animais ou en humanos.[2] O virus causa unha febre hemorráxica extremadamente grave nos humanos e outros primates. O EBOV é un axente selecto encadrado como patóxeno do grupo de risco 4 da Organización Mundial da Saúde (que require un Nivel 4 de bioseguridade), e é un patóxeno prioritario da categoría A para os Institutos Nacionais da Saúde/Instituto Nacional da Alerxia e Enfermidades Infecciosas dos Estados Unidos, e un axente de bioterrorismo de categoría A para o CDC (Centros para o Control e Prevención de Enfermidades) dos Estados Unidos, e está considerado un axente biolóxico para o control da exportación polo Grupo Australia.

O seu nome procede do río Ebola (ás veces ortografado Ébola) en Zaire (hoxe chamado República Democrática do Congo), preto do cal se descubriu.[2]
O EBOV é un virus filamentoso ou cilíndrico cun xenoma de ARN monocatenario de aproximadamente 19 kb, que codifica sete proteínas estruturais, que son: nucleoproteína (NP), cofactor da polimerase (VP35), proteína da matriz (VP40), glicoproteína das espículas (GP), activador da transcrición (VP30), outra proteína da matriz (VP24), e unha ARN polimerase ARN dependente (L).[5]
| Especie | (en cursiva) | Virus | (non en | cursiva) |
|---|---|---|---|---|
| Versión en galego | Nome do ICTV en inglés | Versión en galego | Nome en inglés | Abreviatura |
| Ebolavirus Zaire | Zaire ebolavirus | HOXE: Virus Ebola ANTES: Ebolavirus Zaire |
HOXE: Ebola virus ANTES: Zaire ebolavirus |
HOXE: EBOV ANTES: ZEBOV |
Estrutura editar
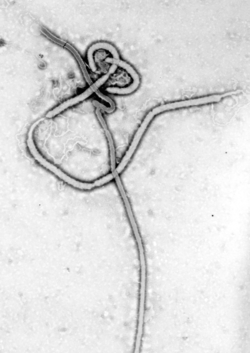
As micrografías electrónicas do EBOV mostran que ten a estrutura filamentosa característica dos filovirus (Filoviridae). O virión é cilíndrico/tubular cunha envoltura viral, unha matriz e unha nucleocápside e leva un xenoma de ARN de sentido negativo (ver imaxe). Os cilindros completos teñen aproximadamente 80 nm de diámetro, e presentan unha glicoproteína viral codificada (GP) que se proxecta formando espículas de 7-10 nm de longo desde a superficie da bicapa lipídica.[6] Os cilindros son de lonxitude variable, xeralmente de 800 nm, pero ás veces chegan a 1000 nm. A envoltura viral externa do virión deriva de evaxinacións da membrana plasmática da célula hóspede nas zonas onde se insiren as espículas de GP durante a súa biosíntese. Cada molécula de GP está separada da seguinte uns 10 nm. As proteínas virais VP40 e VP24 están localizadas entre a envoltura e a nucleocápside, no espazo da matriz.[7] No centro da estrutura do virión está a nucleocápside, que está composta por unha serie de proteínas virais unidas ao ARN de sentido negativo liñal de 18–19 kb sen poliadenilación 3' nin carapucha 5'; o ARN está enrolado helicoidalmente e en complexo coas proteínas NP, VP35, VP30, e L.[8] Esta hélice ten un diámetro de 80 nm e contén unha canle central de 20–30 nm de diámetro.
Xenoma editar
O virión contén unha molécula de ARN monocatenario liñal de sentido negativo de 18.959 a 18.961 nucleótidos. O extremo 3' non está poliadenilado e o 5' non ten carapucha. Codifica sete proteínas estruturais e unha proteína non estrutural. A orde dos xenes é 3′ – líder – NP – VP35 – VP40 – GP/sGP – VP30 – VP24 – L – tráiler – 5′; o líder e o tráiler son rexións non tanscritas, que levan importantes sinais para o control da transcrición, replicación, e empaquetado dos xenomas virais en novos virións. Identificáronse seccións dos xenes da NP e de L de filovirus en forma de xenes endóxenos en xenomas de moitos grupos de pequenos mamíferos.[9] [10][11]
Os 472 nucleótidos do extremo 3' e os 731 nucleótidos do extremo 5' son suficientes para a replicación do "minixenoma" viral, aínda que non son suficientes para a infección.[12] O material xenéntico do minixenoma non é infeccioso de seu, porque as proteínas virais, como a ARN polimerase ARN dependente, son necesarias para transcribir o xenoma viral orixinando ARNms dado que é un virus de sentido negativo, e tamén para a replicación do xenoma viral.
Entrada do virus editar
Unha proteína codificada polo hóspede, a Niemann–Pick C1 (NPC1), parece ser esencial para a infección do Ebola. Dous estudos independentes informaron que para a entrada na célula do virus Ebola e a súa replicación é necesaria esta proteína transportadora de colesterol NPC1.[13][14] Cando as células de pacientes da enfermidade de Niemann-Pick, tipo C se expoñen ao virus Ebola no laboratorio, as células sobreviven e parecen inmunes ao virus, o que indica que o virus depende da NPC1 para entrar na célula. Os mesmos estudos describiron resultados similares co filovirus emparentado co do Ebola, o virus Marburg, que mostraron que tamén necesita da NPC1 para a entrada na célula.[13][14] Ademais, a NPC1 media na infección ao unirse directamente á glicoproteína da envoltura nuclear.[14] Un estudo posterior confirmou o resultado de que a NPC1 é un receptor fundamental para que os filovirus poidan infectar ao unirse á glicoproteína viral, e viuse que o segundo dominio lisosómico da NPC1 é o que intervén nesta unión.[15]
Nun dos estudos orixinais, unha pequena molécula (a bencilpiperacina adamantano diamida) inhibía a infección polo virus Ebola impedindo que a proteína do virus se una á NPC1.[14][16] No outro estudo, os ratos que eran heterocigotos para a NPC1 estaban protexidos do virus Ebola adaptado para ratos.[13] Todo isto indica que a NPC1 pode ser unha diana terapéutica potencial para os fármacos antivirais contra o Ebola.
Replicación editar
Os virus utilizan a maquinaria e metabolismo da célula hóspede para producir moitas copias de si mesmos e ensamblarse dentro das células.[8] O virus Ebola seguiría estes pasos:[17][18][19]
- O virus únese a receptores da célula hóspede polo peplómero glicoproteína (GP) da súa superficie, e é seguidamente endocitado dentro dun macropinosoma (vesícula) na célula hóspede.[20][21]
- A membrana viral fusiónase coa membrana da vesícula, e libérase a nucleocápside no citoplasma.
- O ARN monocatenario de sentido negativo encapsulado utilízase como molde para a síntese (3'–5') de ARNms monocistrónicos poliadenilados.
- Ten lugar a tradución do ARNm viral a proteínas utilizando a maquinaria da célula hóspede.
- Procésanse as proteínas virais, clívase o precursor da glicoproteína (GP0) para dar a GP1 e a GP2, as cales están moi glicosiladas. Estas dúas moléculas ensámblanse, primeiro formando heterodímeros, e despois trímeros que constitúen os peplómeros de superficie. O precursor da glicoproteína segregada (sGP) clívase formando a sGP e o delta-péptido, e ambos son liberados da célula.
- A medida que os niveis de proteínas virais se elevan, ocorre un cambio desde as actividades de tradución ás de replicación. Sintetízase agora un ARN monocatenario de sentido positivo complementario utilizando como molde o ARN xenómico de sentido negativo; despois o ARN de sentido positivo é utilizado como molde para a síntese de novo ARN xenómico de sentido negativo, que é rapidamente encapsidado.
- As nucleocápsides formadas e as proteínas de envoltura asócianse na membrana plasmática da célula hóspede. Finalmente, ten lugar a saída por evaxinación do virus, que destrúe a célula.
Historia editar
Segundo as normas de nomeamento de taxons establecidas polo Comité Internacional de Taxonomía de Virus (ICTV), o nome da especie Ebolavirus Zaire vai escrito con maiúscula e en cursiva, e precedido da palabra "species". Os nomes dos distintos ebolavirus non se escriben en cursiva, polo que se escribirá virus Ebola. As "especies" víricas poden ter varios "virus", pero neste caso esta especie ten un só virus.[2][22]
O virus Ebola (abreviado EBOV)[23][24][25] recibe o seu nome da rexión do río africano Ebola en Zaire (hoxe República Democrática do Congo), onde foi descuberto.[2] O nome do virus cambiou varias veces.[26][27][28][29]
O virus Ebola foi inicialmente presentado como unha posible "cepa" nova do virus Marburg en 1977 por dous equipos diferentes de investigadores.[23][24] Ao mesmo tempo, un terceiro equipo introduciu tamén o nome virus Ebola.[25] En 2000, o nome do virus cambiou a virus Ebola Zaire (Zaire Ebola virus),[30] e en 2002 a Ebolavirus Zaire.[31] Porén, a maioría dos artigos científicos seguiron referíndose ao virus Ebola ou utilizaban os termos virus Ebola e Ebolavirus Zaire en paralelo. En consecuencia, en 2010, un grupo de investigadores recomendou que o nome "Ebola virus" (virus Ebola) fose adoptado como unha subclasificación dentro da especie Zaire ebolavirus, ábreviado como EBOV.[2] Outras abreviaturas previas eran EBOV-Z (para Ebola virus Zaire) e ZEBOV (para Zaire Ebola virus ou Zaire ebolavirus). En 2011, o ICTV rexeitou explicitamente unha proposta (2010.010bV) para recoñecer este nome, xa que o ICTV non designa nomes oficiais para os subtipos, variantes, cepas ou outros agrupamentos no nivel de subespecies.[32] Actualmente, o ICTV non recoñece oficialmente "Ebola virus" (virus Ebola) como rango taxonómico, e segue usando e recomendando só a designación da especie Zaire ebolavirus (Ebolavirus Zaire).[33]
Criterios de inclusión na especie e no virus editar
Un virus do xénero Ebolavirus pertence á especie Ebolavirus Zaire se cumpre os seguintes criterios:[2]
- encóntrase na República Democrática do Congo, Gabón, ou a República do Congo;
- ten un xenoma con dous ou tres solapamentos de xenes (VP35/VP40, GP/VP30, VP24/L);
- ten unha secuencia xenómica que difire do virus tipo en menos do 30%.
Ademais, un Ebolavirus Zaire é un virus Ebola se o seu xenoma non diverxe do da variante Mayinga (EBOV/May) en máis do 10% a nivel de nucleótidos.[2]
Notas editar
- ↑ WHO. "Ebola virus disease".
- ↑ 2,0 2,1 2,2 2,3 2,4 2,5 2,6 2,7 Kuhn, Jens H.; Becker, Stephan; Ebihara, Hideki; Geisbert, Thomas W.; Johnson, Karl M.; Kawaoka, Yoshihiro; Lipkin, W. Ian; Negredo, Ana I; et al. (2010). "Proposal for a revised taxonomy of the family Filoviridae: Classification, names of taxa and viruses, and virus abbreviations". Archives of Virology 155 (12): 2083–103. PMC 3074192. PMID 21046175. doi:10.1007/s00705-010-0814-x.
- ↑ En realidade o ICTV non oficializa nomes por debaixo do nivel de especie, e só son oficiais os nomes das especies, a pesar do cal o nome "virus Ebola" é amplamente usado. A proposta de oficialización presentada en 2010 por Kuhn et al. foi rexeitada en 2011.
- ↑ International Committee on Taxonomy of Viruses. "Virus Taxonomy: 2013 Release".
- ↑ Nanbo, Asuka; Watanabe, Shinji; Halfmann, Peter; Kawaoka, Yoshihiro (4 Feb 2013). "The spatio-temporal distribution dynamics of Ebola virus proteins and RNA in infected cells". Nature 3: 1206. Bibcode:2013NatSR...3E1206N. doi:10.1038/srep01206.
- ↑ Klenk & Feldmann 2004, p. 28
- ↑ Feldmann, H. K. (1993). "Molecular biology and evolution of filoviruses". Archives of virology. Supplementum 7: 81–100. ISSN 0939-1983. PMID 8219816.
- ↑ 8,0 8,1 Biomarker Database. Ebola virus. Korea National Institute of Health. Arquivado dende o orixinal o 22 de abril de 2008. Consultado o 2009-05-31.
- ↑ Taylor, D.; Leach, R.; Bruenn, J. (2010). "Filoviruses are ancient and integrated into mammalian genomes". BMC Evolutionary Biology 10: 193. doi:10.1186/1471-2148-10-193. PMC 2906475. PMID 20569424.
- ↑ Belyi, V. A.; Levine, A. J.; Skalka, A. M. (2010). "Unexpected Inheritance: Multiple Integrations of Ancient Bornavirus and Ebolavirus/Marburgvirus Sequences in Vertebrate Genomes". In Buchmeier, Michael J. PLoS Pathogens 6 (7): e1001030. doi:10.1371/journal.ppat.1001030. PMC 2912400. PMID 20686665.
- ↑ Taylor, D. J.; Ballinger, M. J.; Zhan, J. J.; Hanzly, L. E.; Bruenn, J. A. (2014). "Evidence that ebolaviruses and cuevaviruses have been diverging from marburgviruses since the Miocene". PeerJ 2: e556. doi:10.7717/peerj.556. [1]
- ↑ Klenk, H-D; Feldmann, H (editor) (2004). Ebola and Marburg Viruses: Molecular and Cellular Biology. Horizon Bioscience. ISBN 978-1-904933-49-6.
- ↑ 13,0 13,1 13,2 Carette JE, Raaben M, Wong AC, Herbert AS, Obernosterer G, Mulherkar N, Kuehne AI, Kranzusch PJ, Griffin AM, Ruthel G, Dal Cin P, Dye JM, Whelan SP, Chandran K, Brummelkamp TR (September 2011). "Ebola virus entry requires the cholesterol transporter Niemann-Pick C1". Nature 477 (7364): 340–3. PMC 3175325. PMID 21866103. doi:10.1038/nature10348. Resumo divulgativo – New York Times.
- ↑ 14,0 14,1 14,2 14,3 Côté M, Misasi J, Ren T, Bruchez A, Lee K, Filone CM, Hensley L, Li Q, Ory D, Chandran K, Cunningham J (September 2011). "Small molecule inhibitors reveal Niemann-Pick C1 is essential for Ebola virus infection". Nature 477 (7364): 344–8. PMC 3230319. PMID 21866101. doi:10.1038/nature10380. Resumo divulgativo – New York Times.
- ↑ Miller EH, Obernosterer G, Raaben M, Herbert AS, Deffieu MS, Krishnan A, Ndungo E, Sandesara RG, Carette JE, Kuehne AI, Ruthel G, Pfeffer SR, Dye JM, Whelan SP, Brummelkamp TR, Chandran K (March 2012). "Ebola virus entry requires the host-programmed recognition of an intracellular receptor". EMBO Journal 31 (8): 1947–60. PMC 3343336. PMID 22395071. doi:10.1038/emboj.2012.53.
- ↑ Flemming A (October 2011). "Achilles heel of Ebola viral entry". Nat Rev Drug Discov 10 (10): 731. PMID 21959282. doi:10.1038/nrd3568.
- ↑ Mühlberger, Elke (4 December 2016). "Filovirus replication and transcription". Future Virology 2 (2): 205–215. ISSN 1746-0794. PMC 3787895. PMID 24093048. doi:10.2217/17460794.2.2.205.
- ↑ Feldmann, Heinz; Klenk, Hans-Dieter (1 January 1996). "Filoviruses". Medical Microbiology. Consultado o 4 December 2016.
- ↑ Lai, Kang Yiu; Ng, Wing Yiu George; Cheng, Fan Fanny (28 November 2014). "Human Ebola virus infection in West Africa: a review of available therapeutic agents that target different steps of the life cycle of Ebola virus". Infectious Diseases of Poverty 3: 43. ISSN 2049-9957. PMC 4334593. PMID 25699183. doi:10.1186/2049-9957-3-43.
- ↑ Saeed, M. F.; Kolokoltsov, A. A.; Albrecht, T.; Davey, R. A. (2010). Basler, Christopher F., ed. "Cellular Entry of Ebola Virus Involves Uptake by a Macropinocytosis-Like Mechanism and Subsequent Trafficking through Early and Late Endosomes". PLoS Pathogens 6 (9): e1001110. PMC 2940741. PMID 20862315. doi:10.1371/journal.ppat.1001110.
- ↑ Saeed, M. F.; Kolokoltsov, A. A.; Albrecht, T.; Davey, R. A. (2010). "Cellular Entry of Ebola Virus Involves Uptake by a Macropinocytosis-Like Mechanism and Subsequent Trafficking through Early and Late Endosomes". In Basler, Christopher F. PLoS Pathogens 6 (9): e1001110. doi:10.1371/journal.ppat.1001110. PMC 2940741. PMID 20862315.
- ↑ Brown, Rob (18 July 2014) The virus detective who discovered Ebola in 1976 BBC News Magazine, Retrieved 18 July 2014
- ↑ 23,0 23,1 Pattyn, S.; Jacob, W.; van der Groen, G.; Piot, P.; Courteille, G. (1977). "Isolation of Marburg-like virus from a case of haemorrhagic fever in Zaire". Lancet 309 (8011): 573–4. PMID 65663. doi:10.1016/s0140-6736(77)92002-5.
- ↑ 24,0 24,1 Bowen, E. T. W.; Lloyd, G.; Harris, W. J.; Platt, G. S.; Baskerville, A.; Vella, E. E. (1977). "Viral haemorrhagic fever in southern Sudan and northern Zaire. Preliminary studies on the aetiological agent". Lancet 309 (8011): 571–3. PMID 65662. doi:10.1016/s0140-6736(77)92001-3.
- ↑ 25,0 25,1 Johnson, K. M.; Webb, P. A.; Lange, J. V.; Murphy, F. A. (1977). "Isolation and partial characterisation of a new virus causing haemorrhagic fever in Zambia". Lancet 309 (8011): 569–71. PMID 65661. doi:10.1016/s0140-6736(77)92000-1.
- ↑ Netesov, S. V.; Feldmann, H.; Jahrling, P. B.; Klenk, H. D.; Sanchez, A. (2000). "Family Filoviridae". En van Regenmortel, M. H. V.; Fauquet, C. M.; Bishop, D. H. L.; Carstens, E. B.; Estes, M. K.; Lemon, S. M.; Maniloff, J.; Mayo, M. A.; McGeoch, D. J.; Wickner, R. B. Virus Taxonomy—Seventh Report of the International Committee on Taxonomy of Viruses. San Diego, USA: Academic Press. pp. 539–48. ISBN 0-12-370200-3.
- ↑ Pringle, C. R. (1998). "Virus taxonomy-San Diego 1998". Archives of Virology 143 (7): 1449–59. PMID 9742051. doi:10.1007/s007050050389.
- ↑ Feldmann, H.; Geisbert, T. W.; Jahrling, P. B.; Klenk, H.-D.; Netesov, S. V.; Peters, C. J.; Sanchez, A.; Swanepoel, R.; et al. (2005). "Family Filoviridae". En Fauquet, C. M.; Mayo, M. A.; Maniloff, J.; Desselberger, U.; Ball, L. A. Virus Taxonomy—Eighth Report of the International Committee on Taxonomy of Viruses. San Diego, USA: Elsevier/Academic Press. pp. 645–653. ISBN 0122499514.
- ↑ Mayo, M. A. (2002). "ICTV at the Paris ICV: results of the plenary session and the binomial ballot". Archives of Virology 147 (11): 2254–60. doi:10.1007/s007050200052.
- ↑ Pringle, C. R. (1998). "Virus taxonomy-San Diego 1998". Archives of Virology 143 (7): 1449–59. PMID 9742051. doi:10.1007/s007050050389.
- ↑ Mayo, M. A. (2002). "ICTV at the Paris ICV: results of the plenary session and the binomial ballot". Archives of Virology 147 (11): 2254–60. doi:10.1007/s007050200052.
- ↑ "Replace the species name Lake Victoria marburgvirus with Marburg marburgvirus in the genus Marburgvirus". Arquivado dende o orixinal o 05 de marzo de 2016. Consultado o 09 de xaneiro de 2015.
- ↑ International Committee on Taxonomy of Viruses. "Virus Taxonomy: 2013 Release".
Véxase tamén editar
Outros artigos editar
Ligazóns externas editar
- ICTV Files and Discussions - Discussion forum and file distribution for the International Committee on Taxonomy of VirusesArquivado 07 de outubro de 2011 en Wayback Machine.
- Ebola: Africa’s Bloody Disease
- Ebola Hemorrhagic Fever - CDC.gov
- The Ebola Virus modelos 3D do virus Ebola, preparados por Visual Science, Moscova.
- ICTV Files and Discussions - Discussion forum and file distribution for the International Committee on Taxonomy of VirusesArquivado 07 de outubro de 2011 en Wayback Machine.