Histona
As histonas son proteínas con moitos residuos alcalinos ou básicos presentes no núcleo celular dos eucariotas que empaquetan e ordenan o ADN en unidades estruturais chamadas nucleosomas.[1][2] Son os principais compoñentes proteicos da cromatina, actúan como unha especie de carretes aredor dos cales se enrola o ADN, e xogan un papel importante na regulación xénica. Sen histonas o ADN estaría desenrolado nos cromosomas e sería moi longo (a razón lonxitude/largura sería de máis de 10 millóns a 1 no ADN humano). Por exemplo, cada célula humana ten uns 1,8 metros de ADN, pero se están enrolados sobre as histonas formando cromatina ten só uns 90 milímetros (0,09 m), e, máis tarde, cando se duplica e condensa a cromosomas durante a mitose, ocupa unha lonxitude de só 120 micrómetros.[3]

| Histona | |||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|
 | |||||||||||
| Identificadores | |||||||||||
| Símbolo | Core histone | ||||||||||
| Pfam | PF00125 | ||||||||||
| Pfam clan | CL0012 | ||||||||||
| InterPro | IPR007125 | ||||||||||
| SCOPe | 1hio / SUPFAM | ||||||||||
| |||||||||||
| Histona | |||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|
 | |||||||||||
| Identificadores | |||||||||||
| Símbolo | Linker histone | ||||||||||
| Pfam | PF00538 | ||||||||||
| InterPro | IPR005818 | ||||||||||
| SMART | SM00526 | ||||||||||
| SCOPe | 1hst / SUPFAM | ||||||||||
| |||||||||||
Clases de histonas editar
A secuencia de aminoácidos das histonas está moi conservada evolutivamente e poden ser agrupadas en cinco clases principais: H1/H5, H2A, H2B, H3, e H4.[2][4][5] Estes 5 tipos están agrupados nas dúas superclases seguintes:
- Histonas do octámero ou histonas core – H2A, H2B, H3 e H4.
- Histonas de unión ou enlace ou histonas linker – H1 e H5.
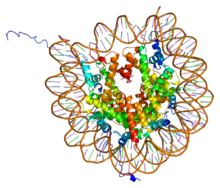
Cada unha das histonas core ensámblase con outra igual, e despois ensámblanse todas xuntas para formar os octámeros dos nucleosomas, orixinando unha especie de carrete no que se enrolan 147 pares de bases de ADN formando unha superhélice que se enrola á esquerda e dá 1,65 voltas ao carrete.[6] As histonas de unión H1 únense ao nucleosoma e aos puntos de entrada e saída do ADN do octámero, de maneira que fixan o ADN no seu lugar[7] o que permite a formación dunha estrutura de orde superior. A máis básica de tales estruturas é a fibra de 10 nm da cromatina ou conformación en colar de doas. Isto implica o enrolamento do ADN arredor dos nucleosomas deixando un tramo de ADN de aproximadamente 50 pares de bases que separa cada dous nucleosomas, chamado ADN espazador. O conxunto das histonas ensambladas e o ADN chámase cromatina. As estruturas de orde superior que pode formar a cromatina son a fibra cromatínica de 30 nm (que forma un zigzag irregular) e a fibra de 100 nm, que se poden atopar en células interfásicas. Durante a mitose e a meiose, unha maior condensación da cromatina orixina os cromosomas, formados polas interaccións entre os nucleosomas e outras proteínas reguladoras.
A seguinte é unha lista das proteínas histonas humanas:
| Superfamilia | Familia | Subfamilia | Membros |
|---|---|---|---|
| Histonas de unión ou enlace (linker) | |||
| H1 | |||
| H1F | H1F0, H1FNT, H1FOO, H1FX | ||
| H1H1 | HIST1H1A, HIST1H1B, HIST1H1C, HIST1H1D, HIST1H1E, HIST1H1T | ||
| Histonas do octámero (core) | |||
| H2A | |||
| H2AF | H2AFB1, H2AFB2, H2AFB3, H2AFJ, H2AFV, H2AFX, H2AFY, H2AFY2, H2AFZ | ||
| H2A1 | HIST1H2AA, HIST1H2AB, HIST1H2AC, HIST1H2AD, HIST1H2AE, HIST1H2AG, HIST1H2AI, HIST1H2AJ, HIST1H2AK, HIST1H2AL, HIST1H2AM | ||
| H2A2 | HIST2H2AA3, HIST2H2AC | ||
| H2B | |||
| H2BF | H2BFM, H2BFO, H2BFS, H2BFWT | ||
| H2B1 | HIST1H2BA, HIST1H2BB, HIST1H2BC, HIST1H2BD, HIST1H2BE, HIST1H2BF, HIST1H2BG, HIST1H2BH, HIST1H2BI, HIST1H2BJ, HIST1H2BK, HIST1H2BL, HIST1H2BM, HIST1H2BN, HIST1H2BO | ||
| H2B2 | HIST2H2BE | ||
| H3 | |||
| H3A1 | HIST1H3A, HIST1H3B, HIST1H3C, HIST1H3D, HIST1H3E, HIST1H3F, HIST1H3G, HIST1H3H, HIST1H3I, HIST1H3J | ||
| H3A2 | HIST2H3C | ||
| H3A3 | HIST3H3 | ||
| H4 | |||
| H41 | HIST1H4A, HIST1H4B, HIST1H4C, HIST1H4D, HIST1H4E, HIST1H4F, HIST1H4G, HIST1H4H, HIST1H4I, HIST1H4J, HIST1H4K, HIST1H4L | ||
| H44 | HIST4H4 |
Estrutura editar
O octámero ou core do nucleosoma está formado por dous dímeros das proteínas H2A-H2B e un tetrámero H3-H4, que forman dúas metades case simétricas na estrutura terciaria (simetría C2; unha macromolécula é a imaxe especular da outra).[6] Os dímeros H2A-H2B e o tetrámero H3-H4 tamén mostran unha simetría pseudodíada. As 4 histonas core (H2A, H2B, H3 e H4) son relativamente semellantes en estrutura e a súa secuencia está moi conservada na evolución, e todas presentan un motivo estrutural "hélice xiro hélice xiro hélice" (que permite unha fácil formación do dímero). Tamén comparten a característica de teren longas "colas" nun extremo da súa cadea de aminoácidos, que é a parte na que se producen as modificacións postraducionais (véxase máis adiante).
Tense proposto que as proteínas histonas están relacionadas evolutivamente coa parte helicoidal do dominio AAA+ ATPase estendido, co dominio C, e co dominio de recoñecemento de substrato N-terminal das proteínas Clp/Hsp100. Malia as diferenzas na súa topoloxía, estes tres pregamentos comparten un motivo homólogo hélice-cadea-hélice.[8]
A distancia entre nucleosomas foi medida por investigadores británicos usando a técnica da resonancia magnética de electróns, e variaba de 59 a 70 Å.[9]
As histonas poden establecer cinco tipos de interaccións co ADN:
- Dipolos de hélice formados nas hélices α nas histonas H2B, H3, e H4 orixinan que se acumule unha carga neta positiva no punto de interacción cos grupos fosfato cargados negativamente do ADN.
- Enlaces de hidróxeno entre o ADN e o grupo amida da cadea principal das proteínas histonas.
- Interaccións non polares entre a histona e os azucres desoxirribosa do ADN.
- Pontes salinas (atraccións electrostáticas) e enlaces de hidróxeno entre as cadeas laterais dos aminoácidos básicos (especialmente lisina e arxinina) e os osíxenos do fosfato do ADN.
- Insercións non específicas do suco menor das colas N-terminais de H3 e H2B en dous sucos menores da molécula de ADN.
A natureza moi básica das histonas, ademais de facilitar as interaccións ADN-histonas, contribúe á súa solubilidade en auga.
As histonas están suxeitas a modificacións postraducionais realizadas por encimas, fundamentalmente nas colas N-terminais, mais tamén nos dominios globulares .[10] Estas modificacións afectan á súa función de regulación xénica (ver funcións). Tales modificacións inclúen:
- metilación, unión encimática de grupos metilo (-CH3).
- citrulinación, conversión de arxinina en citrulina.
- acetilación, unión dun grupo acetilo (-CH3-CO).
- fosforilación, unión dun fosfato.
- SUMOilación, unión das pequenas proteínas reguladoras SUMO (parecidas á ubiquitina, pero con outra función).
- ubiquitinación, unión da pequena proteína reguladora ubiquitina, que marca a proteína para a degradación.
- ADP-ribosilación, adición de restos de ADP-ribosa.
Función editar
Compactación das cadeas do ADN editar
As histonas actúan como carretes arredor dos cales se enrola o ADN. Isto permite a compactación necesaria para acomodar os grandes xenomas eucarióticos dentro do núcleo celular. A molécula compactada é 40.000 veces máis curta ca a molécula sen compactar.
Regulación da cromatina editar
As histonas sofren modificacións postraducionais que alteran a súa interacción co ADN e as proteínas nucleares. As histonas H3 e H4 teñen longas colas que saen do nucleosoma, que poden ser modificadas covalentemente en varios puntos. Estas modificacións inclúen metilación, acetilación, fosforilación, ubiquitinación, SUMOilación, citrulinación, e ADP-ribosilación. Ademais das colas, a parte central das the histonas H2A, H2B, e H3 pode tamén modificarse. Pénsase que certas combinacións de modificacións constitúen un código, o chamado "código de histonas".[11][12] A modificación de histonas intervén en diversos procesos bioloxicos como a regulación xénica, a reparación do ADN e a condensación cromosómica (mitose) e espermatoxénese (meiose).[13]
A nomenclatura normal para indicar as modificacións nas histonas consta de varias parte, e é:
- Ponse primeiro o nome da histona (por exemplo, H3)
- Despois a abreviación dunha letra do nome do aminoácido afectado (por exemplo, K para lisina) e a posición do aminoácido na proteína.
- Finalmente o tipo de modificación (Me: metil, P: fosfato, Ac: acetil, Ub: ubiquitín)
Así H3K4me1 significa monometilación do 4º residuo (unha lisina) empezando polo principio (extremo N-terminal) da proteína H3.
Exemplos de modificacións de histonas na regulación da transcrición son:
| Tipo de modificación |
Histona | ||||||
|---|---|---|---|---|---|---|---|
| H3K4 | H3K9 | H3K14 | H3K27 | H3K79 | H4K20 | H2BK5 | |
| mono-metilación | activación[14] | activación[15] | activación[15] | activación[15][16] | activación[15] | activación[15] | |
| di-metilación | represión[17] | represión[17] | activación[16] | ||||
| tri-metilación | activación[18] | represión[15] | represión[15] | activación,[16] represión[15] |
represión[17] | ||
| acetilación | activación[18] | activación[18] | |||||
Historia editar
As histonas foron descubertas en 1884 por Albrecht Kossel. O nome histona data de finais do século XIX e procede do alemán Histon, de orixe incerta: quizais vén do grego histanai ou de histos. Ata principios da década de 1990, as histonas eran consideradas por moitos investigadores como un material de recheo para o ADN nuclear eucariótico, baseándose en parte nos modelos da "bóla e pau" de Mark Ptashne e outros que crían que a transcrición estaba activada por interaccións proteína-ADN e proteína-proteína sobre moldes de ADN basicamente espidos, como é o caso das bacterias. Durante a década de 1980s, os traballos de Michael Grunstein[19] demostraron que as histonas eucarióticas reprimían a transcrición xénica, e que a función dos activadores transcricionais é superar esta represión. Sabemos agora que as histonas xogan un papel tanto positivo coma negativo na regulación da expresión dos xenes, o que constitúe as bases do código de histonas.
O descubrimento da histona H5 parece datar da década de 1970,[20][21] e na clasificación foi agrupada coa histona H1.[2][4][5]
Conservación entre especies editar
As histonas atopáronse no núcleo das células eucariotas e en certas Archaea, concretamente as euriarqueas, pero non en bacterias. As histonas das arqueas poden ser parecidas aos precursores das histonas eucarióticas. As histonas están entre as proteínas coa secuencia máis conservada entre os eucariotas, o que indica o seu importante papel na bioloxía do núcleo.[2] Polo contrario, as células espermáticas maduras usan en gran medida protaminas para empaquetar o seu ADN xenómico (en vez de histonas), moi probablemente porque isto lles permite acadar un grao de empaquetamento aínda maior.[22]
As histonas do octámero (core) están tan conservadas, que presentan moi poucas diferenzas entre as súas secuencias de aminoácidos en diferentes especies. As histonas de unión (linker) xeralmente presentan máis dunha forma dentro dunha especie e están menos conservadas ca as outras.[23]
Hai algunhas formas variantes nalgunhas das principais clases. Estas variante comparten homoloxía na secuencia de aminoácidos e semellanza estrutural na parte central con algunha clase específica das histonas principais, pero tamén teñen as súas propias características que as distinguen das histonas principais. Estas histonas secundarias normalmente levan a cabo funcións específicas no metabolismo da cromatina. Por exemplo, a histona CenpA (similar á H3) é unha histona que está asociada exclusivamente coa rexión do centrómero do cromosoma. A variante H2A.Z da histona H2 está asociada cos promotores dos xenes que están en activa transcrición e tamén está implicada na prevención da propagación da heterocromatina silenciosa. Outra variante da H2, a H2A.X únese ao ADN que ten roturas na dobre cadea e marca a zona do ADN que está sometida a reparación. A histona H3.3 está asociada con xenes transcritos activamente.[24]
Notas editar
- ↑ Youngson, Robert M. (2006). Collins Dictionary of Human Biology. Glasgow: HarperCollins. ISBN 0-00-722134-7.
- ↑ 2,0 2,1 2,2 2,3 Cox, Michael; Nelson, David R.; Lehninger, Albert L (2005). Lehninger Principles of Biochemistry. San Francisco: W.H. Freeman. ISBN 0-7167-4339-6.
- ↑ Redon C, Pilch D, Rogakou E, Sedelnikova O, Newrock K, Bonner W (2002). "Histone H2A variants H2AX and H2AZ". Curr. Opin. Genet. Dev. 12 (2): 162–9. PMID 11893489. doi:10.1016/S0959-437X(02)00282-4.
- ↑ 4,0 4,1 Bhasin M, Reinherz EL, Reche PA (2006). "Recognition and classification of histones using support vector machine". J. Comput. Biol. 13 (1): 102–12. PMID 16472024. doi:10.1089/cmb.2006.13.102.
- ↑ 5,0 5,1 Hartl, Daniel L., Freifelder, David Snyder, Leon A. (1988). Jones and Bartlett Publishers - Boston, ed. Basic Genetics. ISBN 0-86720-090-1.
- ↑ 6,0 6,1 Luger K, Mäder AW, Richmond RK, Sargent DF, Richmond TJ (1997). "Crystal structure of the nucleosome core particle at 2.8 A resolution". Nature 389 (6648): 251–60. PMID 9305837. doi:10.1038/38444. PDB 1AOI
- ↑ Farkas, Daniel (1996). DNA simplified: the hitchhiker's guide to DNA. Washington, D.C: AACC Press. ISBN 0-915274-84-1.
- ↑ Alva V, Ammelburg M, Lupas AN (2007). "On the origin of the histone fold". BMC Struct Biol 7. PMID 17391511. doi:10.1186/1472-6807-7-17.
- ↑ Ward R, Bowman A, El-Mkami H, Owen-Hughes T, Norman DG (2009). "Long distance PELDOR measurements on the histone core particle". J. Am. Chem. Soc. 131 (4): 1348–9. PMID 19138067. doi:10.1021/ja807918f.
- ↑ Strahl, Brian D.; Allis, C. David (2000-01). "The language of covalent histone modifications". Nature (en inglés) 403 (6765): 41–45. ISSN 0028-0836. doi:10.1038/47412.
- ↑ Strahl BD, Allis CD (Jan 2000). "The language of covalent histone modifications". Nature 403 (6765): 41–5. PMID 10638745. doi:10.1038/47412.
- ↑ Jenuwein T, Allis CD (Aug 2001). "Translating the histone code". Science 293 (5532): 1074–80. PMID 11498575. doi:10.1126/science.1063127.
- ↑ Ning Song, Jie Liu, Shucai An, Tomoya Nishino, Yoshitaka Hishikawa and Takehiko Koji (2011). "Immunohistochemical Analysis of Histone H3 Modifications in Germ Cells during Mouse Spermatogenesis". Acta Histochemica et Cytochemica 44 (4): 183–90. doi:10.1267/ahc.11027. PMC 3168764. PMID 21927517.
- ↑ Benevolenskaya EV (2007). "Histone H3K4 demethylases are essential in development and differentiation". Biochem. Cell Biol. 85 (4): 435–43. PMID 17713579. doi:10.1139/o07-057.
- ↑ 15,0 15,1 15,2 15,3 15,4 15,5 15,6 15,7 Barski A, Cuddapah S, Cui K, Roh TY, Schones DE, Wang Z, Wei G, Chepelev I, Zhao K (2007). "High-resolution profiling of histone methylations in the human genome". Cell 129 (4): 823–37. PMID 17512414. doi:10.1016/j.cell.2007.05.009.
- ↑ 16,0 16,1 16,2 Steger DJ, Lefterova MI, Ying L, Stonestrom AJ, Schupp M, Zhuo D, Vakoc AL, Kim JE, Chen J, Lazar MA, Blobel GA, Vakoc CR (2008). "DOT1L/KMT4 recruitment and H3K79 methylation are ubiquitously coupled with gene transcription in mammalian cells". Mol. Cell. Biol. 28 (8): 2825–39. PMC 2293113. PMID 18285465. doi:10.1128/MCB.02076-07.
- ↑ 17,0 17,1 17,2 Rosenfeld JA, Wang Z, Schones DE, Zhao K, DeSalle R, Zhang MQ (2009). "Determination of enriched histone modifications in non-genic portions of the human genome". BMC Genomics 10: 143. PMC 2667539. PMID 19335899. doi:10.1186/1471-2164-10-143.
- ↑ 18,0 18,1 18,2 Koch CM, Andrews RM, Flicek P, Dillon SC, Karaöz U, Clelland GK, Wilcox S, Beare DM, Fowler JC, Couttet P, James KD, Lefebvre GC, Bruce AW, Dovey OM, Ellis PD, Dhami P, Langford CF, Weng Z, Birney E, Carter NP, Vetrie D, Dunham I (2007). "The landscape of histone modifications across 1% of the human genome in five human cell lines". Genome Res. 17 (6): 691–707. PMC 1891331. PMID 17567990. doi:10.1101/gr.5704207.
- ↑ Kayne PS, Kim UJ, Han M, Mullen JR, Yoshizaki F, Grunstein M. Extremely conserved histone H4 N terminus is dispensable for growth but essential for repressing the silent mating loci in yeast. Cell. 1988 Oct 7;55(1):27-39. PMID 3048701
- ↑ Crane-Robinson C, Dancy SE, Bradbury EM, Garel A, Kovacs AM, Champagne M, Daune M (1976). "Structural studies of chicken erythrocyte histone H5". Eur. J. Biochem. 67 (2): 379–88. PMID 964248. doi:10.1111/j.1432-1033.1976.tb10702.x.
- ↑ Aviles FJ, Chapman GE, Kneale GG, Crane-Robinson C, Bradbury EM (1978). "The conformation of histone H5. Isolation and characterisation of the globular segment". Eur. J. Biochem. (2 ed.) 88: 363–71. PMID 689022. doi:10.1111/j.1432-1033.1978.tb12457.x.
- ↑ Clarke HJ (1992). "Nuclear and chromatin composition of mammalian gametes and early embryos". Biochem. Cell Biol. 70 (10-11): 856–66. PMID 1297351. doi:10.1139/o92-134.
- ↑ Mónica Sancho, Erika Diani, Miguel Beato, Albert Jordan. Depletion of Human Histone H1 Variants Uncovers Specific Roles in Gene Expression and Cell Growth. PLOS Genetics. [1] Cita: Histone H1 in vertebrates is a family of closely related, single-gene encoded proteins, showing much less evolutionary conservation than core histones.
- ↑ Ahmad K, Henikoff S (2002). "The histone variant H3.3 marks active chromatin by replication-independent nucleosome assembly". Mol. Cell 9 (6): 1191–200. PMID 12086617. doi:10.1016/S1097-2765(02)00542-7.
Véxase tamén editar
Outros artigos editar
Ligazóns externas editar
- Animación sobre cromatina, histonas e catepsina
- Nextbio Arquivado 02 de marzo de 2009 en Wayback Machine.