Citoesqueleto procariota
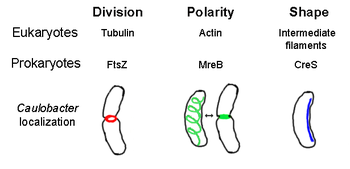
O citoesqueleto procariota é o conxunto de todos os filamentos proteicos estruturais da célula procariota (bacterias e arqueas). Anteriormente pensábase que os procariotas non tiñan citoesqueleto, pero os avances nas tecnoloxías de visualización e determinación da estrutura das células levaron ao escubrimento de filamentos nestas células a principios da década de 1990.[2] Nos procariotas non só se atoparon análogos de todas as principais proteínas citoesqueléticas eucariotas, senón que neles tamén se descubriron proteínas citoesquelétias sen homólogos eucariotas.[3][4][5][6] Os elementos citoesqueléticos exercen funcións esenciais na división celular, protección, determinación da forma e da polaridade en diversos procariotas.[7][8]
Superfamilia da tubulina editar
FtsZ editar
A proteína FtsZ, o primeiro elemento citoesquelético identificado en procariotas, forma unha estrutura anular filamentosa localizada no medio da célula chamada anel Z que constrinxe a célula durante a división celular, similar ao anel contráctil de actina-miosina da citocinese dos eucariotas.[2] O anel Z é unha estrutura moi dinámica que consta de numerosos feixes de protofilamentos que se estenden e encollen, aínda que o mecanimso da contracción do anel Z e o número de protofilamentos implicados non está claro.[1] A FtsZ actúa como unha proteína organizadora e é necesaria para a división celular. É o primeiro compoñente do septo divisorio durante a citocinese, e recruta todas as outras proteínas da división celular coñecidas no sitio da división.[9]
Malia a súa semellanza funcional coa actina, a FtsZ é homóloga da tubulina eucariota. Aínda que a comparación das estruturas primarias da FtsZ e da tubulina revela unha débil relación, as súas estruturas tridimensionais son moi similares. Ademais, igual que a tubulina, a FtsZ monómera únese ao GTP e polimerízase con outros monómeros de FtsZ coa hidrólise do GTP nun mecanismo similar ao da dimerización da tubulina.[10] Como a FtsZ é esencial para a división celular en bacterias, esta proteína é unha diana para o deseño de novos antibióticos.[11] Actualemnte hai varios modelos e mecanismos que regulan a formación do anel Z, pero estes mecanismos dependen da especie. Varias especies de forma bacilar, como Escherichia coli e Caulobacter crescentus, usan un ou máis inhibidores da ensamblaxe da FtsZ que forman un gradiente bipolar na célula, potenciando a polimerización da FtsZ no centro da célula.[12] Un destes sistemas formadores de gradientes consiste nas proteínas MinCDE (véxase máis abaixo).
Superfamilia da actina editar
MreB editar
MreB é unha proteína bacteriana que se cre é homóloga da actina eucariota. A MreB e a actina teñen unha escasa concordancia na súa estrutura primaria, pero son moi similares en estrutura tridimensional e polimerización dos filamentos.
Case todas as bacterias non esféricas dependen de MreB para determinar a súa forma. MreB ensámblase nunha rede helicoidal de estruturas filamentosas xusto debaixo da membrana plasmática, cubrindo toda a lonxitude da célula.[13] MreB determina a forma da célula sendo mediadora da posición e actividade de encimas que sintetizan peptidoglicanos e actuando como un filamento ríxido baixo a membrana que exerce unha presión cara ao exterior para esculpir e dar apoio á célula.[1] MreB condénsase a partir da súa rede helicoidal normal formando un apertado anel no septo en Caulobacter crescentus xusto antes da división celular, un mecanismo que se cre axuda a localizar o seu septo descentrado.[14] MreB é tamén importante para a determinación da polaridade en bacterias polares, xa que é responsable do posicionamento correcto de polo menos catro proteínas polares diferentes en C. crescentus.[14]
ParM e SopA editar
ParM é un elemento citoesquelético que posúe unha estrutura similar á da actina, aínda que se comporta funcionalmente como a tubulina. Ademais, polimerízase bidireccionalmente e mostra unha inestabilidade dinámica, que son ambos comportamentos característicos da polimerización da tubulina.[4][15] Forma un sistema con ParR e parC que é responsable da separación do plásmido R1. ParM fíxase a ParR, unha proteína de unión ao ADN que se une especificamente a 10 repeticións directas na rexión parC do plásmido R1. Esta unión ocorre en ambos os extremos do filamento de ParM. Este filamento esténdese despois, separando os plásmidos.[16] O sistema é análogo á segregación do cromosoma eucariota, xa que ParM actúa como fai a tubulina eucariota no fuso mitótico, ParR actúa como o complexo do cinetocoro, e parC actúa como o centrómero do cromosoma.[17]
A segregación do plásmido F ocorre nun sistema similar onde SopA actúa como o filamento citoesquelético e SopB únese á secuencia sopC do plásmido F, como o cinetocoro e o centrómero, respectivamente.[17] Recentemente, atopouse un homólogo de ParM similar á actina na bacteria grampositiva Bacillus thuringiensis, que se ensambla formando unha estrutura parecida a un microtúbulo e está implicada na segregación de plásmidos.[18]
Actina arqueana editar
A crenactina é un homólogo da actina exclusivo do reino arqueano dos Thermoproteota (antes Crenarchaeota) que se atopou na orde Thermoproteales e no Candidatus Korarchaeum.[19] No momento do seu descubrimento en 2009, tiña a maior similitude de secuencias coas actinas eucariotas de calquera homólogo da actina coñecido.[20] A crenactina foi ben caracterizada en Pyryobaculum calidifontis (A3MWN5) e ten unha alta especificidade polo ATP e GTP.[19] As especies que conteñen crenactina son todas bacilos ou de forma acicular. En P. calidifontis, a crenactina forma estruturas helicoidais que abranguen toda a lonxitude da célula, o que suxire que ten un papel na determinación da forma similar ao de MreB noutros procariotas.[19][21]
Un sistema incluso máis próximo ao sistema da actina eucariota atópase no superfilo proposto dos Asgardarchaeota. Usan versións primitivas da profilina, xelsolina e cofilina para regular o citoesqueleto.[22]
Grupos únicos editar
Crescentina editar
A crescentina (codificada polo xene creS) é un análogo dos filamentos intermedios eucariotas. A diferenza das outras relacións análogas discutidas aquí ata agora, a crescentina ten unha homoloxía primaria bastante grande coas proteínas dos filamentos intermedios ademais dunha semellanza tridimensional; a secuencia de creS ten un 25% de correspondencia de identidade e un 40% de similitude coa citoqueratina 19 e un 24% de correspondencia de identidade e un 40% de similitude coa lamina nuclear A. Ademais, os filamentos de crescentina son de aproximadamente 10 nm de diámetro e están dentro do rango de diámetros dos filamentos intermedios eucariotas (8-15 nm).[23] A crescentina forma un filamento continuo de polo a polo ao longo do lado interno cóncavo da bacteria con forma de lúa crecente Caulobacter crescentus. Tanto a MreB coma a crescentina son necesarias para que C. crescentus poida ter a súa forma característica; crese que MreB moldea a célula dándolle forma de bacilo e que a crescentina a dobra dándolle forma de crecente. O nome da proteína crescentina vén do nome científico da bacteria, crescentus.[1]
Sistema MinCDE editar
O sistema MinCDE é un sistema de filamentos que posiciona no lugar apropiado o septo, no medio da célula en Escherichia coli. Segundo Shih et al., MinC inhibe a formación do septo ao impedir a polimerización do anel Z. MinC, MinD e MinE forman unha estrutura helicoidal que se enrola arredor da célula e está unida á parte interna da membrana por MinD. A hélice de MinCDE ocupa un polo e remata nunha estrutura filamentosa chamada anel E feita de MinE no bordo máis medial da zona polar. Desde esta configuración, o anel E contráese e móvese cara ao polo, desensamblando a hélice de MinCDE a medida que se move. Concomitantemente, os fragmentos desensamblados reensámblanse no extremo polar oposto, reformando o enroscamento de MinCDE no polo oposto mentres que se degrada a hélice MinCDE existente. Este proceso repítese despois, coa hélice MinCDE oscilando de polo a polo. Esta oscilación ocorre repetidamente durante o ciclo celular, mantendo así MinC (e o seu efecto inhibidor do septo) a unha concentración media no tempo máis baixa na zona media da célula que nos extremos desta.[24]
O comportamento dinámico das proteínas Min foi reconstituído in vitro usando unha bicapa lipídica artificial como imitación da membrana celular. MinE e MinD autoorganizábanse en ondas proteicas espirais e paralelas por un mecanismo de tipo reacción-difusión.[25]
Bactofilina editar
A bactofilina (IPR007607) é un elemento citoesquelético β-helicoidal que forma filamentos nas células con forma de bacilo da proteobacteria Myxococcus xanthus.[26] A proteína bactofilina, BacM, cómpre para o mantemento da forma adecuada da célula e a integridade da parede. As células de M. xanthus que carecen de BacM teñen unha morfoloxía deformada caracterizada por un corpo celular dobrado, e os mutantes para bacM teñen unha menor resistencia aos antibióticos que teñen como diana a parede celular bacteriana. A proteína BacM de M. xanthus de lonxitude completa é cortada para permitir a súa polimerización. As bactofilinas foron implicadas na regulación da forma da célula noutras bacterias, incluíndo a curvatura das células de Proteus mirabilis,[27] a formación de pedúnculos por Caulobacter crescentus,[28] e a forma helicoidal de Helicobacter pylori.[29]
CfpA editar
No filo das espiroquetas, varias especies presentan unha estrutura con forma de fita citoplásmica filamentosa formada por filamentos individuais, composta da proteína superenrolada CfpA (Proteína do filamento citoplásmico A ou Cytoplasmic filament protein A, Q56336), ligados entre si por compoñentes que fan de ponte e por áncoras á membrana interna.[30][31] Aínda que está presente nos xéneros Treponema, Spirochaeta, Pillotina, Leptonema, Hollandina e Diplocalyx, está ausente nalgunhas especies como por exemplo Treponema primitia.[32][33][34][35] Cunha dimensión en sección transversal de 5 x 6 nm (horizontal/vertical) está dentro do rango de diámetros dos filamentos intermedios eucariotas (8-15 nm). As células de Treponema denticola que carecen da proteína CfpA forman células concatenadas longas cun defecto na segregación do ADN cromosómico, un fenotipo que tamén afecta á patoxenicidade deste organismo.[36][37] A ausencia doutra ultraestrutura celular, o feixe de filamentos do flaxelo periplásmico, non altera a estrutura da fita citoplasmática.[38]
Notas editar
- ↑ 1,0 1,1 1,2 1,3 Gitai Z (marzo de 2005). "The new bacterial cell biology: moving parts and subcellular architecture". Cell 120 (5): 577–86. PMID 15766522. doi:10.1016/j.cell.2005.02.026.
- ↑ 2,0 2,1 Bi EF, Lutkenhaus J (novembro de 1991). "FtsZ ring structure associated with division in Escherichia coli". Nature 354 (6349): 161–4. Bibcode:1991Natur.354..161B. PMID 1944597. doi:10.1038/354161a0.
- ↑ Gunning PW, Ghoshdastider U, Whitaker S, Popp D, Robinson RC (xuño de 2015). "The evolution of compositionally and functionally distinct actin filaments". Journal of Cell Science 128 (11): 2009–19. PMID 25788699. doi:10.1242/jcs.165563.
- ↑ 4,0 4,1 Popp D, Narita A, Lee LJ, Ghoshdastider U, Xue B, Srinivasan R, Balasubramanian MK, Tanaka T, Robinson RC (xuño de 2012). "Novel actin-like filament structure from Clostridium tetani". The Journal of Biological Chemistry 287 (25): 21121–9. PMC 3375535. PMID 22514279. doi:10.1074/jbc.M112.341016.
- ↑ Popp D, Narita A, Ghoshdastider U, Maeda K, Maéda Y, Oda T, Fujisawa T, Onishi H, Ito K, Robinson RC (abril de 2010). "Polymeric structures and dynamic properties of the bacterial actin AlfA". Journal of Molecular Biology 397 (4): 1031–41. PMID 20156449. doi:10.1016/j.jmb.2010.02.010.
- ↑ Wickstead B, Gull K (agosto 2011). "The evolution of the cytoskeleton". The Journal of Cell Biology 194 (4): 513–25. PMC 3160578. PMID 21859859. doi:10.1083/jcb.201102065.
- ↑ Shih YL, Rothfield L (setembro de 2006). "The bacterial cytoskeleton". Microbiology and Molecular Biology Reviews 70 (3): 729–54. PMC 1594594. PMID 16959967. doi:10.1128/MMBR.00017-06.
- ↑ Michie KA, Löwe J (2006). "Dynamic filaments of the bacterial cytoskeleton" (PDF). Annual Review of Biochemistry 75: 467–92. PMID 16756499. doi:10.1146/annurev.biochem.75.103004.142452. Arquivado dende o orixinal (PDF) o 17 de novembro de 2006.
- ↑ Graumann PL (decembro de 2004). "Cytoskeletal elements in bacteria". Current Opinion in Microbiology 7 (6): 565–71. PMID 15556027. doi:10.1016/j.mib.2004.10.010.
- ↑ Desai A, Mitchison TJ (xullo de 1998). "Tubulin and FtsZ structures: functional and therapeutic implications". BioEssays 20 (7): 523–7. PMID 9722999. doi:10.1002/(SICI)1521-1878(199807)20:7<523::AID-BIES1>3.0.CO;2-L.
- ↑ Haydon DJ, Stokes NR, Ure R, Galbraith G, Bennett JM, Brown DR, Baker PJ, Barynin VV, Rice DW, Sedelnikova SE, Heal JR, Sheridan JM, Aiwale ST, Chauhan PK, Srivastava A, Taneja A, Collins I, Errington J, Czaplewski LG (setembro de 2008). "An inhibitor of FtsZ with potent and selective anti-staphylococcal activity". Science 321 (5896): 1673–5. Bibcode:2008Sci...321.1673H. PMID 18801997. doi:10.1126/science.1159961.
- ↑ Haeusser DP, Margolin W (abril de 2016). "Splitsville: structural and functional insights into the dynamic bacterial Z ring". Nature Reviews. Microbiology 14 (5): 305–19. PMC 5290750. PMID 27040757. doi:10.1038/nrmicro.2016.26.
- ↑ Kürner J, Medalia O, Linaroudis AA, Baumeister W (novembro de 2004). "New insights into the structural organization of eukaryotic and prokaryotic cytoskeletons using cryo-electron tomography". Experimental Cell Research 301 (1): 38–42. PMID 15501443. doi:10.1016/j.yexcr.2004.08.005.
- ↑ 14,0 14,1 Gitai Z, Dye N, Shapiro L (xuño de 2004). "An actin-like gene can determine cell polarity in bacteria". Proceedings of the National Academy of Sciences of the United States of America 101 (23): 8643–8. PMC 423248. PMID 15159537. doi:10.1073/pnas.0402638101.
- ↑ Garner EC, Campbell CS, Mullins RD (novembro de 2004). "Dynamic instability in a DNA-segregating prokaryotic actin homolog". Science 306 (5698): 1021–5. Bibcode:2004Sci...306.1021G. PMID 15528442. doi:10.1126/science.1101313.
- ↑ Møller-Jensen J, Jensen RB, Löwe J, Gerdes K (xuño de 2002). "Prokaryotic DNA segregation by an actin-like filament". The EMBO Journal 21 (12): 3119–27. PMC 126073. PMID 12065424. doi:10.1093/emboj/cdf320.
- ↑ 17,0 17,1 Gitai Z (febreiro de 2006). "Plasmid segregation: a new class of cytoskeletal proteins emerges". Current Biology 16 (4): R133–6. PMID 16488865. doi:10.1016/j.cub.2006.02.007.
- ↑ Jiang S, Narita A, Popp D, Ghoshdastider U, Lee LJ, Srinivasan R, Balasubramanian MK, Oda T, Koh F, Larsson M, Robinson RC (marzo de 2016). "Novel actin filaments from Bacillus thuringiensis form nanotubules for plasmid DNA segregation". Proceedings of the National Academy of Sciences of the United States of America 113 (9): E1200–5. Bibcode:2016PNAS..113E1200J. PMC 4780641. PMID 26873105. doi:10.1073/pnas.1600129113.
- ↑ 19,0 19,1 19,2 Ettema TJ, Lindås AC, Bernander R (maio de 2011). "An actin-based cytoskeleton in archaea". Molecular Microbiology 80 (4): 1052–61. PMID 21414041. doi:10.1111/j.1365-2958.2011.07635.x.
- ↑ Yutin N, Wolf MY, Wolf YI, Koonin EV (febreiro de 2009). "The origins of phagocytosis and eukaryogenesis". Biology Direct 4: 9. PMC 2651865. PMID 19245710. doi:10.1186/1745-6150-4-9.
- ↑ Ghoshdastider U, Jiang S, Popp D, Robinson RC (xullo de 2015). "In search of the primordial actin filament". Proceedings of the National Academy of Sciences of the United States of America 112 (30): 9150–1. PMC 4522752. PMID 26178194. doi:10.1073/pnas.1511568112.
- ↑ Akıl, Caner; Tran, Linh T.; Orhant-Prioux, Magali; Baskaran, Yohendran; Manser, Edward; Blanchoin, Laurent; Robinson, Robert C. (18 de agosto de 2020). "Insights into the evolution of regulated actin dynamics via characterization of primitive gelsolin/cofilin proteins from Asgard archaea". PNAS 117 (33): 19904–19913. PMC 7444086. PMID 32747565. doi:10.1073/pnas.2009167117.
- ↑ Ausmees N, Kuhn JR, Jacobs-Wagner C (decembro de 2003). "The bacterial cytoskeleton: an intermediate filament-like function in cell shape". Cell 115 (6): 705–13. PMID 14675535. doi:10.1016/S0092-8674(03)00935-8.
- ↑ Shih YL, Le T, Rothfield L (xuño de 2003). "Division site selection in Escherichia coli involves dynamic redistribution of Min proteins within coiled structures that extend between the two cell poles". Proceedings of the National Academy of Sciences of the United States of America 100 (13): 7865–70. Bibcode:2003PNAS..100.7865S. PMC 164679. PMID 12766229. doi:10.1073/pnas.1232225100.
- ↑ Loose M, Fischer-Friedrich E, Ries J, Kruse K, Schwille P (maio de 2008). "Spatial regulators for bacterial cell division self-organize into surface waves in vitro". Science 320 (5877): 789–92. Bibcode:2008Sci...320..789L. PMID 18467587. doi:10.1126/science.1154413.
- ↑ Koch MK, McHugh CA, Hoiczyk E (maio de 2011). "BacM, an N-terminally processed bactofilin of Myxococcus xanthus, is crucial for proper cell shape". Molecular Microbiology 80 (4): 1031–51. PMC 3091990. PMID 21414039. doi:10.1111/j.1365-2958.2011.07629.x.
- ↑ Hay NA, Tipper DJ, Gygi D, Hughes C (abril de 1999). "A novel membrane protein influencing cell shape and multicellular swarming of Proteus mirabilis". Journal of Bacteriology 181 (7): 2008–16. PMC 93611. PMID 10094676. doi:10.1128/JB.181.7.2008-2016.1999.
- ↑ Kühn J, Briegel A, Mörschel E, Kahnt J, Leser K, Wick S, Jensen GJ, Thanbichler M (xaneiro de 2010). "Bactofilins, a ubiquitous class of cytoskeletal proteins mediating polar localization of a cell wall synthase in Caulobacter crescentus". The EMBO Journal 29 (2): 327–39. PMC 2824468. PMID 19959992. doi:10.1038/emboj.2009.358.
- ↑ Sycuro LK, Pincus Z, Gutierrez KD, Biboy J, Stern CA, Vollmer W, Salama NR (maio de 2010). "Peptidoglycan crosslinking relaxation promotes Helicobacter pylori's helical shape and stomach colonization". Cell 141 (5): 822–33. PMC 2920535. PMID 20510929. doi:10.1016/j.cell.2010.03.046.
- ↑ Izard J, McEwen BF, Barnard RM, Portuese T, Samsonoff WA, Limberger RJ (febreiro de 2004). "Tomographic reconstruction of treponemal cytoplasmic filaments reveals novel bridging and anchoring components". Molecular Microbiology 51 (3): 609–18. PMID 14731266. doi:10.1046/j.1365-2958.2003.03864.x.
- ↑ You Y, Elmore S, Colton LL, Mackenzie C, Stoops JK, Weinstock GM, Norris SJ (xuño de 1996). "Characterization of the cytoplasmic filament protein gene (cfpA) of Treponema pallidum subsp. pallidum". Journal of Bacteriology 178 (11): 3177–87. PMC 178068. PMID 8655496. doi:10.1128/jb.178.11.3177-3187.1996.
- ↑ Izard J (2006). "Cytoskeletal cytoplasmic filament ribbon of Treponema: a member of an intermediate-like filament protein family". Journal of Molecular Microbiology and Biotechnology 11 (3–5): 159–66. PMID 16983193. doi:10.1159/000094052.
- ↑ Murphy GE, Matson EG, Leadbetter JR, Berg HC, Jensen GJ (marzo de 2008). "Novel ultrastructures of Treponema primitia and their implications for motility". Molecular Microbiology 67 (6): 1184–95. PMC 3082362. PMID 18248579. doi:10.1111/j.1365-2958.2008.06120.x.
- ↑ Izard J, Renken C, Hsieh CE, Desrosiers DC, Dunham-Ems S, La Vake C, Gebhardt LL, Limberger RJ, Cox DL, Marko M, Radolf JD (decembro de 2009). "Cryo-electron tomography elucidates the molecular architecture of Treponema pallidum, the syphilis spirochete". Journal of Bacteriology 191 (24): 7566–80. PMC 2786590. PMID 19820083. doi:10.1128/JB.01031-09.
- ↑ Izard J, Hsieh CE, Limberger RJ, Mannella CA, Marko M (xullo de 2008). "Native cellular architecture of Treponema denticola revealed by cryo-electron tomography". Journal of Structural Biology 163 (1): 10–7. PMC 2519799. PMID 18468917. doi:10.1016/j.jsb.2008.03.009.
- ↑ Izard J, Samsonoff WA, Limberger RJ (febreiro de 2001). "Cytoplasmic filament-deficient mutant of Treponema denticola has pleiotropic defects". Journal of Bacteriology 183 (3): 1078–84. PMC 94976. PMID 11208807. doi:10.1128/JB.183.3.1078-1084.2001.
- ↑ Izard J, Sasaki H, Kent R (2012). "Pathogenicity of Treponema denticola Wild-Type and Mutant Strain Tested by an Active Mode of Periodontal Infection Using Microinjection". International Journal of Dentistry 2012: 549169. PMC 3398590. PMID 22829826. doi:10.1155/2012/549169.
- ↑ Izard J, Samsonoff WA, Kinoshita MB, Limberger RJ (novembro de 1999). "Genetic and structural analyses of cytoplasmic filaments of wild-type Treponema phagedenis and a flagellar filament-deficient mutant". Journal of Bacteriology 181 (21): 6739–46. PMC 94139. PMID 10542176. doi:10.1128/JB.181.21.6739-6746.1999.